目录
1.问题描述
基因序列包含四种核苷酸,分别用A,C,T和G四个字母简单地表示。 编写一个程序,按以下规则比较两个基因并确定它们的相似程度。
规则:使用对齐方法,可以在基因的适当位置插进空格,让两个基因的长度相等,然后根据基因分值矩阵计算分数。
例如,给AGTGATG插入一个空格,就得到AGTGAT-G;给GTTAG插入三个空格,就得到-GT--TAG。空格用减号(-)表示。
把?AGTGAT-G与-GT--TAG 对齐以后,有四个基因是相配的:第二位的G,第三位的T,第六位的T和第八位的G。根据下列基因分值矩阵,每对匹配的字符都有相应的分值。 * 表示空格对空格是不允许的。上面对齐的字符串分值是: (-3)+5+5+(-2)+(-3)+5+(-3)+5=9。
| A | C | G | T | - | |
| A | 5 | -1 | -2 | -1 | -3 |
| C | -1 | 5 | -3 | -2 | -4 |
| G | -2 | -3 | 5 | -2 | -2 |
| T | -1 | -2 | -2 | 5 | -1 |
| - | -3 | -4 | -2 | -1 | * |
另一种对齐方式(不同数量的空格插进不同的位置): AGTGATG与-GTTA-G 这种对齐方式的分值是(-3)+5+5+(-2)+5+(-1) +5=14,这种对齐方式是最优的,没有其他的方式能得到更高的分值了,所以这两个基因的相似度是14。
2.一些细节
(1)基因分值矩阵的表示:
int score[5][5] = {{5, -1, -2, -1, -3},
{-1, 5, -3, -2, -4},
{-2, -3, 5, -2, -2},
{-1, -2, -2, 5, -1},
{-3, -4, -2, -1, 0}};(2)原矩阵的下标是'A','C','G','T'和'-',可以采用switch语句转换,这里采用map数组转换:
char map[128]中:map['A'] = 0; map['C'] = 1; map['G'] = 2; map['T'] = 3; map['-'] = 4;
3.思路
本题类似最长公共子序列(LCS)问题
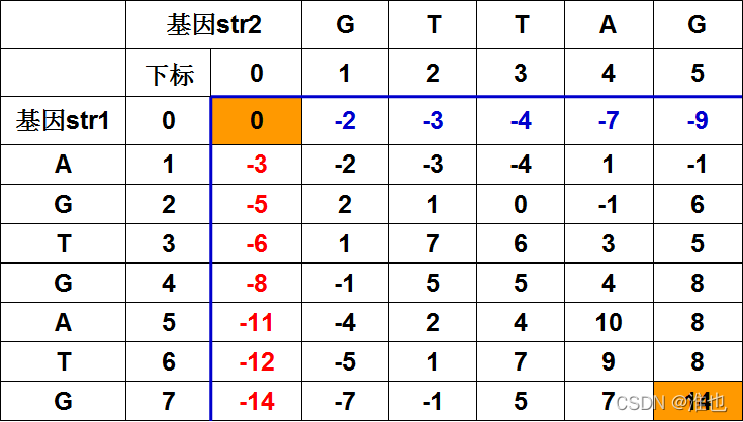
gene[i][j]表示基因子串str1[0…i-1]和str2[0…j-1]的分值:
- str1取第i-1个字母,str2取'-': m1 = gene[i-1][j] + score[map[str1[i-1]]][4];
- str1取'-',str2取第j-1个字母: m2 = gene[i][j-1] + score[4][map[str2[j-1]]];
- str1取第i-1个字母,str2取第j-1个字母:m3 = gene[i-1][j-1] + score[map[str1[i-1]]][map[str2[j-1]]];
gene[i][j] = max(m1, m2, m3)
最终结果是gene[first][second]
注意:字符串str1, str2的下标从0开始,数组gene中的下标从1开始,所以下标差1
初始化:
- 当i=0, j=0时,gene[0][0] = 0;
- 当i=0时,即为gene [0,1…second]
- for(i = 1 ; i <= second;i++)
- gene[0][i] = gene[0][i-1]+score[4][map[str2[i-1]]];
- for(i = 1 ; i <= second;i++)
- (3) 当j=0时,即为gene [1…first, 0]
- for(i = 1 ; i <= first;i++)
- gene[i][0] = gene[i-1][0]+score[map[str1[i-1]]][4];
- for(i = 1 ; i <= first;i++)
4.代码
#include <iostream>
#include <cstdio>
using namespace std;
#define MAX 101
int score[5][5]={
{5,-1,-2,-1,-3},
{-1,5,-3,-2,-4},
{-2,-3,5,-2,-2},
{-1,-2,-2,5,-1},
{-3,-4,-2,-1,0}};
int gene[MAX][MAX];
int main()
{
int n,length1,length2;
scanf("%d",&n);
char str1[MAX],str2[MAX];
char map[100];
map['A']=0;map['C']=1;map['G']=2;
map['T']=3;map['-']=4;
while(n--){
scanf("%d%s",&length1,str1);
scanf("%d%s",&length2,str2);
gene[0][0]=0;
for(int i=1;i<=length2;i++)
gene[0][i]=gene[0][i-1]+score[4][map[str2[i-1]]];
for(int i=1;i<=length1;i++)
gene[i][0]=gene[i-1][0]+score[map[str1[i-1]]][4];
int m1,m2,m3;
for(int i=1;i<=length1;i++)
for(int j=1;j<=length2;j++){
m1=gene[i-1][j]+score[map[str1[i-1]]][4];
m2=gene[i][j-1]+score[4][map[str2[j-1]]];
m3=gene[i-1][j-1]+score[map[str1[i-1]]][map[str2[j-1]]];
gene[i][j]=max(m1,max(m2,m3));
}
printf("%d",gene[length1][length2]);
}
return 0;
}