????????本文对应第一个实例:basic_example.zip,分为七部分。七个部分即运行七个脚本,需要安装python后,在cmd命令行下运行。所有文件应该放在同一个文件夹中。我的在d盘demo文件夹中。
运行“cmd”(或者打开modeller程序)
输入“d:”回车
输入“cd demo”回车(你得告诉程序脚本要处理的文件在哪儿放着,把运行目录设置好了)

一、搜模版(build_profile.py脚本)

???????蓝框里的文件名都可以改成自己的,当然,不改肯定不会出错。
?????? pdb_95.pir :必要的输入文件,含有自然界95%蛋白结构的氨基酸序列数据库。
??????????????????????????? 如果搜索不到自己想要的结构可以更换完整数据库pdball.pir,官网上有。
?????? Build_profile.prf :相当于程序的输出日志。
?????? Build_profile.ali :搜索到的氨基酸序列。
???????TvLDH.ali :包含有需要建模的氨基酸序列。(蓝色部分需要自己粘贴进去,以星号“*”结尾)

????????设置好了参数运行后就是等了,用95%的库搜索很快,用全库就比较慢。完成后就会有Build_profile.ali与Build_profile.prf两个输出文件。(我用的是10.1的modeller程序与群里的python版本,其他版本可能需要在脚本名前面输入python再回车。)
?
?打开pir后缀看一下日志:

????????第11列是你提交的氨基酸序列与下面蛋白的相似度;第12列分值越接近0,说明这个模版越适合你;短横线是缺少这一块氨基酸。
????????我们选都是0的几个模版(1bdm:A, 5mdh:A, 1b8p:A, 1civ:A, 7mdh:A),下载好pdb文件。为下一步做准备。
二、对比模版(compare.py脚本)

????????1b8p指的是蛋白质编号,A指的是A链。都可以根据情况适当更改。直接运行脚本,结果只会在cmd界面打印出来。而通过compare.py>compare.log的命令方式可以将信息输出到log文件里。

? ? ? ?第一个主要信息是一个表,表以蓝色的对角线分成两个部分,对角线上是这个蛋白的氨基酸个数,对角线上方的三角是模版间的相似氨基酸个数,对角线下面是模版间的相似度。
?????? 第二个主要信息是下面这个树形结构。“@1.9”指的是pdb的分辨率为1.9埃。
?????? 选模版的标准主要有几种:模版间的相似性(适中最好)、晶体分辨率(越小越好)、与要建模的序列同一性(越大越好,在第一步的pir输出文件里有体现)、晶体R因子(越小越好,需要下载cif格式的模版蛋白质,用记事本打开,搜索R_factor)。
?????? 比较表明1civ:A和 7mdh:A在顺序和结构上几乎相同。然而,7mdh:A晶体学分辨率好2.4?,排除了 1civ:A。第二组结构(5mdh:A、1bdm:A和 1b8p:A)有一些相似之处。在该组中, 5mdh:A的分辨率最差,仅考虑1bdm:A和 1b8p:A。1smk:A是整套可能模板中最多样化的结构。然而,它是与查询序列具有最低序列同一性 (34%) 的序列。最终选择1bdm:A 而不是1b8p:A和 7mdh:A,因为它具有更小的晶体学 R 因子 (16.9%) 和更高的序列同一性 (45%)。
三、对齐模版(align2d.py脚本)

?
?????? 运行的事,后面不会再说。蓝框里的东西都可以改名,但是你如果老报错,就不要改了(像TvLDH.ali、TvLDH),只把涉及的到的部分修改就好(像1bdm、1bdmA、1bdm.pdb)。
四、建模(model_single.py脚本)

? ? ? ?脚本按照 “TvLDH-1bdmA.ali”文件的对齐方式进行建模;knows的值即模版+链的方式;sequence的值指的是“TvLDH.ali”文件里第二行“sequence:”字段后的名字,如果前面几步操作的时候改了名字,后面都要注意更改。
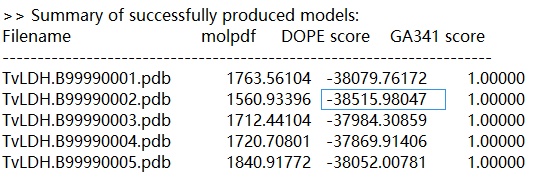
??????? 在建模的同时,脚本会对模型进行评分,评分方式有molpdf(越小越好)、DOPE(越小越好)、GA341(越接近1越好),其中molpdf默认输出,DOPE、GA341需要在代码中指定。如果蛋白原子数特别多,建模就慢,我试过注释掉assess.GA341(不用这个评价方式)可以略微加快速度。
?????? 运行完成后输出5个pdb文件,及其打分情况。可以看出“TvLDH.B99990002.pdb”文件最好。
五、打分建模的模型(evaluate_model.py脚本)

????????运行完后,每个氨基酸对应的dope得分会存入“TvLDH.profile”文件中,我们这里不需要打开它。
六、打分对照的模版(evaluate_template.py脚本)

????????与第五步类似,区别在于需要指定模版蛋白的链,这里只用到它的A链
七、作图(plot_profiles.py.脚本)
????????此脚本运行之前需要python安装一个作图插件matplotlib,这个网上不少教程,不在此赘述。

????????由对齐文件、五六步打分的输出文件作为程序的输入文件,做了一个10:6、dpi为65的图像。?

????????打分数值不是绝对好坏的标准,只能粗略的看一下,通过比较两条线形状上的差异,看潜在的错误。纵坐标-0.03是一个经常用的标准(我还没搞明白为啥);横坐标表示氨基酸,90-100、220-250氨基酸能量明显偏高。DOPE 指示的可能错误不一定是实际错误,尤其是当它突出显示活动位点或蛋白质-蛋白质界面时(网站上的原话)。
?????? 对结构进行优化,可以通过对loop区的精炼、在模版中加入必要配体,限制配体与蛋白原子间距离、循环迭代建模等方法。这些方法下期再讲。?
?
?