背景
在ncbi上查一个细菌的全基因组,搜取全基因组上的这五种信息:gene,locus_tag,note,location,product,蛋白序列的前10个氨基酸(为什么爬这个最后讲),并分析这个基因组上对于蛋白的注释note和功能product,来找到基因组上对自己有用的信息。以它为例。
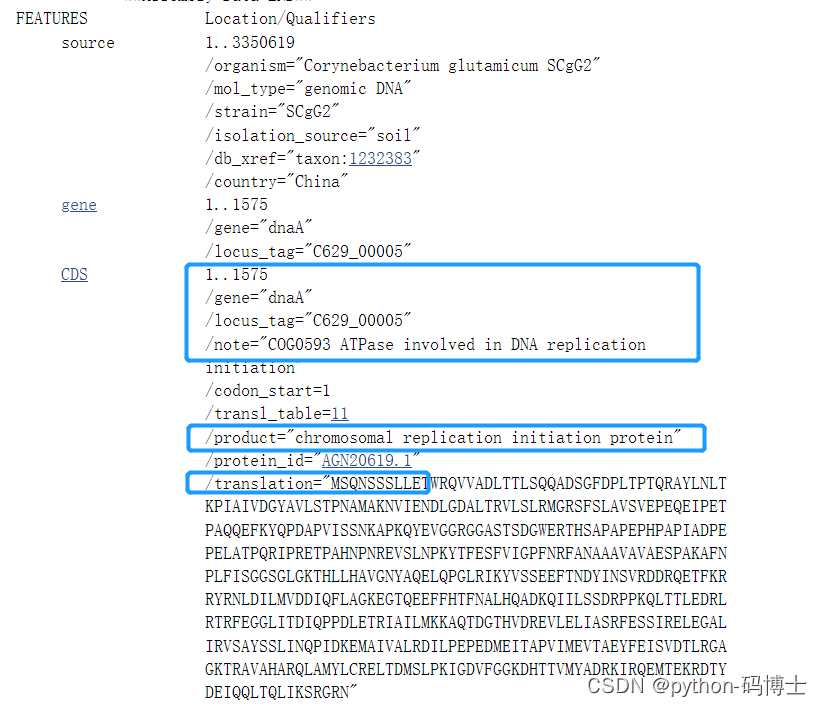
效果图
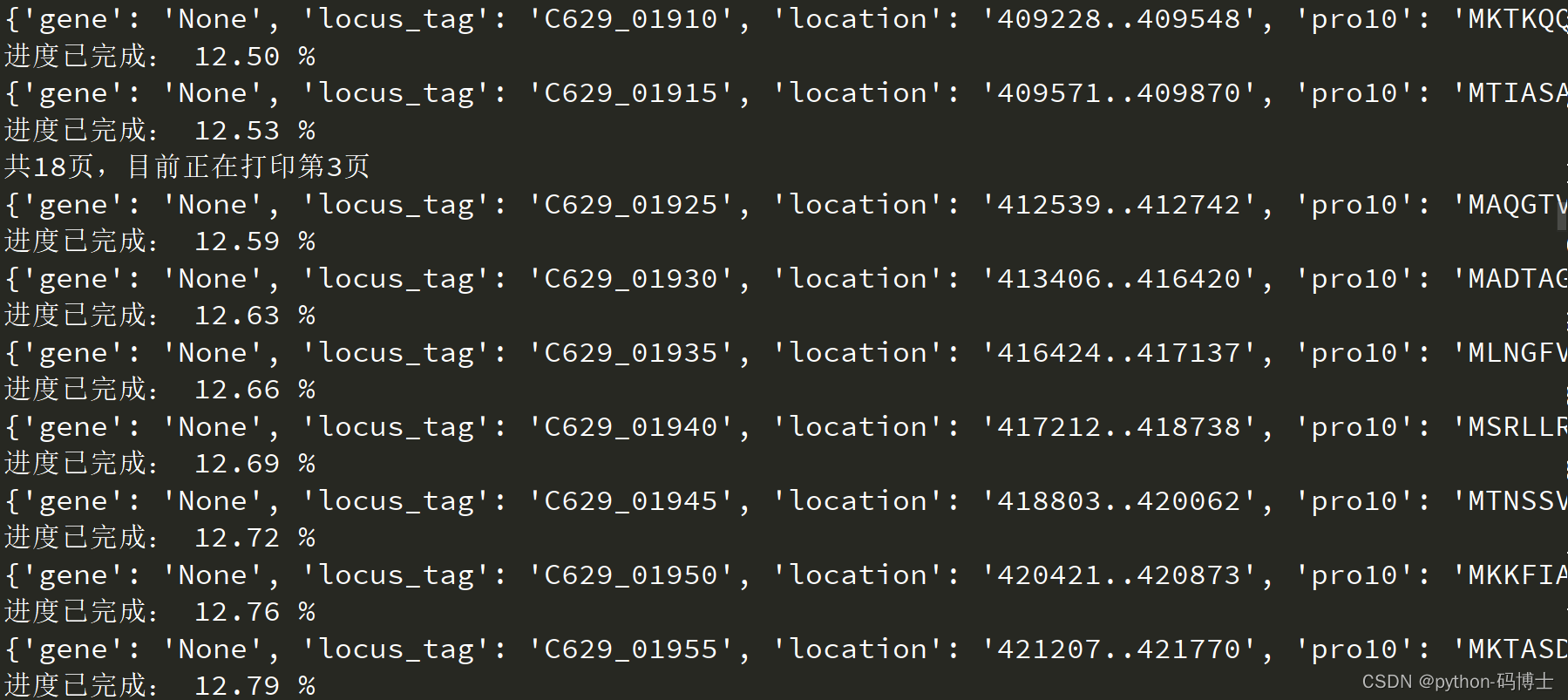
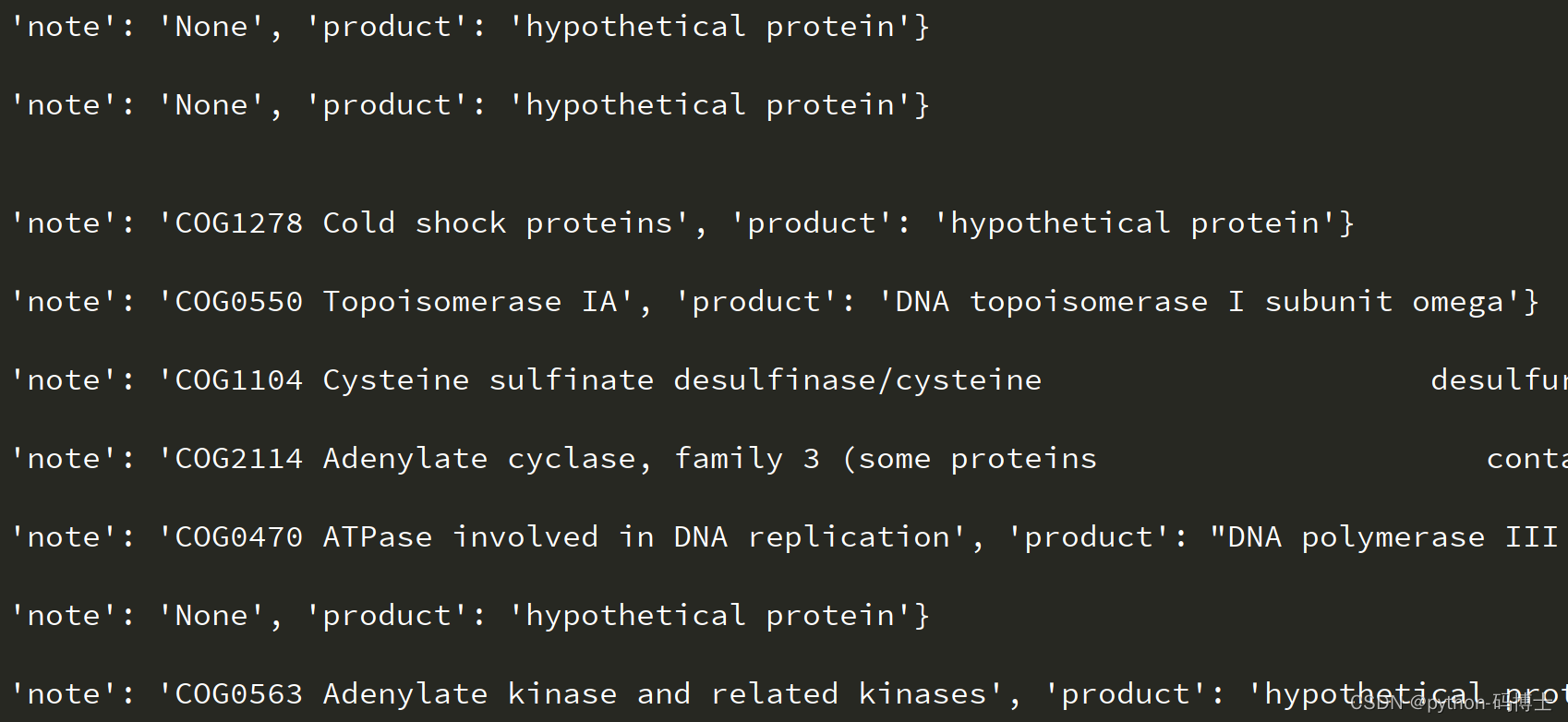
写入csv文件的样子
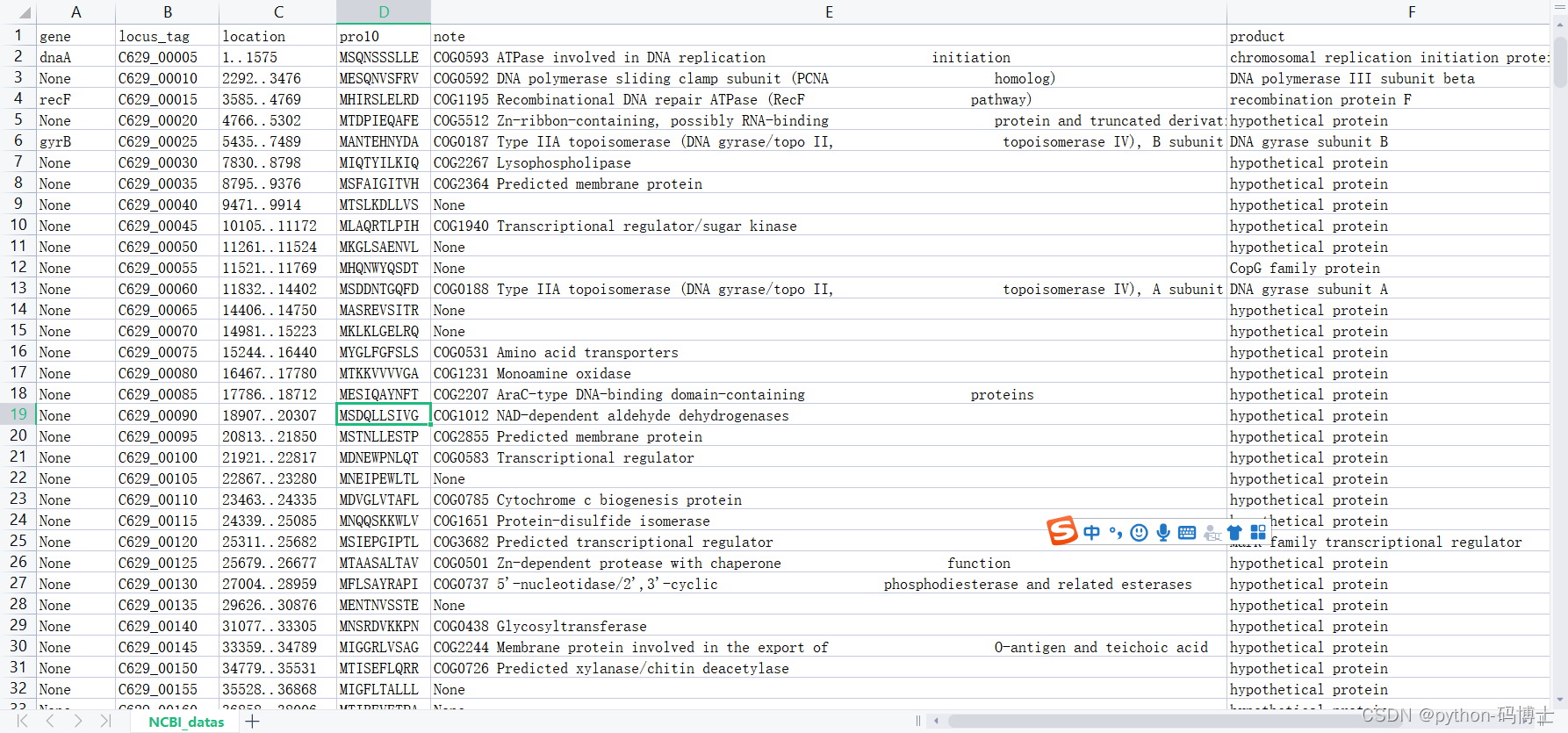
代码思路
首先观察网页原代码,发现要搜寻的数据并不在原网页,于是深度挖掘,发现数据的url地址在这一块,且存储在了17个url里面
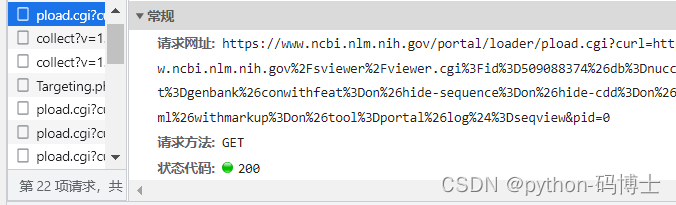
通过对这个网址的请求,发现数据确实在里面且可以访问。于是接下来进行数据的处理,说多了都是泪,直接上源代码吧,大约100来行代码,纯手撸!!!有编程基础的朋友可以试着实现一下。
import requests
import random
import re
import csv
import time
import copy
def html_source(url,headers):
resp = requests.get(url,headers)
if resp.status_code == 200:
content = resp.text
text_process(content)
else:
print('error 404')
def text_process(content):
content = content.replace('\n','')
sort_rule = re.compile('CDS\s(.*?/translation="[A-Z]{10}).*?')
sort_list = sort_rule.findall(content)
for one_list in sort_list:
gene_rule = re.compile('/gene="(.*?)"')
locus_tag_rule = re.compile('locus_tag="(.*?)"')
location_rule = re.compile('\s[a-z(]{0,11}(\d+..\d+)[)]{0,1}\s')
pro10_rule = re.compile('/translation="([A-Z]{10}).*?')
note_rule = re.compile('/note="(.*?)"')
product_rule = re.compile('/product="(.*?)"')
gene = gene_rule.findall(one_list)
locus_tag = locus_tag_rule.findall(one_list)
location = location_rule.findall(one_list)
pro10 = pro10_rule.findall(one_list)
note = note_rule.findall(one_list,re.S)
product = product_rule.findall(one_list)
if len(gene) == 0:
dict['gene'] = 'None'
else:
dict['gene'] = gene[0]
if len(locus_tag) == 0:
dict['locus_tag'] = 'None'
else:
dict['locus_tag'] = locus_tag[0]
if len(location) == 0:
dict['location'] = 'None'
else:
dict['location'] = location[0]
if len(pro10) == 0:
dict['pro10'] = 'None'
else:
dict['pro10'] = pro10[0]
if len(note) == 0:
dict['note'] = 'None'
else:
dict['note'] = note[0]
if len(product) == 0:
dict['product'] = 'None'
else:
dict['product'] = product[0]
num = int(dict['locus_tag'][5:])
timer = num/15285*100
print(dict)
print('进度已完成:',"%.2f"%timer,'%')
lst.append(copy.deepcopy(dict))
'''
在一个列表中,如果几个元素的id相同,则内容会变一致,即后来的覆盖前面的
因为在这个循环中dict共用一个id,后面的会把前面的覆盖掉,所以要用深拷贝(深拷贝是另外开辟一块内存)
'''
time.sleep(random.uniform(0.2,0.5 ))
def write(list):
j = 0
with open('NCBI_datas.csv', 'w', newline='', encoding='utf-8') as file_obj:
title = ['gene', 'locus_tag', 'location', 'pro10', 'note', 'product']
writer = csv.DictWriter(file_obj, fieldnames=title)
writer.writeheader()
# writer.writerow(list)
for li in list:
j = j + 1
writer.writerow(li)
print(f'靠北啦!!!一共写了{j}条数据,累死了')
if __name__ == '__main__':
lst = []
USER_AGENTS = []
COOKIES = []
headers = {}
for i in range(18):
dict = {}
h = i + 1
print(f'共18页,目前正在打印第{h}页')
url = ''.format(i)
html_source(url,headers)
time.sleep(random.randint(1,2))
write(lst)
ps
虽然可以省去一个一个看的麻烦,但是数据量还是蛮大的,我的有3000多条数据,可以适当的把没有note或gene的数据删除,两种方法:一种是改源代码,另一种是数据分析。
接背景:为什么要前10个氨基酸序列
因为我发现有的蛋白是没有gene这个选项或者通过gene搜不到,可以通过前10个氨基酸序列去snapgene上匹配,就可以找到这个序列了。见下图(通过dnaA搜不到却可以通过前10个氨基酸序列搜到)


今天说话不丝滑,有好多点没有讲出来,就先这样吧!!!小白一枚,不喜勿喷。
