文章目录
Github地址:https://github.com/AIM-Harvard/pyradiomics
官方文档地址:https://pyradiomics.readthedocs.io/en/latest/features.html
1. 使用插件
从pyradiomics的Github仓库中可以知道,pyradiomics也有一个3D Slicer的插件——SlicerRadiomics(插件安好了需要重启),考虑先用插件试一下。
1.1 插件安装
1.2 插件使用
先打开一个图,然后在Module这里选择Informatics->Radiomics
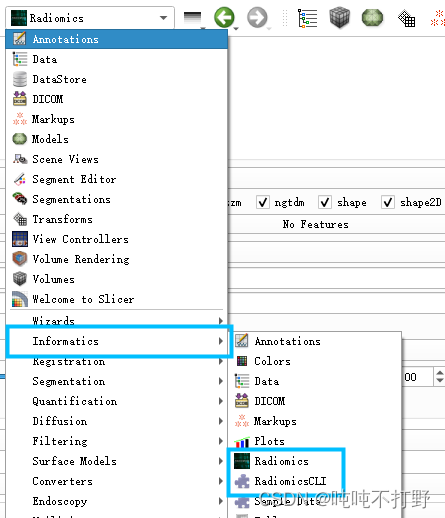
然后左侧就会弹出一些选项,
-
选好自己的
图像volume和对应的mask部分
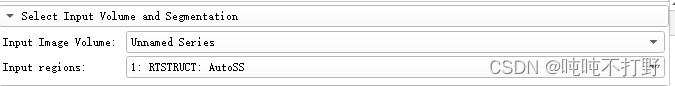
-
直接选择要所有特征
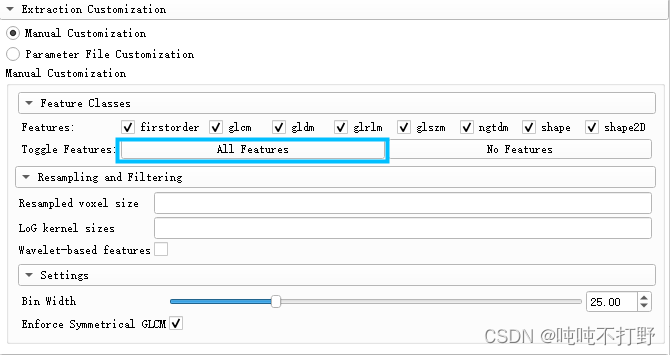
这里的特征其实就对应于官网的内容,即:- firstorder → First Order Statistics (19 features)
- glcm → Gray Level Cooccurence Matrix (24 features)
- gldm → Gray Level Dependence Matrix (14 features)
- glrlm → Gray Level Run Length Matrix (16 features)
- glszm → Gray Level Size Zone Matrix (16 features)
- ngtdm → Neighbouring Gray Tone Difference Matrix (5 features)
- shape → Shape-based (3D) (16 features)
- shape2D → Shape-based (2D) (10 features)
-
在
output table中选择Create a new table,选完之后点击Apply

-
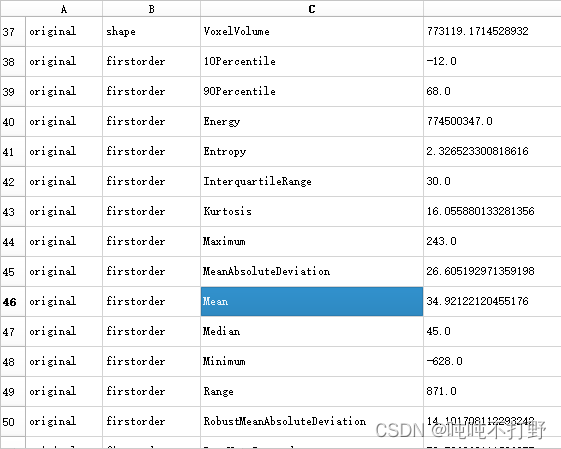
然后一般可以在右下角看到
Radiomics Features影像组学特征(我这里的示例使用的是DIOCM图,同时有5种mask,所以可以看到后面有5列mask)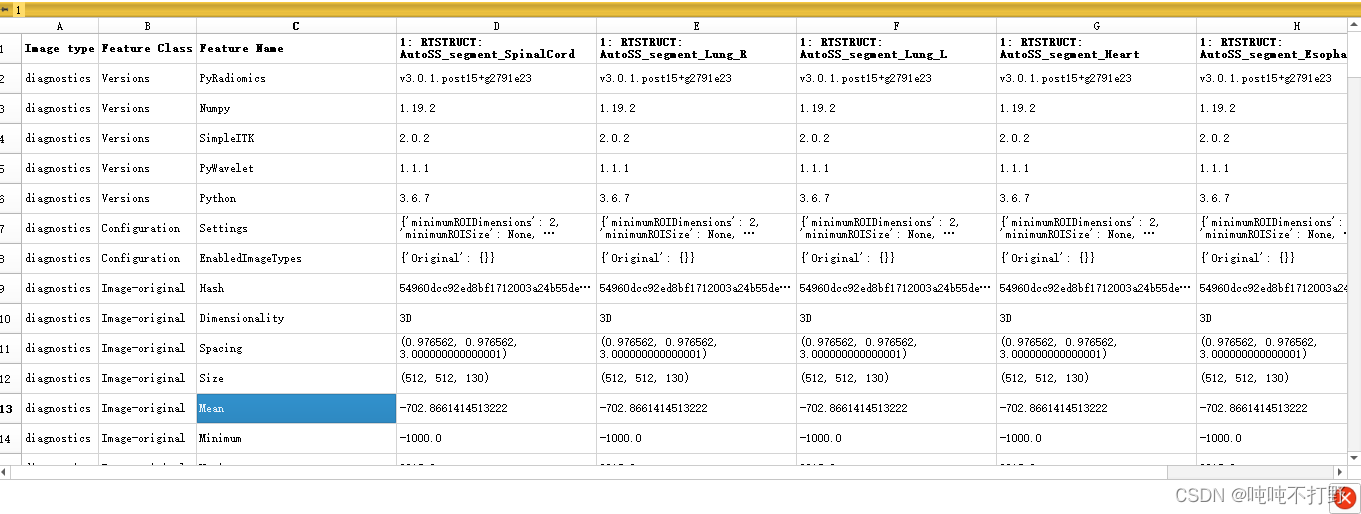
类似的,也可以看看别的图的组学特征结果,但是每个字段的意义可能需要了解一下
2. 使用python代码
使用了3D Slicer的插件之后,大概了解这个东西是做什么的,接下来就用python来进行批量处理了。
2.1 安装
最基本的
python -m pip install pyradiomics
2.2 使用
2.2.1 官方示例
其实官方给了示例的,在官方文档的Example中可以知道, 示例代码位于pyradiomics的github中,链接
- 示例数据在
pyradiomics/data中,链接 - jupyter文件位于
pyradiomics/notebooks中,链接 - 教程视频中所使用的参数文件位于
pyradiomics/examples/exampleSettings中,链接 - 如果没有安装jupyter,可以直接运行python代码:
- 位于
pyradiomics/examples,链接 python helloRadiomics.py(segment-based extraction)python helloVoxel.py(voxel-based extraction)
- 位于
2.2.2 快速使用
主要就是命令行调用来生成相应的特征
1. 确认可以调用命令行
切换到你安装了pyradiomics的conda环境下,例如,base环境,
先用 -h 看一下能不能在命令行调用pyradiomics"
(base) C:\XXXX> pyradiomics -h
usage: pyradiomics image|batch [mask] [Options]
optional arguments:
-h, --help show this help message and exit
--label N, -l N (DEPRECATED) Value of label in mask to use for
feature extraction.
--version Print version and exit
Input:
Input files and arguments defining the extraction:
- image and mask files (single mode) or CSV-file specifying them (batch mode)
- Parameter file (.yml/.yaml or .json)
- Overrides for customization type 3 ("settings")
- Multi-threaded batch processing
2. 提取单个图像及对应mask的特征
pyradiomics <path/to/image> <path/to/segmentation>
"例如:"
pyradiomics "./data/Test-S1-101.nii.gz" "./label/Test-S1-101.nii.gz"
"默认结果输出到命令行中,如果想保存到文件里,可以"
pyradiomics <path/to/image> <path/to/segmentation> -o results.csv -f csv
"例如:"
pyradiomics "./data/Test-S1-101.nii.gz" "./label/Test-S1-101.nii.gz" -o results.csv -f csv
"然后就会在执行命令所在目录看到生成的那个文件了"
3. 提取批量
批量提取需要提前做一个.csv文件,第一行是表头,剩下的是数据,至少要包含:
- 第一列是image的图像路径
- 第二列是对应的mask的路径
- 对应第一列和第二列的表头必须是:
Image和Mask(大小写也不能错)
例如:
--All.csv--
Image,Mask
./data/Test-S1-101.nii.gz,./label/Test-S1-101.nii.gz
./data/Test-S1-102.nii.gz,./label/Test-S1-102.nii.gz
./data/Test-S1-103.nii.gz,./label/Test-S1-103.nii.gz
./data/Test-S1-104.nii.gz,./label/Test-S1-104.nii.gz
./data/Test-S1-201.nii.gz,./label/Test-S1-201.nii.gz
./data/Test-S1-202.nii.gz,./label/Test-S1-202.nii.gz
./data/Test-S1-203.nii.gz,./label/Test-S1-203.nii.gz
./data/Test-S1-204.nii.gz,./label/Test-S1-204.nii.gz
执行命令
pyradiomics <path/to/input> -o results.csv -f csv
"例如"
pyradiomics "./All.csv" -o results.csv -f csv
计算可能会比较慢,等会就行
2.2.3 其它使用
jupyter示例中展示了很多其它操作,可以看看
TBD
另外,
3. 组学特征字段意义说明
- 一阶统计特征,反应所测体素的对称性、均匀性以及局部强度分布变化。包括中值,平均值,最小值,最大值,标准差,偏度,峰度等。
- 形态特征,定量描述感兴趣区的几何特性,如肿瘤的表面积、体积、表面积和体积比、球形度、紧凑性和三维直径等,这些特征可以描述肿瘤三维的大小和形态信息。此外,还有原始图像或使用不同滤波方法对影像进行滤波后的
- 二阶及高阶纹理特征,这些特征能够反映图像体素灰度之间的空间排列关系。
特征字段比较多,这里只研究自己关心的内容,主要是:
- mask部分对应的图像的强度值范围 → HU值(窗宽,窗位)
- mask部分形状在长度、宽度和深度三个轴上的最值(shape范围) → 预处理时的crop方式
其实官方文档写的还是比较清晰的,这里对上面自己关心部分的文档进行简单说明
3.1 HU值范围判定(归一化时的上界和下界)
在一阶特征中,可以知道

使用
X
X
X表示ROI区域(mask区域)的
N
p
N_p
Np?个体素点

其中Minimum和Maximum分别表示mask对应的ROI区域中最大的和最小的灰度强度值。
但是统计之后发现,
- 这里的ROI区域的最大值和最小值,有些竟然是和全图的最大值或者最小值保持一致的。。
- 所以如果想通过这种方式去确定HU值的范围,应该还需要看均值,不能单纯看最大值或者最小值
- 另外,最好是查看每个图ROI区域的直方图,然后统计出所有图的最高频率的ROI直方图的范围,来作为最后的上界和下界。
- 暂时还不知道怎么操作,TBD
参考:
-
?CSDN博客:【影像组学pyradiomics教程】(七)影像组学特征,翻译了一下,但是也有点用吧。另外这个博主也有相关的专栏,可以关注。
-
?pyradiomics-features:pyradiomics官网文档关于特征的说明,有计算公式和说明,但是是英语的,看起来会比较费劲。
-
?英文pdf:Image biomarker standardisation initiative Reference manual,中文直译是
图像生物标志物标准化计划参考手册。部分目录截图如下

-
百度文库的一个ppt:https://wenku.baidu.com/view/1c10a9071a2e453610661ed9ad51f01dc281570d.html
-
百度文库的一个文档:https://wenku.baidu.com/view/40282667bd1e650e52ea551810a6f524ccbfcb00.html
-
百度文库的一个文档:https://wenku.baidu.com/view/e4950c0a463610661ed9ad51f01dc281e53a568a.html
-
微信文章合集:
其它可能有参考价值的内容:
- ?Radiomics学习总结,写的挺全面的(对我这个小白很全面了),这个博主的其它相关文章也可以看看
- 医学影像组学特征值(Radiomics Features)提取
- 影像组学学习笔记(22)-从atlas抠出想要的ROI做成mask文件
- B站相关领域UP主:卢家峰教授影像组学教学
- 对应的内容:MR Radiomics Platform (MRP)
& Machine-Learning Models - 这个人维护了一个用来看组学特征的软件,可以考虑学着用一下
- 对应的内容:MR Radiomics Platform (MRP)
- 知乎专栏文章:有Li影像组学
- 还有一个论坛:Li’s 影像组学视频学习笔记(1)-基础讲解
- 也有CSDN博客:有Li的博客
- 知乎专栏文章:组学浅析之通俗理解影像组学的定义~,这个博主也是有个专栏,也可以看看其它文章,例如:Radiology:影像组学特征的生物学意义
- 简书文章:【影像组学导论】第一部分:官方定义解读。这咋整?读懂这个频道就够了!
