目录
前言
读了 Frontiers in Biocatalysis: Profiling Function across Sequence Space 有一些感受。
本文是对于 Site-Selective C?H Halogenation Using Flavin-Dependent Halogenases Identified via Family-Wide Activity Profiling. ACS Cent. Sci. 2019. (DOI: 10.1021/acscentsci.9b00835. )的评述文章。
Frontiers中归纳了三种序列可视化工具:MSA, 系统发育树和SSN。MSA是序列数据最直接的展现;系统发育树展现了序列随物种的进化关系(存疑);SSN是聚类算法结果直观的展现,可以用于标注各种信息。
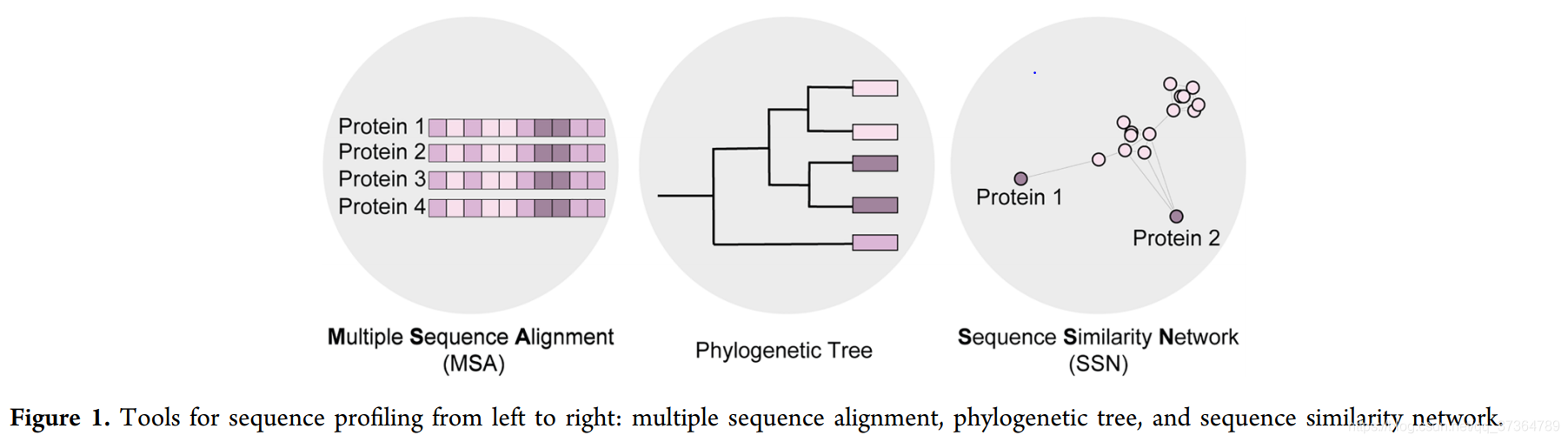
其启示在于,从序列可视化的角度而言,是否有其它工具的选择?序列的评估更应依赖于算法,但其概念的直观展现以及需要研究人员作出判断的标准仍须由可视化工具来给出。简单而言,可视化是与人的交互。
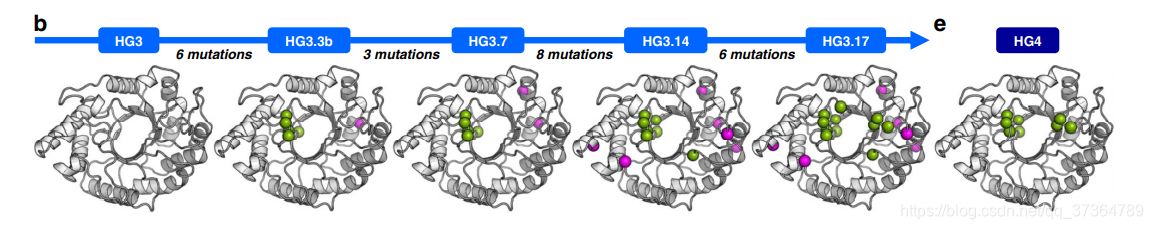
例如,酶工程中,我们可以按照酶的(定向)进化或者设计的轮次以及所获得效果来组织序列数据,如 NC 的《计算酶设计重现定向进化效果》一文中的图(https://doi.org/10.1038/s41467-020-18619-x)。
又或者,在酶-物质相互作用中的热图,实际上也是一种可视化方案(来自本文)。
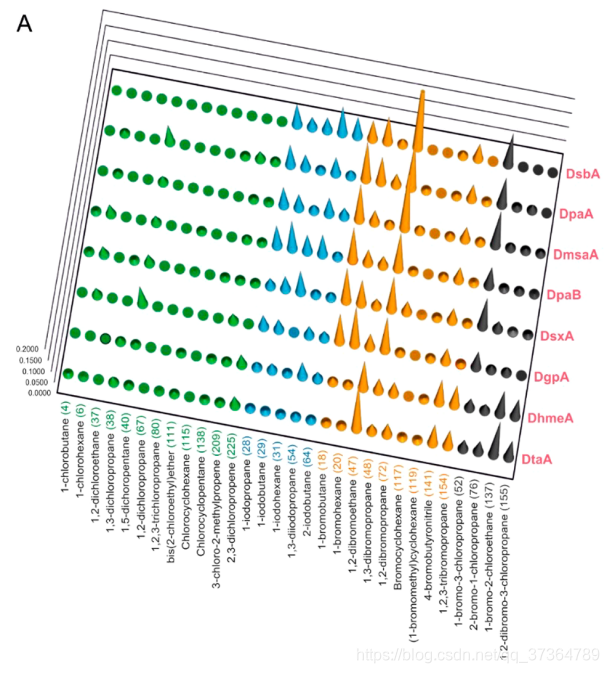
相应的可视化方法在研究中已大量采用,但因为数据规范、数据标注和数据库的原因,并未被固定化为一种通用工具。
论文复现
Exploration of Enzyme Diversity by Integrating Bioinformatics with Expression Analysis and Biochemical Characterization (ACS Catal. 2018, 8, 3, 2402–2412)
Clustal Omega
搜索Clustal Omega.
[100.txt] 作为输入,里面包含了100条序列。
得到结果后,Download Alignment File - 保存为 100.clustal_num。
Results Viewers
下载 Jalview 软件查看结果。
Jalview Cite as:
Waterhouse A.M., Procter J.B., Martin D.M.A., Clamp M., Barton G.J. (2009)
Jalview Version 2-a multiple sequence alignment editor and analysis workbench.
Bioinformatics 25: 1189-1191. Pubmed: 19151095 DOI: doi:10.1093/bioinformatics/btp033
下载大概需要10分钟。
Jalview
EBI 的 Help 页面:https://www.ebi.ac.uk/seqdb/confluence/display/JDSAT/Jalview
Jalview 的 Help 页面:http://www.jalview.org/Help/Getting-Started
Jalview is a free program for multiple sequence alignment editing, visualisation and analysis. Jalview has been developed in the Barton Group at the University of Dundee, and can be used to view and edit sequence alignments, analyse them with phylogenetic trees and principal components analysis (PCA) plots and explore molecular structures and annotation.
安装时需要支持的文件格式已经能看出来是个多功能的Viewer了,不只能支持序列,还能支持结构,以及各种树的构建。
把菜单栏的项目过了一遍,有各种标记、统计、计算、风格化的选项,在使用的时候需要去了解所做操作的生物学含义。
可以去学一学生物信息学的课程,搜一搜对应的概念。
The criteria for a complete catalytic pentad were as follows: **(i) Asp in the position of the nucleophile (the position corresponding to Asp108 of LinB), (ii) Asp or Glu in at least one of the catalytic acid positions (those corresponding to Asp260 of DhlA or Glu132 of LinB), (iii) His in the position of the base (position His272 of LinB) and (iv) Asn, Gln, Trp, Tyr or His in at least two halide-binding positions (position Asn38 or Trp109 of LinB, or Trp175 of DhlA).
LinB中的关键氨基酸残基以及按WP_1103158.2的位置来选择:
| 作用 | 残基 | MSA中的编号 |
|---|---|---|
| 亲核残基 | Asp108 D | 111D |
| 酸残基 | Glu132 E | 135E |
| 碱残基 | His272 H | 275H |
| 卤素结合残基 | Asn38, Trp109 N,W | 41N, 112W |
选中上述五列,View - Hide - All but Selected Region (Ctrl + Shift + H)。
NDWEH,可以看到各个序列中该五个位点的情况。

-
如何实现聚类? 感觉不行,要不为什么要用 USEARCH 和 MC-UPGMA program。
可以做PCA或者发育树。 -
如何实现筛选规则?Select 中似乎可以实现功能。
Select 可能并不能做这个任务。但是Jalview中可能可以筛选,通过一些自定义的特征。
下载期间看看别的项目。
Result Summary
可以下载输入、输出等各种文件。