论文解读:基于注意力的多标签神经网络,用于集成预测和解释12种广泛发生的RNA修饰
前言
最近的研究表明,通过转录后RNA(核糖核酸)修饰的表转录组调节对所有类型的RNA都至关重要。精确鉴定RNA修饰位点对于理解RNA的功能和调节机制至关重要。在这里,我们介绍了MultiRM,一种从RNA序列中整合预测和解释转录后RNA修饰的方法。MultiRM建立在基于注意力的多标签深度学习框架之上,不仅能同时预测12个广泛存在的转录组修饰(m6A、m1A、m5C、m5U、m6Am、m7G、ψ、I、Am、Cm、Gm和Um)的位点,还能返回对积极预测贡献最大的关键序列内容。重要的是,我们的模型从相关序列背景的角度揭示了不同类型的核糖核酸修饰之间的强关联。我们的工作提供了一种检测多种RNA修饰的解决方案,能够对这些RNA修饰进行综合分析,并更好地理解基于序列的RNA修饰机制。
一、
转录后RNA修饰增加了RNA分子的结构和功能多样性,并调节RNA生命的所有阶段。因此,精确鉴定RNA修饰位点对于理解各种RNA的功能和调节机制至关重要。已鉴定出100多种不同类型的RNA修饰,其中,N6-甲基腺苷(m6A)是最常见的真核mRNA修饰。M6A发生在新生的前mRNA上,调节其稳定性和翻译。它参与许多生物学过程,如昼夜节律钟、从幼稚多能性的分化和热休克反应。在癌症、乳腺肿瘤、胃癌、抗肿瘤免疫等疾病发病机制中也发挥着多种作用。除了m6A,还有许多具有重要生物学功能的RNA修饰。例如,n1甲基腺苷(m1A)可以阻断沃森-克里克界面,对tRNA稳定性和HIV-1复制至关重要。
到目前为止,已经提出了许多从初级RNA序列中计算机预测RNA修饰位点的计算方法,包括:iRNA工具包3–11,SRAMP12,D e e p P r o m i s e13,W H I S T L E14,Gene2vec15,M6 A atlass 16,R M D i s e a s e17,P E A18,P P P U S19,B E R M P20,m5Upred21和m6AmPred22。还特别关注了内含子、lncRNAs24as以及各种组织和细胞系中RNA修饰的预测。总之,这些工作极大地促进了我们对不同物种在不同条件下多种核糖核酸修饰类型定位的理解。然而,现有的方法受到以下限制。
首先,现有的研究大多只关注单一的RNA修饰类型,主要是m6A,而未能通过整合的预测模型同时支持多种RNA修饰。因此,对不同修饰之间相互作用的研究是有限的。主要由陈、林和周开发的iRNA toolkit 3–11是支持从RNA序列预测各种RNA修饰的最早也是最全面的方法,并已被广泛用作基准测试不同RNA修饰预测方法性能的金标准。
然而,iRNA工具包是以多项独立研究的形式提出的,每项研究都针对一项单独的修改。iMRM网络服务器旨在通过友好的网络图形用户界面同时支持五种RNA修改;然而,它仍然基于分别对应于五个RNA修饰的五个独立的二元预测因子,而没有考虑不同修饰之间的潜在相互作用。考虑到不同RNA修饰的内在生化和生物物理特性,为一种修饰类型建立的预测框架通常可以方便地移植到另一种修饰的预测中。因此,在多个RNA修饰上同时测试计算框架是有益和有效的。最近,通过利用生成对抗网络(GAN),MR-GAN方法被发展来预测八个RNA修饰。然而,支持的一些修饰可能是罕见的修饰,如m1G(仅29个位点)、m2G(仅59个位点)和D(仅162个位点)30,其在人类转录组中的广泛出现尚未得到证实。鉴于这种罕见的核糖核酸修饰有大量阴性(不可修改)位点,基于序列的预测在实践中可能会产生相当大比例的假阳性预测,应格外谨慎使用。
其次,大多数现有的工作依赖于来自单一来源的有限数量的数据(单一数据库或由单一实验生成的数据集),未能充分利用可用的表转录组信息。例如,m5U修饰的广泛发生先前已经通过两种不同的方法(miCLIP和FICC-seq)31报道的数千个m5U位点得到证实。尽管如此,MR-GAN只使用了30个位点进行训练,这可能会严重限制其对这种特定修改的预测能力。此外,先前已经报道了不同表位转录组分析技术之间的实质性差异,例如m5C32和ψ33。因此,利用从多种正交技术生成的数据来尽可能减少潜在的技术偏差至关重要。
第三,该领域的大部分工作,如SRAMP12and和imrm 29,侧重于预测精度,但未能对其预测结果提供清晰直观的解释。尽管一些现有的方法仔细地解释了他们训练有素的预测模型,据我们所知,没有一个现有的工作为他们的个人预测决策过程提供了深刻的见解。可解释的核糖核酸/脱氧核糖核酸模型的最新进展使得能够提取低级的有线电视新闻网核,并将其可视化为位置权重矩阵。然而,这些模式只能提供模糊的见解,尤其是对于多层脱氧核糖核酸,不能提供核苷酸水平的解释。然而,确定直接有助于正向RNA修饰预测的关键序列内容仍然很有意义,这将有助于我们理解单个RNA修饰位点的序列依赖性形成机制。
最后,还没有为某些RNA修饰类型(如m6Am)开发预测框架,尽管已经开发了其基础分辨率表转录组分析技术miCLIP,并且分析数据是公开的。
由于这些原因,有很强的动机利用最先进的深度学习技术来开发一个统一的预测框架,该框架通过集成从多种技术生成的数据集来支持多种核糖核酸修改。
我们在这里介绍MultiRM,一种基于注意力的多标记神经网络方法,用于整合预测和解释来自初级RNA序列(或相应的脱氧核糖核酸序列)的核糖核酸修饰。我们的模型支持12种RNA修饰类型,包括m6A、m1A、m5C、m5U、m6Am、m7G、ψ、I、Am、Cm、g m和Um。据我们所知,这些是唯一可以用现有碱基解析技术在转录组范围内进行描述的广泛发生的RNA修饰,这是RNA修饰对可靠的大规模预测的高度期望的特征。我们的方法的多标签体系结构能够容纳不同修改的共享结构,同时充分利用它们的不同特征。由于一些修改仍然比其他修改更丰富,为了处理多标签学习中的不平衡训练数据问题,使用了在线硬示例挖掘(OHEM)和不确定权重。一些被广泛采用的最先进的机器学习算法XGBoost37and和CatBoost38were也被实现为基准。重要的是,我们使用综合梯度(IG)和注意力权重来获得对训练的整体模型的洞察,并解释每个单独的预测。最后,开发了一个网络服务器,可以免费访问,为研究团体服务。链接: link.
二、结果
1.MultiRM框架
我们的框架使用深度神经网络预测了12种广泛发生的RNA修饰,如图1所示。给定一组碱基分辨率可修改的位点,MultiRM学习位点序列上下文和修改类型之间的映射。一旦这个映射被学习,注意机制和IG方法使我们能够解释模型,并提取序列内容。
对阳性预测贡献最大的是序列基序。所提出的使用多标签框架的集成模型也被认为有利于学习不同RNA修饰之间的潜在关联。
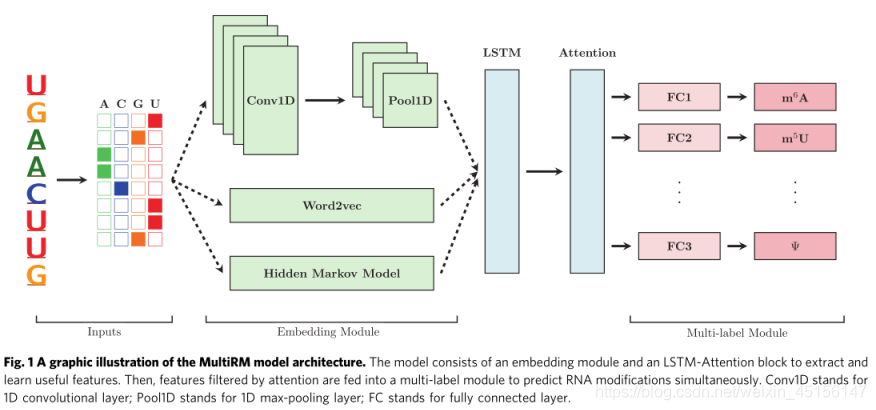
图1多模型体系结构的图示。该模型由一个嵌入模块和一个LSTM-Attention模块组成,用于提取和学习有用的特征。然后,通过注意力过滤的特征被输入多标签模块,以同时预测RNA的修饰。Conv1D代表1D卷积层;Pool1D代表1D最大池层;FC代表全连接层。
MultiRM由一个嵌入模块组成,该模块利用核苷酸之间固有的短程和长程相互作用来表示输入的RNA序列。嵌入的表示被馈送到LSTM层,以提取所有修改共享的底层序列特征。然后,注意机制使模型能够根据需要针对每种特定的修饰类型关注输入RNA序列的相关区域。最后,包含两个完全连接(FC)层的多标签模块同时预测多个可修改的位点。该框架使用由OHEM和不确定加权增强的交叉熵损失来训练。
2.多重性能
我们研究的主要目的是建立一个可解释的预测因子,在从一级RNA序列中识别多种广泛存在的RNA修饰方面达到最先进的准确性。
我们首先尝试根据AUCb(b代表二进制)来优化输入序列的长度。AUCbis基于每个修饰的阳性和相应阴性样本(例如,m1A的A)计算的ROC曲线下面积。使用Word2vec嵌入,我们以21-bp、51-bp和101-bp的RNA序列作为输入来评估我们的多标签模型。
如表1所示,51-bp序列的输入获得了所有修饰的最佳平均性能。
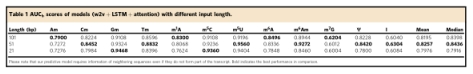
值得一提的是,输入序列的51-bp也是XGboost方法的最佳值(补充表1)。
随后,为了解决不平衡的训练数据问题,我们在优化的多标签模型上实现了OHEM、不确定性加权(UW)和聚焦损失,并用51-bp的输入序列测试了它们的性能。如表2所示,基于优化模型的AUCb值得到了改善。OHEM和UW都有利于一些修改。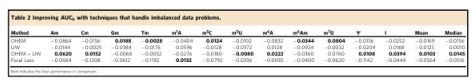
例如,OHEM将m7G的预测值提高了0.08个澳柯布核心,UW将澳柯布核心的预测值提高了0.06个。在我们将两者结合后,它帮助平均AUCbscore提高了0.0145。
通过优化设置(51-bp输入,UW+ OHEM),我们将新开发的方法MultiRM与基线方法和其他嵌入技术进行了比较。要比较的每个模型的优化超参数可以在补充表2中找到。如表3所示==,新提出的方法MultiRM获得了最佳的平均和中值性能,AUCbof分别为0.8361和0.8581,并且在所考虑的12种RNA修饰中的6种(Am、Cm、m5U、m6A、ψ和I)上获得了最佳性能,在测试的5种方法中平均排名为1.667。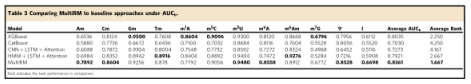
广泛采用的XGBoost算法在四种修改(Gm、m1A、m5C和m7G)上获得了最佳性能,并在所考虑的五种方法中以0.8035和0.8122的AUCbof获得了平均和中值性能,平均排名为2.25。
随后,我们基于它们各自的ROC曲线,为每个具有最大G均值41值的修改选择最佳阈值。计算了相应的性能评估指标,包括灵敏度(Sn)、特异性(Sp)、准确性(Acc)和每个修改的马修斯相关系数(MCC),并在表4中提供。
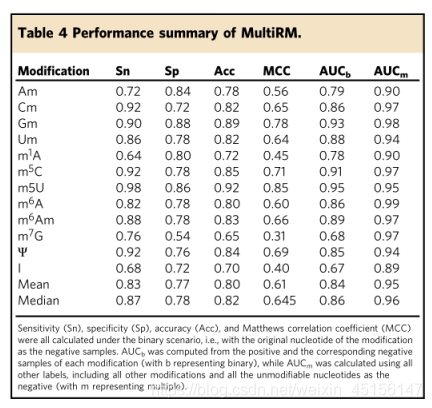
在补充图1和2中提供了MultiRM方法的精度和召回率(PRs)曲线和接收器工作特性曲线(ROCs)曲线。多标签分类42方案下的MultiRM性能指标见补充表3。
3.对模型的解释
到目前为止,结果强调了我们的方法在分类方面的性能。为了深入了解预测背后的驱动特征,我们应用了能够提供模型可解释性的技术,以识别对预测核糖核酸修饰有重要意义的关键输入序列内容(参见“方法”)。关注内的序列内容对RNA修饰的影响更大,这些区域内的突变更容易导致RNA修饰位点的获得或丢失,如补充图3所示。
此外,我们汇总并检查了在MultiRM模型中起关键作用的共识主题。有趣的是,其中许多与传统的基序发现方法dreme43和STREME44所揭示的序列模式相匹配。
为了进一步量化通过MultiRM和DREME/斯特雷梅获得的模体之间的相似性,模体比较工具TOMTOM45was被应用来产生p值。足够小的pvalues表示一定程度的一致性(见图2)。
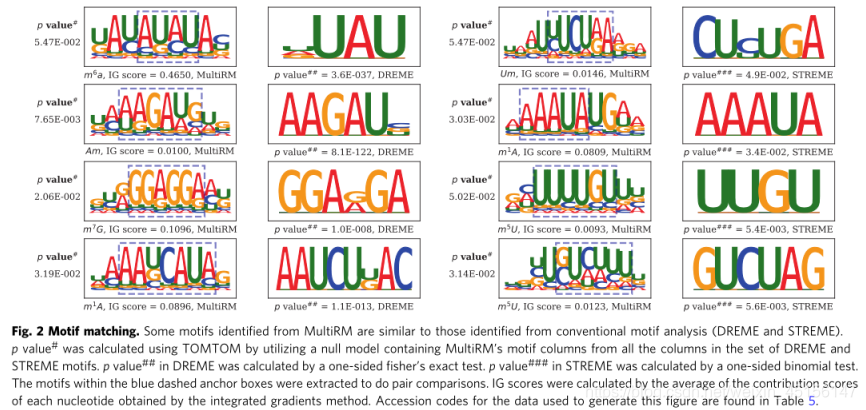
图2模体匹配。从MultiRM中鉴定的一些基序类似于从常规基序分析中鉴定的基序(DREME和斯特雷梅)。p值#是使用TOMTOM通过利用包含来自DREME和STREME基序集合中所有列的MultiRM基序列的空模型来计算的。DREME的p值# #是通过片面的费雪精确检验计算出来的。STREME中的p值# # #是通过单侧二项检验计算的。蓝色虚线锚框内的图案被提取出来进行配对比较。通过积分梯度法获得的每个核苷酸的贡献分数的平均值计算免疫球蛋白分数。
用于生成该数字的数据的登录代码见表5。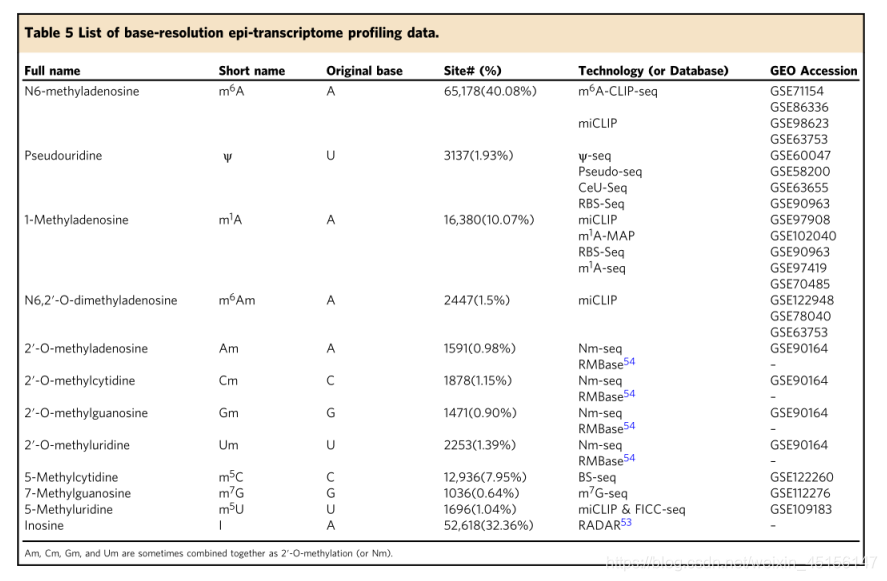
值得一提的是,MultiRM使用注意力权重最高的6聚体构建了一个RNA修饰的基序,而这并不一定包含RNA修饰位点本身。这与大多数新的模体发现算法如dreme 43和STREME44是一致的。
所提出的集成模型的一个主要优点是能够学习不同核糖核酸修饰之间的潜在联系。先前表明,在蛋白质的不同翻译后修饰之间以及在不同组蛋白和染色质修饰之间存在明显的进化和功能串扰=。可以想象,在不同的核糖核酸修饰中,这种关联也可能存在于表转录组层。为了更好地理解不同RNA修饰之间的内在共享结构,我们提取了注意机制内前馈神经网络的权重。这些权重是分别对应于12个RNA修饰的12个载体,并且与所提出的模型的所有其他组件一起被联合学习。计算每对载体的皮尔逊相关(ρ)以揭示由综合预测模型揭示的两个任意RNA修饰的相关性。一个令人惊讶的发现是,所有的RNA修饰都显示出彼此之间强而显著的正关联,包括那些来源于不同核苷酸的修饰(见图3)。
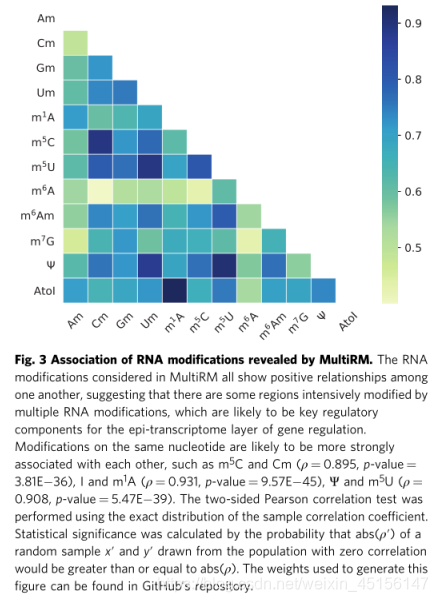
图3由MultiRM揭示的RNA修饰的关联。MultiRM中考虑的RNA修饰都显示出相互之间的正关系,这表明有一些区域被多个RNA修饰强烈修饰,这些区域可能是基因调控的外转录组层的关键调控成分。同一个核苷酸上的修饰很可能更强地相互关联,例如
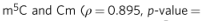
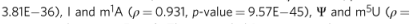
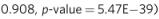
使用样本相关系数的精确分布进行双边皮尔逊相关检验。统计显著性是通过从零相关总体抽取的随机样本x′和y′的abs(ρ′)大于或等于ABS(ρ)的概率来计算的。用于生成这个数字的权重可以在GitHub的存储库中找到。
这表明存在被多种RNA修饰强烈修饰的区域,这些区域可能是基因调控的外转录组层的关键调控成分。重要的是,这些关键调控区的序列特征在很大程度上被不同的RNA修饰(包括那些修饰不同核苷酸的修饰)所共享,并被我们的模型成功捕获。最强关联的修饰源自相同的核苷酸,如m5C和Cm (ρ= 0.895,p值= 3.81 e-36)、I和m1A (ρ= 0.931,p值= 9.57 e-45)、ψ和m5U (ρ =0.908,p值= 5.47 e-39)。
值得注意的是,m6A仅显示出与其他修饰的轻微关联,这意味着它作为最丰富的mRNA修饰在转录后调控中的相对特殊的作用。
还值得注意的是,上述分析没有考虑RNA修饰的上下文特异性(或RNA修饰在不同生物条件下的动力学)。它并不直接表明不同的核糖核酸修饰应该在相同的生物环境中共同发生,或者它们具有协同效应,尽管后者极有可能是我们在组蛋白修饰的表基因调控中所看到的。
为了进一步验证上述发现,我们计算了两个任意核糖核酸修饰之间的成对距离,并将其与随机距离进行了比较。虽然我们不能完全排除实验偏差的可能性,例如,聚腺苷酸选择,但我们确实在本分析中考虑的大多数核糖核酸修饰中观察到强烈的聚集效应。很明显,两个任意RNA修饰之间的距离可能比随机的更近(补充图4)。
4.MultiRM网络服务器
构建了一个具有友好图形用户界面的网络服务器,以便在研究社区之间适当地共享所构建的MultiRM模型。它以核糖核酸序列为输入,返回预测的核糖核酸修饰位点以及驱动阳性预测的关键序列内容(图4)。

图MultiRM网络服务器的输出。网络服务器支持对来自RNA(或DNA)序列的12种广泛存在的RNA修饰(m6A、m1A、m5C、m5U、m6Am、m7G、ψ、I、Am、Cm、Gm和Um)进行站点预测和结果解释。该图显示了预测的RNA修饰位点(上图)和对输入序列的第101个和第150个核苷酸之间的阳性预测贡献最大的序列成分(下图)。RNA修改的预测概率、统计显著性和注意力分数都可以作为单独的文件从网络服务器下载。在第123个核苷酸处,来自U的多个RNA修饰被预测为p值小于0.05,这在一定程度上反映了之前揭示的不同RNA修饰之间的关联(见图3)。
通过与阴性位点产生的结果进行比较,也提供了预测的统计学意义。对于在线预测,当作业完成时,可以选择向提供的电子邮件地址发送通知电子邮件。对于离线预测,可以下载经过训练的MultiRM模型和Python代码,以便在本地计算机上使用。
讨论
在这项工作中,我们开发了一个多标签模型,可以同时预测十二种广泛发生的核糖核酸修饰,并呈现对预测贡献最大的关键序列成分。为了充分利用输入序列的固有结构,我们在我们的模型上实验了三种不同的嵌入技术,并发现Word2vec极大地增强了它的预测能力。我们还发现,输入更长的核糖核酸序列不一定会导致更高的预测精度。为了处理不平衡标签问题,我们实现了OHEM和不确定加权策略。令人鼓舞的是,我们发现我们的MultiRM模型的整体性能比经典的机器学习模型XGBoost和一些最先进的多标签学习者要好。
我们仅使用从多种正交技术和多项研究中生成的高质量表转录组谱,仔细整理了预测模型的训练和测试数据。鉴于多种技术的表转录组分析结果之间的差异,这种补救措施对于确保预测器的稳健性和可靠性至关重要。
为了最好地共享新构建的核糖核酸修饰位点预测因子,构建了一个网络服务器。除了友好的用户界面和详细的在线使用文档外,它还包含经过培训的MultiRM模型和Python代码,这些代码可以下载到本地计算机上进行命令行使用。
虽然MultiRM能够预测12种不同类型的核糖核酸修饰,但目前仅限于人类,尚未扩展到其他模式生物。这主要是因为缺乏其他生物的碱基分辨率表转录组谱数据。在其他物种(如小鼠和酵母)上测试MultiRM的性能,以及在将来有此类数据时纳入新出现的转录组修饰(如ac4C48、49和hm5c 50)将会很有趣。
需要注意的是,MultiRM目前没有考虑不同RNA修饰的不同丰度。因此,即使在相同的设置(p值截止)下,假阳性预测的比例在较丰富的RNA修饰(如m6A)和较不丰富的RNA修饰(如m1A)之间也有很大差异,即较不丰富的RNA修饰的假阳性率要高得多。这个问题部分是由于现有生物技术在分析核糖核酸修饰方面的一致性有限。例如,尽管亚硫酸氢盐测序报告了10,581个以上的m5C位点,但AzaIP和miCLIP分别报告了617个和1084个m5C位点32,这可能是由于它们不同的技术偏好和敏感性。我们在补充图5中提供了建议的MultiRM模型在不平衡样本量上的性能指标,这反映了我们目前对现实世界中修改分布的了解;但是,需要注意的是,收集的RNA修饰位点的数量受到生物技术的检测灵敏度和可用实验数据的强烈影响,而不是它们的真实丰度。当这些核糖核酸修饰的总体丰度更容易获得时,未来将需要更可靠的错误发现率控制。
我们的模型第一次揭示了所有12种RNA修饰在序列偏好方面的正关联。研究一般核糖核酸修饰和表转录组调控的关键调控区域应该是当前感兴趣的。同样令人感兴趣的是它们在不同生物条件下的动态串扰,这要求在这些数据更加丰富的情况下,对条件特异性表转录组分布进行综合预测。例如,通过在多标签学习框架下扩展相关研究。
以前,由于缺乏匹配的生物条件下的上转录组数据集,多个RNA修饰的串扰主要是通过分子水平研究的相关RNA修饰酶的表达水平。
虽然基于酶的分析可以部分揭示不同RNA修饰之间的相互作用,但需要注意的是,已知的酶基因除了书写或擦除RNA修饰外,还具有多种生物学功能,这可能会污染结果。相比之下,直接分析表转录组可能更可靠。随着深度学习方法的进步,应该有可能更深入地挖掘和揭示协同RNA修饰相互作用及其软序列语法,正如在协同转录因子调控中所做的那样。
方法
1.原始数据和预处理
为了训练和测试的目的,RNA修饰位点预测模型的开发通常需要基本分辨率的转录组范围的谱数据。对数据集进行了选择,对从多项研究中得出的和用不同技术生成的数据集进行了优先排序。没有使用由有缺陷的技术(如普通的亚硫酸氢钠测序)或方法(如普通的MeRIP-seq结合模体搜索)产生的数据。
如表5所示,我们最终从12种不同类型的RNA修饰(m6A、m1A、m5C、m5U、m6Am、m7G、ψ、I、Am、Cm、Gm和Um)的15种不同碱基分辨率技术中获得了20个表转录组图谱。据我们所知,我们的数据涵盖了所有广泛的核糖核酸修饰,这些修饰可以在转录组范围内以碱基分辨率进行分析。特别注意构建预测因子最可靠的阴性对照数据(未修饰的核苷酸)。从含有阳性位点的同一转录物的未修饰碱基中随机选择阴性位点。
肌苷(I)位点是从RADAR数据库中收集的,而Am、Cm、Gm和Um位点是从由Nm-seq报告的位点补充的RMB ase中收集的。当存在代表修饰的基序,即m6A的DRACH基序和m6Am的BCA基序时,该基序被用来进一步限制相应修饰的正负数据。对于m6A,因为需要检查使用各种技术的现有大量碱基分辨率研究的可靠性,所以之前确定的总共87,616个m6A位点(补充表4)只是通过将其从阴性m6A位点中排除而间接使用。
最终,收集了超过30万个站点。我们将每个类中的RNA序列(对应于一种RNA修饰)分成三个集合,即训练集、验证集和测试集。这里,训练集在不同类别(修饰类型)之间是不平衡的,即对于不同的RNA修饰,位点的数量是不同的,而验证集和测试集分别具有大小为150和50的平衡样本。通常,超参数基于验证集进行优化,而报告的最终预测性能是在测试集上实现的。传统上,K倍交叉验证用于减轻许多机器学习问题中的过度拟合,尤其是对于那些训练数据较少的问题。这是因为使用K-fold来验证模型可以更好地估计模型的结果将如何推广到独立的数据集,特别是在有限的数据集内,其中小的测试不能反映数据的整个分布。然而,在我们的场景中,我们总共有大约30万个训练数据。因此,5%的测试/验证集已经给了我们一个很好的估计。
2.Embeddings
为了开发高精度的计算方法,明智地表示或嵌入序列数据至关重要。假设我们有原始数据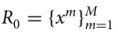
其中M是序列的数量,每个序列
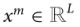
是一个RNA序列。
每个实体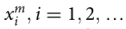
位置L的i从字母表中取值
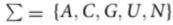
从一个长度为l的序列
我们考虑了以下三种方案来将RNA序列R0映射到嵌入空间R’。
传统上,one-hot是一种简单但非常有效的表示序列数据的编码方法。
对于每个RNA序列
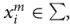
我们把它绘制成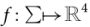
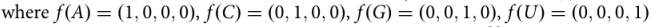
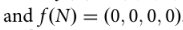
之后,R0变成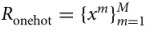
RNA序列里的每一个
隐马尔可夫模型由于能够捕捉远程交互,因此适用于序列数据建模。Seq2vec58已经成功应用,它使用神经网络来加速隐马尔可夫模型中的参数化。
它构建了一个非线性特征嵌入f : ∑7!RNA将每个RNA序列XM转化为一个n维向量。
此外,f是两个非线性算子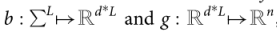
的组合
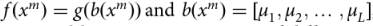
其中每个μl总结了xm中不同位置的潜在远程交互,g将聚合交互信息和整个数据集的固定维度嵌入。
在我们的工作中,我们在递归模块和多标签模块之前加入了HMM层,形成了从训练到预测修改位置的端到端解决方案。
自从2013年由Mikolov首次发明以来,word 2 vec 59增强了各种NLP任务的性能。作为一种统计语言模型,它遵循skipgram和连续词包(CBOW)架构,并使用神经网络来学习基于上下文关系的词嵌入。我们通过将每个RNA序列视为一个句子,并将k个连续的RNA核苷酸(k-mer)视为该句子中的单词来训练我们自己的RNA嵌入。在数学上,我们定义了从单个核苷酸到k-mers载体表达的映射,如下所示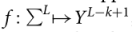
然后将其输入神经网络以获得二维嵌入
已经在基因2 vec 15中证明,3-mers在m6A位点上具有最佳的预测性能。因此,在我们的工作中,我们遵循3-mers惯例来嵌入我们的输入数据。更具体地说,一个3-nt滑动窗口以1-nt的步长在1001-nt个样本序列上移动,以创建999个重叠单词的序列。每个单词对应于所有可能的3-mer组合集合中的一个索引(在我们的训练数据中有104种不同的组合)。然后,利用Gensim package60with实现了Word2vec,它有一个五字长的相邻词窗口,学习内在关系,生成一个300维特征向量。最后,每个嵌入的RNA序列被转换成999乘300的矩阵。
2.模型设计
在这项工作中,两种类型的DNN结构,卷积神经网络(CNNs)和递归神经网络(RNNs)被用来学习RNA修饰的序列特征。具体来说,实现了长短期记忆(LSTM)来解释特征的可能的长期依赖性。
该模型主要由三部分组成(见图1)。
第一个模块是嵌入模块,它以RNA序列的一热编码为输入,通过三种不同的嵌入技术进行嵌入。然后,每个嵌入被馈送到LSTM和巴林注意力层39。聚集LSTM层内部的隐藏状态ci和所学习的特征表示yi,以获得每个目标类别的注意力权重。然后通过计算yi和每个注意权重的内积得到12个不同的上下文向量。预计这些向量可以很好地压缩每个预测分支所需的重要信息。多标签模块包含12个具有ReLU激活功能的并行FC层,将每个上下文向量同时映射到每个修改的概率。脱落层用于减轻过拟合。通过对不同任务的二元交叉熵损失进行加权,优化了整个模型。
重要的是,为了评估在我们的模型中使用的嵌入方法的贡献,我们利用了嵌入的三种变体。对于一次性编码,美国有线电视新闻网提前添加并作为嵌入模块的一部分执行。由于RNA序列的原始一热编码是稀疏输入,CNNs将有助于以密集的方式提取序列模式,并生成这些基序的高维表示。此外,池层用于修剪信息量较少的要素。
值得注意的是,不同标签(类)之间的数据高度不平衡,即在更丰富的修饰(如m6A和I)和不太丰富的修饰(如Am和Gm)之间,位点的数量差异很大。传统上,这个问题有两种解决方式。一种方法是改变原始的不平衡数据,使用像SMOTE61这样的过采样算法来平衡它。另一个潜在的更有效的方法是在网络的末端权衡每个类的损失。由于我们的输入是严格生成的核糖核酸序列,生成人工核糖核酸序列可能会降低原始数据集的可信度。因此,我们把重点放在第二个选择上。作为一个基准,我们首先基于样本的有效数量为每个任务使用一个恒定的权重。随后,我们通过在训练过程中学习每个任务的权重来设计我们的多标签模型,并且只反向传播具有较高损失的样本(OHEM 35),以便它可以在整个训练过程中联合优先化任务和样本。我们还测试了focal loss的性能,它基本上在常见的二元交叉熵损失上降低了分配给分类良好的样本的损失。
2.评估指标
在训练集上训练之后,我们在验证集和测试集上评估我们的模型。分类性能由接收器操作特性(ROC)表征,并由ROC曲线下的区域(表示为AUROC或AUC)评估,AUC是反映模型性能的非参数指标。
具体来说,我们计算了两种类型的AUC: AUCband AUCm。从每个修饰的阳性和相应的阴性样本计算AUCbwas代表二进制),而使用所有其他标记计算AUCmwas,包括所有其他修饰和所有不可修饰的核苷酸作为阴性(m代表倍数)。因此,auckbre表示一个特定修饰相对于其原始不可修饰碱基的预测性,例如m1A对A,而auckbre表示所有其他情况,包括其他修饰和不可修饰碱基。尽管AUCmis为多标签学习任务提供了匹配的评估方案,但AUCbwas提供了与现有作品保持可比性的方法,其中大多数作品以不可修改的原始核苷酸作为阴性样本进行二元分类。
值得注意的是,在计算AUCm时,也考虑了非原始核苷酸。因为根据特定修饰的非原始核苷酸做出正确的阴性预测是很简单的
(例如,核苷酸C不能形成m6A),AUCmis显著高于AUCb。
广泛采用的评估指标,包括灵敏度(Sn)、特异性(Sp)、准确性(Acc)和马修斯相关系数(Mcc)也用于评估预测性能,可表示为:

其中TP代表真阳性样本,TN代表真阴性样本,FP代表假阳性样本,FN代表假阴性样本。基于最大化G均值40为每个类别选择最佳阈值,以对特定修改的正样本和负样本进行分类。这些指标都是在二元分类方案下计算的,使用特定基因修饰的阳性和阴性样本,因此与大多数现有RNA修饰位点预测工作中的报告表现相当。AUCb由于其非参数特征和与现有文献中相关工作的报告表现的可比性,被用作主要评价指标。
3.统计显著性
预测概率的统计显著性通过p值的上限来评估,表明在相同核苷酸的所有出现中观察到的概率有多极端。根据推定的RNA修饰位点的相对排名计算,即如果只有1%的核苷酸报告概率大于特定位点,则该位点的p值上限为0.01。这被用作预测的截止值。但是,需要注意的是,截止控制仅控制第一类错误。即使有相同的p值截止值,在较丰富的RNA修饰和较不丰富的RNA修饰之间,假阳性预测的比例仍然有很大的不同。
4.解释
除了对核糖核酸修饰的准确预测之外,掌握模型预测背后的想法通常也很有吸引力。在我们的模型中,我们使用了注意力权重和IG来直观地解释模型是如何做出具体决策的。具体来说,我们在做出不同的预测时,专注于我们的模型最重视的方面,并通过注意力权重和免疫球蛋白获得了在做出积极预测时贡献最大的核苷酸。
Bahdanau attention40was最初是作为处理序列到序列模型的长输入序列的解决方案引入的。在这里,我们通过将输入的RNA序列映射到12个上下文载体,将其移植到我们的方法中。由于它可以访问整个输入的核糖核酸序列,并且能够从序列中挑选出特定的元素来产生输出,因此该机制使模型可以根据需要自由地关注和或多或少地关注每个预测任务的输入核糖核酸序列的相关核苷酸。因此,通过可视化表示每个预测任务中输入核糖核酸序列的每个核苷酸的权重的注意力权重,我们可以在做出不同预测的同时识别模型中输入序列的最关键部分。
通过计算输出神经元相对于其输入的梯度,基于梯度的属性方法可以反映输入特征通过网络对特定输出的贡献。在我们的工作中,我们使用了一种称为IGs39的归因方法。这里,感兴趣的目标神经元是每次修改的分类层。当输入沿着从基线或参考到输入的线性路径变化时,IG计算输出神经元的平均梯度。它测量每个输入对修饰预测的贡献,并为输入序列中的重要核苷酸分配更高的分数。基于每个输入核苷酸位置的贡献分数,我们将属性图可视化为序列标识,其中高度表示该位置在预测中的重要性。正向核苷酸的大小代表了预测核糖核酸修饰出现的重要水平。
特定RNA修饰的每个输入序列的属性图的可视化不仅给出了做出积极预测时的重要位置,还揭示了其相应修饰的潜在目标基序(或重复模式)。为了计算对每个RNA修饰贡献最大的共有基序,在之前的研究64之后,我们累积了对应于所有预测分数在前10%的真阳性样本的每个位置的归因值。然后,对于每个样本,我们通过在期望长度的滑动窗口中取最高的平均分数,移除其邻域,并再次重复下一个基元,来搜索整个属性图中的前k个基元。经过多次序列比对,UMAP65was用于嵌入顶级模体,DBSCAN66was用于聚类这些嵌入模体。
最后,我们通过计算它们的脉宽调制将这些图案聚集在每个簇中,并使用序列标志将其可视化。
5.基线性能
由于基于树的分类算法对于许多生物信息学问题29,56通常具有最佳的现成准确性,因此在本工作中,我们将我们的模型与两个梯度增强决策树进行了比较,即XGBoost37and和CatBoost38。XGBoost已被广泛用于生物信息学预测。这里使用CatBoost是因为它内置了处理分类变量的技术,从而避免了在这个问题中将核苷酸转换为单热编码的额外步骤。梯度增强决策树被用作所有13个类别的多类别分类器,包括12个广泛发生的核糖核酸修饰和不可修饰的核苷酸类别。为了产生最佳结果,我们通过协调体面搜索每种方法的超参数,优化选择在补充表2中提供。
6.基于注意力的DNN(脱氧核糖核酸)
我们在101-bp、51-bp或21-bp的短核糖核酸序列上训练了基于注意力的脱氧核糖核酸(见模型设计小节)。对于Word2vec(参见嵌入部分),我们对1001 BP序列上的RNA 3-mers进行预训练,然后从中提取相应长度的短RNA子序列。对于美国有线电视新闻网和隐马尔可夫模型,我们以端到端的方式设计它们,以核糖核酸序列的一热编码为输入。在训练过程中,我们使用128的小批量作为输入,并在1台英伟达RTX 2080Ti上进行了100多个时期的训练。此外,我们在训练中使用了亚当优化器67和128的小批量。同时,对合适的模型实施指数和余弦退火68学习率衰减,并在五个连续时期内泛化损失增加时引入早期停止69,以防止对训练数据的过度拟合。最后,验证集用于为单个模型搜索最佳超参数,测试集用于在各种模型中选择性能最佳的模型。