蛋白质结构预测
写在前面
开源链接: https://github.com/deepmind/alphafold
论文链接:https://www.nature.com/articles/s41586-021-03819-2_reference.pdf
一、what is embedding?
embedding在深度学习领域的最初切入点是manifold hypothesis(流形假设):自然的原始数据是低纬的流形嵌入于(embedding into)原始数据所在的高维空间(比如一个三维球体上的每一个点都可以用二维经纬度来表示)。在深度学习中,embedding是将高纬的原始数据(图像,句子)映射到低维流形(比如映射为一个表征向量)并使其变得可分。
二、基础知识
1.蛋白质结构
蛋白质预测任务的定义:输入氨基酸序列,预测其三维结构坐标(即蛋白质中每个原子的三维坐标)。碳基生物中的NH2-CH-COOH是固定的,而R不固定,常见的可以组成生命体的R有20种,所以可以简单将他们当作一个21个词的词典做去做embedding(其余不可组成生命的R统一记作UNK。
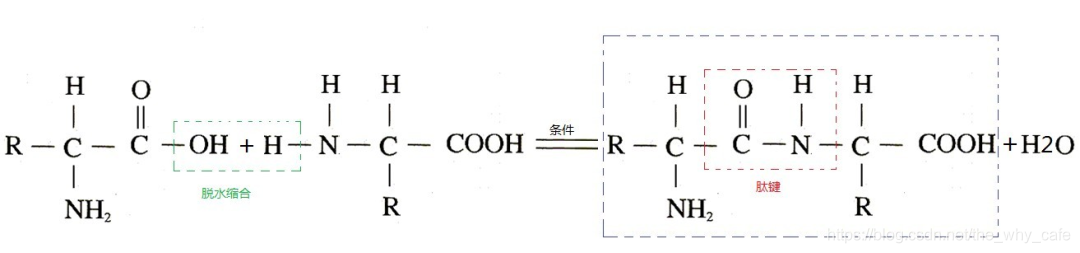
氨基酸的一级结构:氨基酸的顺序
氨基酸的二级结构:折叠后规则的片段(比如周期性结构构象,肽键的alpha螺旋上升,通过氢键形成的beta折叠等)
氨基酸的三级结构:完整的三维坐标
氨基酸的四级结构:两个以上的蛋白质通过分子对接形成的复杂结构。
2.MSA(Multiple Sequence Alignment)
将相同的或相似的氨基酸序列对齐,进行序列对比,对相似程度进行打分。
可以通过补空位,左右移动位置等方式,使得匹配的全剧得分达到最高。
MSA的目的是通过共进化分析找到保守区域和其他特征。保守区域的突变往往影响蛋白质的功能而影响生物体的存活。
三、研究蛋白质结构的重要性
1.分析蛋白功能
生物的基因通过表达为蛋白质等生物分子来进行各种生命活动。分析预测蛋白质的结构可以帮助我们了解其活性区间和靶点。
2.制药需求
新药的研制流程包括,首先确定病的成因靶点,即哪个蛋白质/哪个基因出了问题,需要去抑制这个靶点还是去激活这个靶点。然后是针对靶点进行对应小分子药物的设计,比如模拟体内的小分子再修改其功能,修改已有相似药物的功能,根据靶点的三维结构去设计对应结构可以结合上的小分子,或者对已知可合成的小分子进行高通量筛选。之后是对这一步中所有可能的小分子进行缩小范围,比如判断其毒性,跨模型,在体内会残留多久,会不会对其他器官/蛋白质产生损害等等。
3.节约大量制药资金和时间
AlphaFold2的精度可以达到和冷冻电镜精度相似。
4.进一步了解生命体结构和历史。
MSA中,同源的蛋白质的氨基酸有多大差异,就往往代表他们从同一个祖先分离出来之后独立进化了多久,我们可以分析氨基酸序列来追溯进化的历史,找到那些还未被发现的共同祖先;序列建模出三维结构,我们进一步可以建模病毒(大多病毒就是DNA/RNA外面罩一个蛋白质壳子)。
四、AlphaFold1
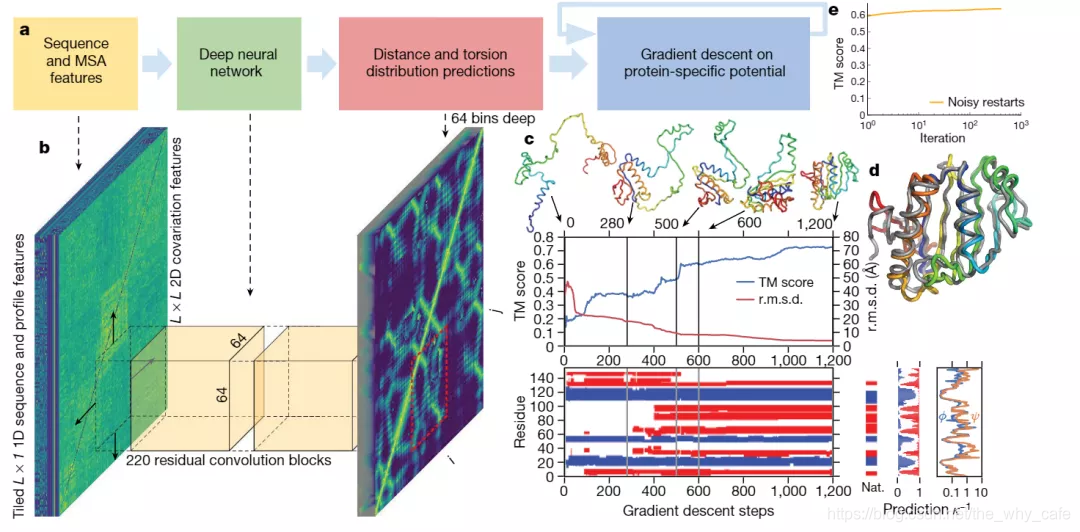
1.分析蛋白功能
输入MSA feature (feature为传统算法得出的已知的feature),输出氨基酸两两间的距离。
五、AlphaFold2
1.算法实现
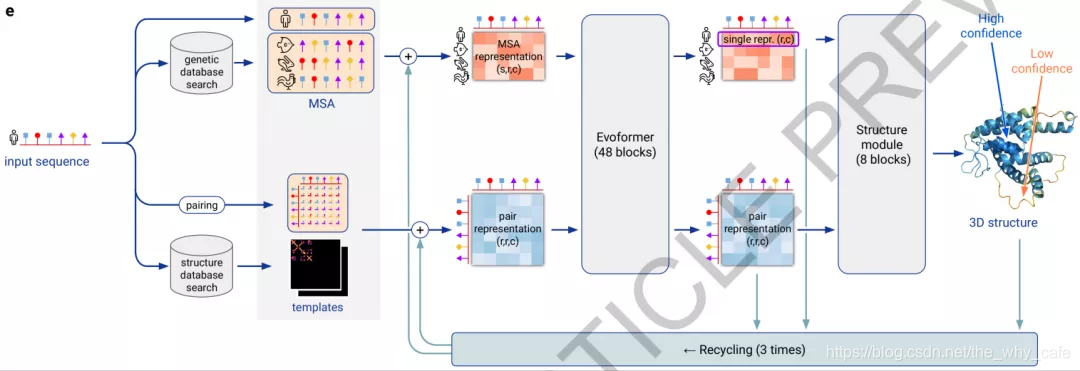
evoformer:输入MSA,已知的氨基酸序列,输出MSA信息和pairwise features
(残基对关系)
structure module:去掉MSA中的其他氨基酸序列,只保留目标序列,输入pairwise features,计算更新backbone frames(碳、氧、氮的链,即骨架链),预测所有氨基酸的方位和距离,肽键的长度,氨基酸内部的扭转角等等。