重要参数、属性及接口
-
criterion
回归树衡量分支质量的指标,支持的标准有三种:
1)输入“mse”使用均方误差(mean squared error),父节点和子节点之间的均方误差的差额来作为特征选择的标准,通过使用叶子节点的均值来最小化L2损失。
2)输入"friedman_mse"使用费尔德曼均方误差,这种指标使用费尔德曼针对潜在分支中的问题改进后的均方误差。
3)输入"mae" 使用绝对平方误差,使用节点的中值来最小化L1损失
其他属性也包括feature_importances_,接口有apply,fit,predict,score等核心的。
M S E = 1 N ∑ i = 1 N ( f i ? y i ) 2 MSE=\frac{1}{N}\sum_{i=1}^{N}(f_i-y_i)^2 MSE=N1?i=1∑N?(fi??yi?)2在回归树中MSE既是分支质量的衡量标准也是回归树回归质量的标准,在回归中,MSE越小越好,而接口score返回的是R2,R2定义是:
R 2 = 1 ? u v , u = ∑ ( f i ? y i ) 2 , ( 残 差 平 方 和 ) v = ∑ ( y i ? y h a t ) ( 总 平 方 和 ) R^2=1-\frac{u}{v},u=\sum(f_i-y_i)^2,(残差平方和)v=\sum(y_i-y_{hat})(总平方和) R2=1?vu?,u=∑(fi??yi?)2,(残差平方和)v=∑(yi??yhat?)(总平方和),其中y-hat是所有标签的平均值。
虽然均方误差恒为非负,但考虑到作为损失,变成了负的。
交叉验证
交叉验证是用来观察模型的稳定性的一种方法,将数据划分为n份,依次使用其中的一份作为测试集,其他n-1份作为训练集,多次计算模型的准确性来评估模型的平均准确程度。(这吃瓜的时候学过,现在听到居然毫无印象。。。。)
相关操作:
from sklearn import tree
# 交叉验证的库
from sklearn.medel_selection import cross_val_score
# 随便搞一个数据集
from sklearn.datasets import load_boston
# 实例化一个回归模型
regressor = tree.DecesionTreeRegressor(random_state=0)
# 参数分别是实例化的模型,样本数据矩阵,样本标签,验证次数,衡量标准(这里是均方误差的相反数)
cross_val_score(regressor, boston.data, boston.target, cv=10,scoring='neg_mean_squared_error')
回归树的例子
用回归树拟合正弦函数曲线
import numpy as np
import matplotlib.pyplot as plt
from sklearn import tree
rng = np.random.RandomState(1) # 随机数种子
X = np.sort(5*rng.rand(80,1), axis=0)
# y数据必须是一维的,用ravel()展平
# numpy数组中一维数组不分行和列
y = np.sin(X).ravel()
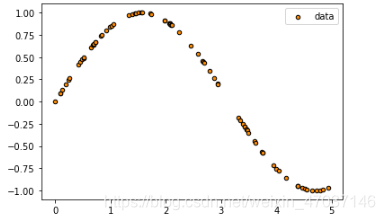
plt.figure()
# plt.scatter专门画散点图
plt.scatter(X, y, s=20, edgecolor='black', c='darkorange', label='data')
plt.legend()
# np.random.rand(数组结构),生成随机数组的函数
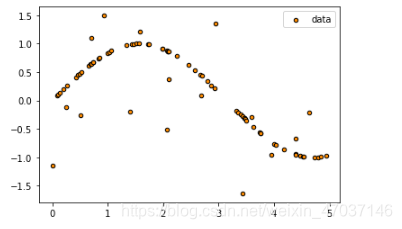
# 加噪声
y[::5] += 3*(0.5-rng.rand(16))
plt.scatter(X, y, s=20, edgecolor='black', c='darkorange', label='data')
plt.legend()
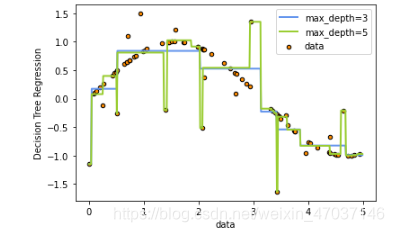
regressor1 = tree.DecisionTreeRegressor(max_depth=3)
regressor2 = tree.DecisionTreeRegressor(max_depth=5)
regressor1.fit(X, y)
regressor2.fit(X, y)
X_test = np.arange(0.0, 5.0, 0.01)[:, np.newaxis]
y_1 = regressor1.predict(X_test)
y_2 = regressor2.predict(X_test)
plt.figure()
plt.scatter(X, y, s=20, edgecolor='black', c='darkorange', label='data')
plt.plot(X_test, y_1, color='cornflowerblue', label='max_depth=3', linewidth=2)
plt.plot(X_test, y_2, color='yellowgreen', label='max_depth=5', linewidth=2)
plt.xlabel('data')
plt.ylabel('Decision Tree Regression')
plt.legend()
plt.show()