前言
本文记录了QuantifyPoly(A)1包使用过程中的一些问题
一、文章内容
这个R包出自老牌生信期刊Briefings in Bioinformatics上的文章 QuantifyPoly(A): reshaping alternative polyadenylation landscapes of eukaryotes with weighted density peak clustering,两位通讯作者分别为厦门大学的助理教授叶从庭和福建农林的副教授林俊城(曾在厦大做博后)。作者中还有李庆顺老师,李老师是植物多腺苷化研究的大拿。此前厦大已经在这个领域发表了不少文章,包括很多研究型文章、一些生物信息学方法以及植物APA数据库等,可以说已经形成一套完整的Poly(A) site(PAS)使用的分析流程。但本文依然是对先前方法的一个突破。
很多建库方法都能帮助研究者单碱基精度地识别polyA site的位置,但实际上合并相邻的位点成poly(A) cluster(PAC)、考虑PAC的使用才更合理。虽然我们常说poly(A) Site,但这个概念只在讨论某一次转录事件时有意义。但实际上,即使是对于编码同一蛋白的转录本的转录过程,多腺苷化并不一定精确的发生在某一个碱基上,发生的区间长度可能在几个碱基到十几个碱基之间。对于注释较好的模式物种,这些位点通常就在注释的3’端附近。(从这个角度看UTR似乎是个很精巧设计,一个缓冲区,转录终止即使不那么精确也能翻译出正确的蛋白)
过去合并cluster一般仅仅依靠PAS之间的距离(比如距离在24个碱基以内就合并),因而合并之后PAC大小会分布在一个很广的范围里,比如数据库plantAPAdb中的PAC大小从1个碱基到好几百的都有。很多大的PAC明显是不合理的。在这篇文章中,研究者们就提出了一种新的算法,不仅通过长度,还要考虑每个点上的read count,更准确地去定义cluster。
当然,这篇文章的意义不止于此。还有一些关于动植物PAC附近motif差异的发现,提出了动物中的保守的polyA signal(AAUAAA)在植物中可能不是最重要的信号,并且发现了植物PAC上游富集的UGUA 和 UAAA motif。
接下来让我们一起看看这个包到底如何使用。
名词缩写
- Poly(A) Cluster – PAC
- Poly(A) Site – PAS
- alternative polyadenylation – APA
二、安装
下载链接及相关依赖包可以参考 官方教程
网站针对R版本不同(以R 3.5.0为分界,因为bioconductor的用法不同)给出了两套依赖包安装代码。
实际安装过程中,如果是单独安装一个新的R环境,建议安装3.6以上版本(笔者用了3.6.3)。之前先尝试用3.5.0安装,发现困难重重,可能因为安装时如果不指定包的版本一般会下载并安装最新版,在这个R包的一长串依赖包中,存在着(依赖包的)依赖包依赖更高版本的R(例如DEseq2的一个依赖包 latticeExtra),这时直接按照教程安装就会装不上。3.6以上的R可以避免这类问题。
如果在conda环境中的R里安装,安装不上某些包时,也可以尝试直接用conda装(conda install r-包名),比如proj4就可以这样装:conda install -c conda-forge r-proj4。conda安装时也要注意包的版本,有时装包会有部分依赖被替代成更旧的版本,运行时出现不兼容的情况。
这个包安装的难点在于依赖包多,依赖包都安装完就很容易了。装完包跑一下测试数据。
三、使用
测试数据的运行–以拟南芥的数据为例
参考官方教程 第四节:
4.1 Application of QuantifyPoly(A) in an Arabidopsis dataset
数据准备
测试数据来源于Transcriptome Analyses of FY Mutants Reveal Its Role in mRNA Alternative Polyadenylation, 分析了植物中的3’ 加工因子FY(在物种间保守,与人的WDR33,酵母Pfs2p同源)在识别polyA信号(AAUAAA及一些相似序列)中起到的作用。除去fy-3,另一个用到的突变体是oxt6,CPSF30的突变体,也是在3’端加工中非常重要的一个蛋白,CPSF30和FY的关系也是文章的一个重点。因为CPSF30和FY在人里的同源蛋白已经被证明在polyA信号识别过程中起作用了,虽然植物中AAUAAA作为poly(A)信号不如动物中的保守(动物超过50%,植物10%左右但已经是占比最大的了),依然可以预期这两个突变体中的polyA位点使用情况与野生型会有比较大的差异,也会有一些全新的polyA位点,对polyA位点使用的分析显得格外重要。文章使用的是PAT-seq测序数据,实际上我们也可以使用其他富集polyA尾信息的建库方法比如PAS-seq或者DRS等,进行一些上游分析后得到polyA位点的位置和丰度信息并存储在bed文件中。
值得一提的是,测试数据的bed文件并不是标准的bed文件,由4列数据组成,依次是Chromosome,strand,start,read count。如果用自己的数据需要对标准的bed进行转换。
运行步骤
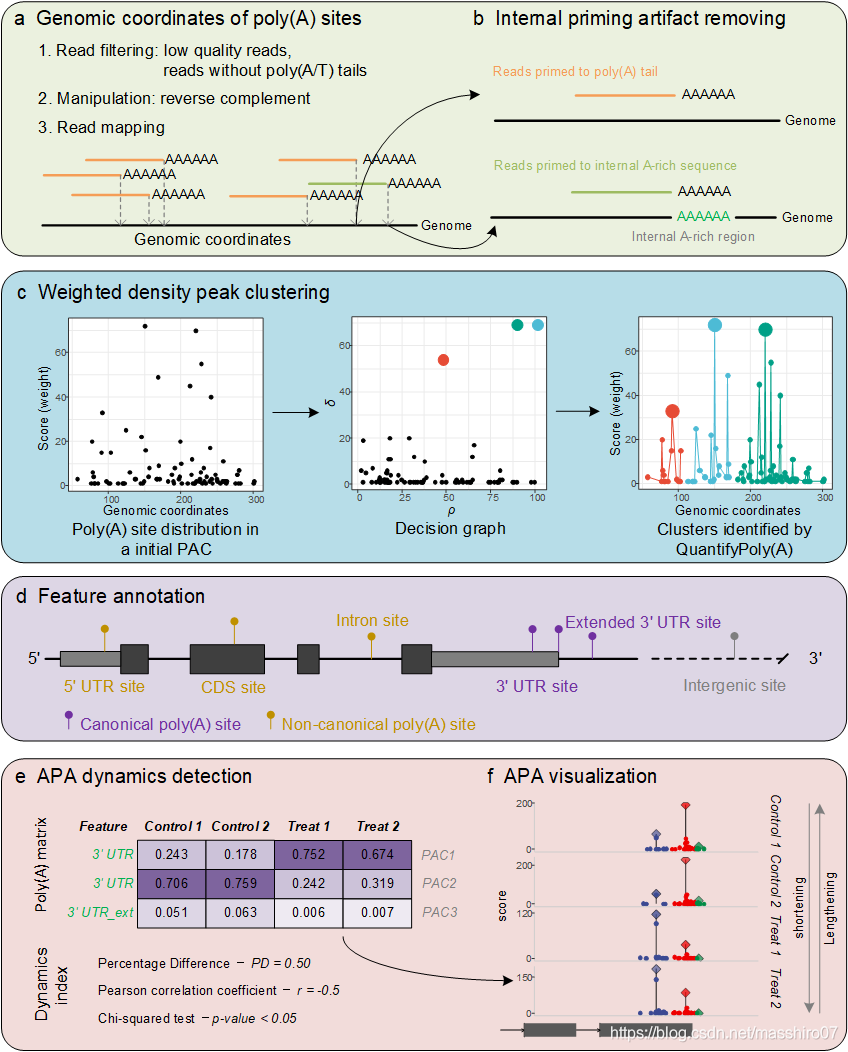
- 加载数据:把所有bed都放在一个文件夹内,用
dir指定文件夹位置读取所有文件,也可以直接指定要读取的文件名, 文件名的命名格式是“样本名_重复”,比如“wt_1” - 去除internal priming造成的假polyA site: 这个部分沿用李庆顺组一贯的标准,在polyA位点前后10bp共20bp的区间内,如果由连续6个A(AAAAAA)或者在10bp的区间内有不少于7个A,就认为是internel priming。对于基因内连续六个A可能引起internal priming我表示支持,但是对于非连续的A在热力学上是否有利于oligodT的结合表示存疑。Anyway,测试数据很干净没有这些,我自己的数据(3’mRNA-seq)大概会去掉10%,根据建库的方法不同可能会有差异(比如之前有文章说用anchored oligodT可以减少internal priming)。
- poly(A) site聚类:这个包最核心的功能之一,在普通的按距离(default: 24 nt)聚类的基础上增加了一步类似peak calling的步骤,把大的cluster又划分成小的sub-cluster,每个sub-cluster找到了一个高点(peak?)作为中心。(效果在可视化部分有体现,这部分我不确定理解的是否准确)
- 注释:注释基本是按照注释文件进行的,但对于3’UTR进行拓展,把下游两倍3’UTR长度以内的区域称为ext_3’UTR, 在3’UTR及ext_3’UTR的cluster被认为是经典的polyA位点,其他的地方就归类成非经典的polyA位点。编码基因的exon区域会细分成UTR和CDS,非编码的就是exon。(问题:对于没有UTR区域的non-coding gene,3’下游区域中的PAS是不是就不考虑了)
- 除去read数过少的PAC:除去在所有样本中read count都小于10的PAC,阈值可以更改
- APA动态的量化:首先需要分组,设置组名,之后可以比较组间差异。这个包提供了好几种比较方式,比较有用应该是Quantify.CNCAPA(比较经典PAC和非经典PAC的使用),此外还有Quantify.GeneAPA(比较基因间所有PAC使用的差异),Quantify.CanonicalAPA(比较经典PAC之间使用的差别)。文章定义了一个叫Percentage Difference(PD)的量来衡量PAC使用的差异, P D = ∑ i , j , k ∣ p i , k ? q j , k ∣ 2 × m × n \mathrm{PD}=\frac{\sum_{i,j,k}\left|{p}_{i,k}-{q}_{j,k}\right|}{2\times m\times n} PD=2×m×n∑i,j,k?∣pi,k??qj,k?∣?还做计算了pearson相关系数,做了chi-square test获得p值(让我有点诧异的是这个p值是个平均值)。PD值的边界很容易理解,0是完全没变,比如经典PAC与非经典PAC使用比例完全没变,1是变化极大,比如对照中只用经典PAC但实验组完全不用经典PAC只用一个novel的非经典PAC。但是如果生物学重复在某些基因上重复性不好,重复间变化趋势不一致,也会得到较大的PD值。但我并不太理解能不能用r或p去去除这些基因的干扰。从这步可以得到一些PAC使用有差异的基因,并在下一步骤中绘图。
- poly(A) profile的可视化:流程中f的图,包含基因结构和PAC的使用信息,可以直观的反映样本间的PAC使用差异。不同PAC颜色不同;对一个PAC,cluster的中心点就是棒棒糖图那个棒的位置,cluster里的read总数就是棒棒糖的高度,raw count显示成点。
- DESeq2差异分析:这个里面的差异分析意义不太大,但是可以画PCA或者UMAP图,看下生物学重复重复性好不好、还有样本间差异的大小。
几个小问题
- 教程提供链接对应的fas文件染色体名字(格式:X)和bed文件中的格式(格式:ChrX)不对应,自己更改下或者使用别的TAIR10的fasta文件都可以
- gtf文件下载链接基本没速度,不知道是不是我自己的问题,也可以用已有的TAIR10版本的文件代替
- 这套数据可能已经去除过internal priming,运行第二步并没有去除任何疑似internal priming artifacts,我用bedtools提取序列检查PAS附近的序列,确实没有AAAAAA
四、总结
QuantifyPolyA能帮助我们定义PAC并且比较样本间PAC使用的差异。但是里面有一些结果不太准确。