Labeled Fishes in the Wild数据集处理(适用于yolov5)
文章目录
一、前言
Labeled Fishes in the Wild数据集包括鱼类、无脊椎动物和海底图像。附件数据文件(.dat、.vec 和 .info)中包含注释数据,这些文件描述了图像中标记的鱼目标的位置。此数据集有三个组成部分:训练和验证正图像集(有鱼),负图像集(非鱼)和测试图像集。
数据集下载地址:https://swfscdata.nmfs.noaa.gov/labeled-fishes-in-the-wild/
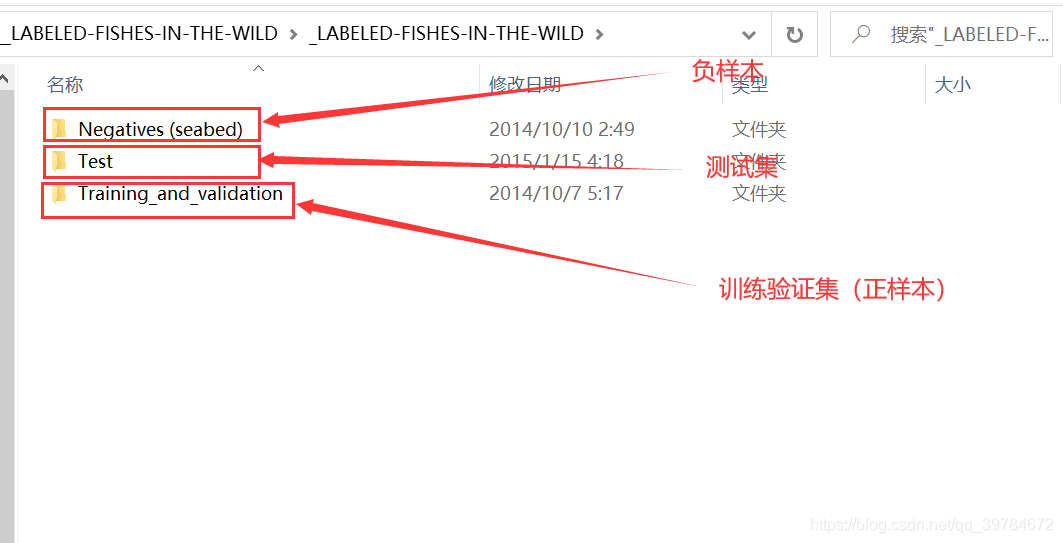
下载得到一个压缩包,解压后文件结构如下图所示:
├─Negatives (seabed)
├─Test
└─Training_and_validation
└─Positive_fish
└─vec_Positive_fish_(ALL)
这里因为我们是制作yolov5的数据集,所以需要获取包含鱼类的图片和对应的标注文件。关于yolov5数据集的组成,可以参见:YOLOV5训练自己的数据集(踩坑经验之谈),文章中博主介绍了yolov5数据集的制作和文件结构。可是文中对于制作数据集并未详述,于是就有了本文。下文中将详述如何制作适用于yolov5的野生鱼类数据集。
二、制作过程
1.训练图片筛选
下载的Labeled Fishes in the Wild数据集,最开始应该是适用于opencv通过Haar cascade训练分类器进行鱼类检测的,包含正负样本,其标注文件格式也不是VOC或者yolov5。因此,我们需要对图片进行筛选。首先,我们取出Positive_fish文件夹。其中包含一个含义vec标注文件的文件夹、两个标注文件(.dat和.info)、1326张图片(不知道为什么这1326张图片居然有很多张都是没有鱼的,这可能是其标注文件不足1000的原因吧)。
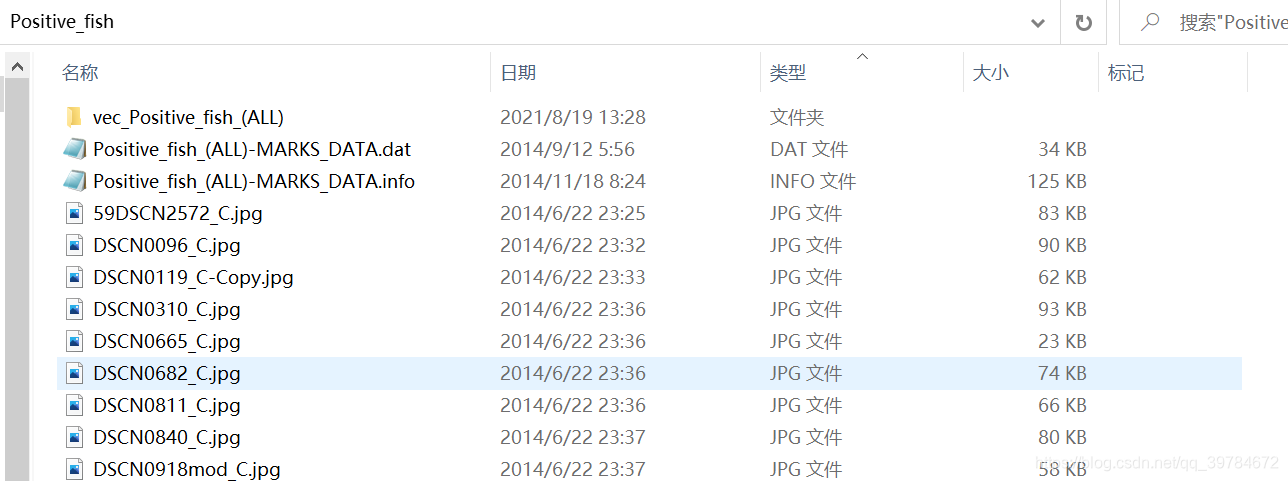
关于标注文件,vec貌似是opencv下使用的,所以我们去掉这个文件。至于.dat和.info,前者包含了图片的标注信息,后者包含了图片的尺寸信息和标注信息。两者选其一就好,我使用的是.dat文件。最终得到的文件夹目录如下:
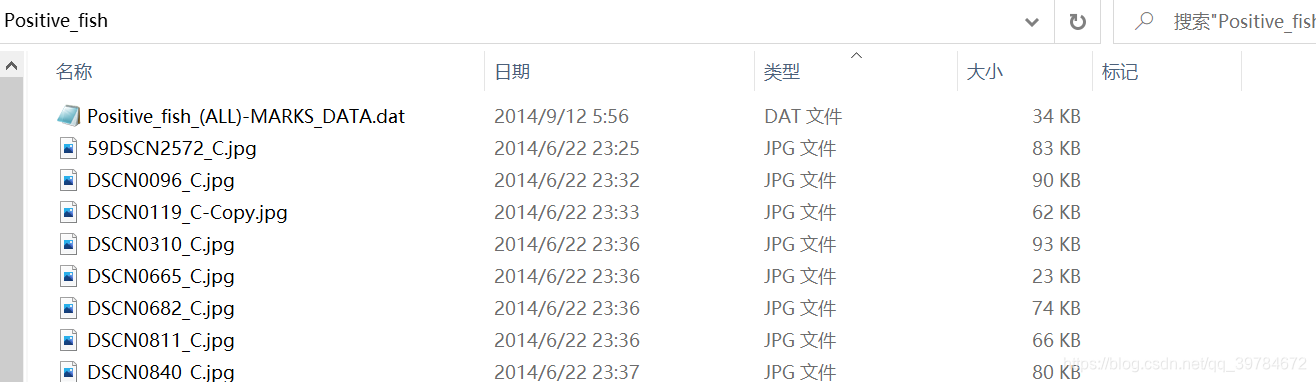
.dat文件信息如下:

接下来,通过遍历.dat,从1326张图片中筛出具有标注的图片。
在python脚本如下:
import os
from shutil import copy
# 待筛选图片路径
source_path = r'D:\OneDrive\桌面\Positive_fish'
# 标注图片保存路径
save_dir = r'D:\OneDrive\桌面\images'
with open("Positive_fish_(ALL)-MARKS_DATA.dat", "r") as f:
data = f.read()
data_list = data.split("\n")
count = 0
img_name_list = []
for d in data_list:
image_name = d.split(" ")[0]
img_name_list.append(image_name)
copy(source_path + "\\" + image_name, save_dir + "\\" + image_name)
print(str(count) + "\t" + image_name + " " + "finished!")
count += 1
运行脚本,得到包含具有标注信息的图片文件夹images。
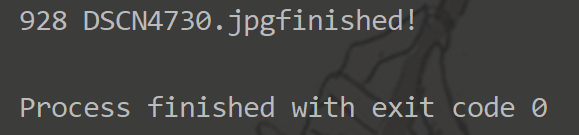
从打印结果看出,标注信息有928条,但是标注图片实际只有578张(有的图片中有多个目标)。
2.获取VOC标签
得到具有标注的图片后,下一步便是从从.dat文件中提取出标签。
提取出VOC标签文件,python脚本如下:
import os
import xml.etree.ElementTree as ET
from PIL import Image
# Positive_fish_(ALL)-MARKS_DATA.dat文件位置
source_path = r"./"
# 标签存放位置
save_path = r"D:\OneDrive\桌面\Annotations"
# 训练图片存放位置
image_path = r"D:\OneDrive\桌面\images"
class GenAnnotations:
def __init__(self, filename, witdh = None, height = None, depth = 3, foldername = "images", sourcename = "Unknown",
isExist = False):
if not isExist:
self.root = ET.Element("annotation")
self.foleder = ET.SubElement(self.root, "folder")
self.filename = ET.SubElement(self.root, "filename")
self.path = ET.SubElement(self.root, "path")
self.source = ET.SubElement(self.root, "source")
self.size = ET.SubElement(self.root, "size")
self.width = ET.SubElement(self.size, "width")
self.height = ET.SubElement(self.size, "height")
self.depth = ET.SubElement(self.size, "depth")
self.foleder.text = foldername
self.filename.text = filename
self.path.text = image_path + "\\" + filename
self.source.text = sourcename
self.width.text = str(witdh)
self.height.text = str(height)
self.depth.text = str(depth)
else:
self.tree = ET.parse(filename)
self.root = self.tree.getroot()
def savefile(self, filename):
tree = ET.ElementTree(self.root)
tree.write(filename, xml_declaration = False, encoding = 'utf-8')
def add_object(self, label, xmin, ymin, xmax, ymax, tpose = "Unspecified", ttruncated = 0, tdifficult = 0):
object = ET.SubElement(self.root, "object")
namen = ET.SubElement(object, "name")
namen.text = label
pose = ET.SubElement(object, "pose")
pose.text = str(tpose)
truncated = ET.SubElement(object, "truncated")
truncated.text = str(ttruncated)
difficult = ET.SubElement(object, "difficult")
difficult.text = str(tdifficult)
bndbox = ET.SubElement(object, "bndbox")
xminn = ET.SubElement(bndbox, "xmin")
xminn.text = str(xmin)
yminn = ET.SubElement(bndbox, "ymin")
yminn.text = str(ymin)
xmaxn = ET.SubElement(bndbox, "xmax")
xmaxn.text = str(xmax)
ymaxn = ET.SubElement(bndbox, "ymax")
ymaxn.text = str(ymax)
if __name__ == '__main__':
# 读取数据
with open(source_path + "\\" + "Positive_fish_(ALL)-MARKS_DATA.dat", "r") as f:
data = f.read()
data_list = data.split("\n")
# print(data_list)
# 循环创建标签
for item in data_list:
info = item.split(" ")
filename = info[0]
object_num = int(info[1])
im = Image.open(image_path + "\\" + filename) # 返回一个Image对象
im_width, im_height = im.size[0], im.size[1]
if not os.path.exists(save_path + "\\" + filename[:-4] + ".xml"):
anno = GenAnnotations(filename, witdh = im_width, height = im_height)
else:
anno = GenAnnotations(filename = save_path + "\\" + filename[:-4] + ".xml", isExist = True)
for i in range(object_num):
xmin = int(info[2 + i * 4 + 0])
ymin = int(info[2 + i * 4 + 1])
xmax = int(info[2 + i * 4 + 0]) + int(info[2 + i * 4 + 2])
ymax = int(info[2 + i * 4 + 1]) + int(info[2 + i * 4 + 3])
anno.add_object("fish", xmin, ymin, xmax, ymax)
anno.savefile(save_path + "\\" + filename[:-4] + ".xml")
Annotations文件夹下便会生成voc格式的标签文件。

建议把图片的后缀名统一成
.jpg,因为其中存在的后缀为.JPG的图片在训练过程中可能会读取失败。
统一后缀名,python脚本如下:
import os
path = r'D:\OneDrive\桌面\images'
list = os.listdir(path)
for l in list:
if l[-3:] == "JPG":
os.rename(path + "\\" + l, path + "\\" + l[:-3] + "jpg")
3. 完善标注信息
其实如果在LabelImg工具中打开得到的voc标注文件,就会发现官方标注其实并不是十分完善。有一些图片存在漏标的情况,所以建议最好是打开LabelImg一张一张过一遍,发现没有标注的鱼类目标时,进行完善一下。
4. yolov5训练处理
因为yolov5支持的数据集标注文件格式是.txt,所以需要进行转换。
具体处理详见博客:YOLOV5训练自己的数据集(踩坑经验之谈)。
最后,会得到一个数据集文件夹,如下图所示:

其中
test_videos文件夹下存放的是原本数据集中Test文件夹下的两个测试视频。
5. 处理数据集下载
处理后的yolov5鱼类数据集下载链接:https://download.csdn.net/download/qq_39784672/21361115