Swin-Unet: Unet-like Pure Transformer for Medical Image Segmentation 【用于医学图像分割的Unet形的纯Transformer】
Abstract
【前面阅读过不少混合结构,这篇纯Transformer结构是否参照swin-transformer?】
- 介绍大背景,Transformer结构为啥走进CV
- 在过去的很多年里,卷积神经网络CNN已经达到了医学图像分析中的里程碑 ,特别是基于U形结构和跳跃连接深度神经网络,这种结构已经被广泛的应用在各种医学图像任务中。【大背景CNN在医学影像中几乎存在统治地位】
- 然而,虽然CNN取得了惊人的性能,但是由于卷积算子的局部性,它不能很好的学习全局和长程语义信息的相互作用【这里就是本文大的出发点,CNN存在这样的问题,但是这个问题可以被transformer解决】
- 介绍本文将要提出的Transformer模型
- 在本文中,我们提出了Swin-Unet,它是一个用于医学图像分割的Unet形的纯Transformer。
- 标记化图像块(tokenized patches)被送入到带有跳跃连接的基于Transformer的U形编码解码结构,来实现局部-全局特征的学习。
- 特别地:我们使用带有滑动窗口的分层Swin Transformer 作为编码器来提取上下文特征,一个对称的带有块扩展层的基于Swin Transformer的解码器被设计用来执行上采样操作,通过上采样操作来恢复特征图的空间分辨率。
- 输入和输出的直接下采样和上采样为4x
- 在本文中,我们提出了Swin-Unet,它是一个用于医学图像分割的Unet形的纯Transformer。
- 介绍这个模型的性能
- 在多器官和心脏分割任务上的实现证明:
- 基于纯Transformer的U形编码解码网络优于那些全部使用卷积或者Transformer与CNN结合的方法。【大致意思就是我的纯Transformer干掉了混合结构和纯CNN】
- 代码开源,代码地址:https://github.com/HuCaoFighting/Swin-Unet
- 在多器官和心脏分割任务上的实现证明:
1 Introduction
- 介绍医学影像分割的发展
- 得益于深度学习的发展,计算机视觉技术已经被广泛应用于医学图像分析中。
- 图像分割是医学图像分析中的重要部分,特别地,准确和鲁棒地医学图像分割在计算机辅助诊断和影像引导临床手术的基石
- 介绍现行医学图像分割方法
- 现行的医学图像分割方法主要依赖于具有U形结构的全卷积神经网络(FCNN),典型的U形结构有 U-Net,由一个具有跳跃连接的堆成的编码解码结构组成。
- 在编码器,一系列的卷积层和持续的下采样层被用来提取大感受野的深度特征。
- 在解码器,解码器上采样提取到的特征至输入分辨率大小进行像素级语义预测。
- 来自编码器不同尺度的高分辨率特征被使用跳跃连接的方式进行融合,以减轻由于下采样造成的空间信息的损失,
- 正因为如此优美的结构设计,U-Net在多种医学图像应用中取得了巨大的成功,遵循这种 技术路线,很多算法被开发出来用于各种医学影像模态的图像和体素分割,比如3D U-Net, Res-UNet,U-Net++,UNet3+等
- 这些基于FCNN的方法在心脏分割、器官分割和病变分割中的优异性能证明了CNN具有很强的学习鉴别特征的能力。
- 现行的医学图像分割方法主要依赖于具有U形结构的全卷积神经网络(FCNN),典型的U形结构有 U-Net,由一个具有跳跃连接的堆成的编码解码结构组成。
- 介绍Transformer in Vision
- 最近,虽然基于CNN的方法在医学图像分割领域取得了极佳的性能,但是它们仍然不能完全满足医学应用当中对分割准确率的严格要求。
- 在医学图像分析中图像分割仍然是一个挑战性的任务
- 因为卷积算子固有的局部性,对于基于CNN的方法学习明确的全局和长程语义信息相互作用是困难的。 一些研究使用空洞卷积层,自注意力机制,图像金字塔等策略来解决这个问题。然而这些方法在建模长程依赖上仍然有局限性。
- 最近,受到Transformer在自然语言处理领域取得重大成功的启发,研究者开始尝试将Transformer带入视觉领域。
- VIT被提出来进行图像分类任务,使用具有位置嵌入的2D图像块作为输入,并在大规模数据集上进行预训练,相比基于CNN的方法,VIT取得了竞争性的性能。
- DeiT表明Transformer可以被训练在中等规模的数据集上,通过结合知识蒸馏可以获得一个更加鲁棒的Transformer。【知识蒸馏+Transformer】
- 分层的Swin-Transformer 作为视觉backbone,在图像分类、目标检测,语义分割上实现了最优性能
- ViT, DeiT , Swin Transformer在图像识别上取得的成功证明了Transformer被应用在视觉领域的潜力。
- 介绍本文开展的研究
- 受到Swin Transformer成功的启发 ,我们提出了Swin-Unet来利用Transformer的能力,在本次工作中进行2D医学图像分割。
- 据我们所知,Swin-Unet是第一个包含编码器,bottleneck 解码器和跳跃连接的基于纯Transformer的U形结构。Encoder, bottleneck 和 decoder都是基于Swin Transformer块建立。
- 输入的医学图像被分割为不重叠的图像块。每一个块被当作是一个token,将其送入到基于Transformer的编码器学习深度特征表示。
- 然后,被提取上下文特征通过具有块扩展层的解码器进行上采样,通过跳跃连接融合来自编码器的多尺度特征,以便恢复特征图的空间分辨率,进一步进行分割预测。
- 介绍本文开展的实验情况以及主要贡献
- 在多器官和心脏分割数据集上的广泛实验证明,提出的方法具有极高的分割准确率和鲁棒泛化能力
- 具体的,我们的贡献总结如下:
- 1 基于Swin Transformer块,我们建立了具有跳跃连接的对称的编码解码结构,在编码器,实现从局部到全局的自注意力。在解码器,全局特征被上采样至输入分辨率执行相应的像素级分割预测。
- 2 块扩展层被开发来实现上采样,不使用卷积和插值操作实现特征维度增加。【这个可以用一下】
- 3 实验中表明跳跃连接对于Transformer仍然有效,因此一个基于纯transformer的具有跳跃连接的U形编码解码结构最终实现,叫Swin-Unet
2 Realted work
2.1 CNN-based methods
- 主要介绍基于CNN的图像分割方法
- 早期的医学图像分割方法主要是基于轮廓的和基于传统及其学习算法,随着深度CNN的发展,U-Net被提出进行医学图像分割。
- 由于U形结构的简单和高性能,各种Unet形的方法不断出现。比如:Res-Unet,Dense-Unet,U-Net++ ,UNet3+.
- 同样还有3D医学图像分割领域,比如3D-Unet和V-Net。
- 目前,基于CNN的方法以其强大的表示能力在医学图像分割领域取得了巨大的成功。
- 早期的医学图像分割方法主要是基于轮廓的和基于传统及其学习算法,随着深度CNN的发展,U-Net被提出进行医学图像分割。
2.2 Vision transformers
- 介绍Transformer in Vision发展
- Transformer首先被提出来用于机器翻译任务,在NLP领域 ,基于Transformer的方法在各种任务中都取得了最优的性能。受到Transformer成功的驱动,研究者引入了首创的VIT,该模型在图像识别任务中取得了速度和精度的平衡的令人鼓舞的结果。
- 相比基于CNN的方法,ViT的缺点是需要在自己的大数据集上进行预训练。为了减轻ViT训练困难,DeiT提出了几种训练策略,这种策略可以允许ViT在ImageNet上训练的好。
- 最近,多种极好的工作都是基于ViT,值得注意的是,一个高效的有效的对称视觉Transformer,叫Swin Transformer,被提出作为视觉的backbone。基于滑动窗口机制,Swin Transformer在多项任务上实现了最优的性能,比如图像分类,目标i检测,语义分割。
- 本文,我们尝试使用SwinTransformer模块作为基础单元,来建立一个带有跳跃连接的U形编码解码结构。利用该结构进行医学图像分割。从而为医学图像领域中Transformer的发展提供一个基准比较
2.3 Self-attention/Transformer to complement CNNS
- 介绍CNN与Transformer的混合发展
- 近年来,研究者开始尝试将自注意力机制引入CNN来提高网络的性能
- 附加注意力们的跳跃连接 被集成到U形结构i中来进行医学图像分割,然而,这依然是基于CNN的方法
- 最近,为了打破CNN在医学图像分割中的主导地位,正在努力将CNN和Transformer结合起来。
- 结合Transformer与CNN构成一个强大的编码器,进行2D医学图像分割【】
- 利用CNN与Transformer来提高模型的分割能力【Transfuse,Transbts】
- 目前,Transformer与CNN的各种组合被应用于多模态脑肿瘤分割和3D医学图像分割。【Transbts和cotr】
- 近年来,研究者开始尝试将自注意力机制引入CNN来提高网络的性能
- 与以上方法不同的是,我们尝试去探索纯Transformer在医学图像分割中的应用潜力。
3 Method
3.1 Architecture overview
- 介绍整体结构
- 提出的Swin-Unet的整体结构如图1所示。Swin-Unet由编码器,bottleneck,解码器和跳跃连接组成。Swin-Unet的基础单元是Swin-Transformer块。
- 对于编码器,为了将输入转化为序列嵌入,医学图像被切分为不重叠的图像块,其中图像块的大小为4x4。通过这种划分方式,每一个块的特征维度变为4x4x3=48。更进一步的,线性嵌入层被用来将特征维度投影为任意维度(表示为C)。
- 转换后的块tokens通过多个Swin Transformer 块和块合并层来生成分层特征表示。特别地,块合并层负责下采样和增加维度,Swin Transformer块负责特征表示的学习,受到U-Net的启发,我们设计了一个基于Transformer的对称解码器。
- 解码器由Swin Transformer块和块扩展层组成,提取的上下文特征通过跳过连接与来自编码器的多尺度功能融合,以补充由下采样引起的空间信息的丢失。与块合并层形成对比,
- 块扩展层是一个特地设置来执行上采样的,块扩展层将相邻尺寸的特征映射到具有2x分辨率提升的大特征图。最终,最后的块扩展层用来执行4X上采样恢复特征图的分辨率到输入图分辨率(WxH).
- 然后应用线性投影层来对这些上采样的特征输出像素级预测,我们在下面将详细阐述每一个块。
- 提出的Swin-Unet的整体结构如图1所示。Swin-Unet由编码器,bottleneck,解码器和跳跃连接组成。Swin-Unet的基础单元是Swin-Transformer块。

3.2 Swin Transformer block
- 阐述一下Swin Transformer 块
-
与卷积多头注意力模块不同的是,Swin Transformer 模块是基于滑动窗口构成的,如图2所示,展示了两个连续的Swin Transformer块。

- 每一个swin Transformer 是由层归一化(LN),多头自注意力模块,残差连接,带有GELU非线性的两层MLP。
- 基于窗口的多头自注意力模块(W-MSA)和基于滑动窗口的多头自注意力模块被分别用在两个连续transformer模块。
- 基于这写窗口分区机制,连续的swin transformer块可以公式化为:
z ^ l = W ? M S A ( L N ( z l ? 1 ) ) + z l ? 1 z l = M L P ( L N ( z ^ l ) ) + z ^ l , z ^ l + 1 = S W ? M S A ( L N ( z l ) ) + z l z l + 1 = M L P ( L N ( z ^ l + 1 ) ) + z ^ l + 1 \begin{gathered} \hat{z}^{l}=W-M S A\left(L N\left(z^{l-1}\right)\right)+z^{l-1} \\ z^{l}=M L P\left(L N\left(\hat{z}^{l}\right)\right)+\hat{z}^{l}, \\ \hat{z}^{l+1}=S W-M S A\left(L N\left(z^{l}\right)\right)+z^{l} \\ z^{l+1}=M L P\left(L N\left(\hat{z}^{l+1}\right)\right)+\hat{z}^{l+1} \end{gathered} z^l=W?MSA(LN(zl?1))+zl?1zl=MLP(LN(z^l))+z^l,z^l+1=SW?MSA(LN(zl))+zlzl+1=MLP(LN(z^l+1))+z^l+1?
其中 z ^ l \hat{z}^{l} z^l 和 z l z^{l} zl分别代表(S)W-MSA模块 和 MLP模块的第i个块的输出,与前人的工作类似,自注意力可以计算如下:
Attention ? ( Q , K , V ) = Soft ? Max ? ( Q K T d + B ) V \operatorname{Attention}(Q, K, V)=\operatorname{Soft} \operatorname{Max}\left(\frac{Q K^{T}}{\sqrt{d}}+B\right) V Attention(Q,K,V)=SoftMax(d?QKT?+B)V
其中 Q , K , V ∈ R M 2 × d Q, K, V \in \mathbb{R}^{M^{2} \times d} Q,K,V∈RM2×d 代表 query, key and value 矩阵。 M 2 M^{2} M2 和 d d d 分别代表在窗口中块的数量以及query和key的维度, B B B中的值来自偏置矩阵 B ^ ∈ \hat{B} \in B^∈ R ( 2 M ? 1 ) × ( 2 M + 1 ) \mathbb{R}^{(2 M-1) \times(2 M+1)} R(2M?1)×(2M+1).
-
3.3 Encoder
- 介绍编码器
- 在编码器中,具有分辨率为 H 4 × W 4 \frac{H}{4} \times \frac{W}{4} 4H?×4W?的C维标记化输入被送入两个连续的Swin Transformer 块执行表示学习,特征维度和分辨率保持不变。
- 同时块合并层减少标记的数量(2倍下采样),增加特征维度为原始维度的两倍,此过程在编码器中重复三次。
- 块合并层
- 输入块被分为四个部分,通过块合并曾进行连接,通过这种操作,特征分辨率将会2倍下采样,因为连接操作导致特征维度增加四倍,在连接的特征上应用线性层,以将特征尺寸统一为原始尺寸的2。
3.4 Bottleneck
- 介绍bottleneck
- 因为Transformer太深难以优化,仅仅两个连续的Swin Transformer块被用来构成bottleneck来学习深度特征表示
- 在bottleneck中,特征的维度和分辨率保持不变。
3.5 Decoder
- 介绍解码层和特征上采样
- 与编码器一致,堆成的解码器也是基于Swin Transformer块构成。
- 最终,与在编码器中使用的块合并层相反的是,我们在解码器中使用块扩展层对提取的深度特征进行上采样。
- 块扩展层将相邻维度的特征图重塑为更高分辨率的特征图(2倍上采样),并相应的将特征深度减少至原深度的一半
- 介绍上采样-块扩展层
- 以第一个块扩展层为例
- 在上采样之前,一个线性层被用在输入特征上 ( W 32 × H 32 × 8 C ) \left(\frac{W}{32} \times \frac{H}{32} \times 8 C\right) (32W?×32H?×8C),来增加特征维度到原来维度的2倍 ( W 32 × H 32 × 16 C ) \left(\frac{W}{32} \times \frac{H}{32} \times 16 C\right) (32W?×32H?×16C).
- 然后我们使用重排操作扩展输入特征的分辨率为输入分辨率的两倍,同时减少特征至输入维度的四分之一 ( W 32 × H 32 × 16 C → W 16 × H 16 × 4 C ) \left(\frac{W}{32} \times \frac{H}{32} \times 16 C \rightarrow \frac{W}{16} \times \frac{H}{16} \times 4 C\right) (32W?×32H?×16C→16W?×16H?×4C).
- 我们将探讨使用块扩展层执行上采样的影响
- 以第一个块扩展层为例
3.6 Skip connection
- 介绍跳跃连接的作用
- 与U-Net相似,跳跃连接被用来融合来自编码器的多尺度特征和上采样特征,我们连接了浅部特征和深度特征,以此减少下采样造成的空间信息的损失。
- 然后是线性层,串联特征的与输入特征的维度保持一致。
- 我们将详细讨论跳跃连接数量对我们模型性能的影响。
4 Experiments
4.1 Datasets
- 介绍器官分割和心脏分割数据集
- Synapse multi-organ segmentation dataset (Synapse): 腹部器官分割数据集
- 数据集包括30个病例。共3779张轴向腹部临床CT图像,遵循前人的操作,18个被分为训练集,12个病例被分为测试集
- 平均Dice相似系数(DSC)和平均Hausdorff距离(HD)被作为评价指标在八种腹部器官上评估我们的方法【(aorta, gallbladder, spleen, left kidney, right kidney, liver, pancreas, spleen,stomach) (主动脉、胆囊、脾脏、左肾、右肾、肝、胰腺、脾脏、胃) 】
- Automated cardiac diagnosis challenge dataset (ACDC) 心脏数据集
- 使用MRI扫描仪从不同患者收集到的ACDC数据集,对于每一个病人的MRI图像,左心室,右心室,心肌层被标注。
- 数据集被分为70个训练样本,10个验证样本,20个测试样本,与前人一致,在该数据集上仅仅Dice相似系数(DSC)被用来评估我们的方法。
- Synapse multi-organ segmentation dataset (Synapse): 腹部器官分割数据集
4.2 Implementation details
- 介绍具体实验怎么做的
- Swin-Unet是使用Python3.6和Pytorch1.7实现的,对于所有的训练实例,例如翻转,旋转的数据增强方法被用来增加数据多样性。
- 输入图像的大小为224x224,块大小为4。
- 我们在具有显存32G的v100上训练我们的模型,权重是在ImageNet预训练的,被用来初始化模型参数
- 在训练周期中,batchsize是24,流行的带有动量为0.9的SGD优化器,权重衰减为1e-4被用来优化反向传播模型
4.3 Experiment results on Synapse dataset
- 介绍实验结果
-
如表1所示是提出的Swin-Unet和最新的最优方法在多器官CT数据集上的实验效果。不同于TransUnet,我们添加了我们自己实现的U-Net和Att-Unet。

-
实现结果证明我们的Unet形纯Transformer取得了最优的性能,分割准确率为79.13%(DSC)和HD为21.55%。与Att-Unet和最新的TransUnet相比,虽然我们的方法没由在DSC评价指标上提升很多,我们在HD评价指标上提升了大概4%和10%。这表明我们的方法可以实现更好的边界预测。
-
在多器官CT数据集上不同方法的分割结果如图3所示,从图3中可以看到基于CNN的方法有过分割的问题,这或许是由于卷积算子的局部性导致的。

-
在本次工作中,我们证明了集成Transformer和带有跳跃连接的U形结构,不带卷积的纯Transformer可以更好的学习全局和长程语义信息的相互作用,使得分割结果更好。
-
4.4 Experiment results on ACDC dataset
- 在心脏数据集上的分割结果:
- 与器官分割数据集类似,提出的Swin-Unet在ACDC数据集上训练来执行医学图像分割,实验的结果如表2所示,使用MR形式的图像数据作为输入,Swin-Unet仍然能取得极高的性能,准确率为90.00%,这表明了我们的方法有很好的泛化性和鲁棒性。
4.5 Ablation study
- 消融实验
- 为了去探究不同因素在模型性能上的影响,我们在器官分割数据集上进行消融实验的研究,特别地,上采样,跳跃连接数量,输入大小,模型尺寸的探讨如下:
- Effect of up-sampling: 介绍上采样的影响
- 为了与编码器中的块合并层一致,我们特地在解码器中涉及了块扩展层来执行上采样和特征维度增加。
- 为了探索提出的块扩展层的有效性,我们在多器官分割上使用双线性插值,转置卷积,块扩展层进行了实验,实验结果如表3

- 最终证明我们的Swin-Unet结合块扩展层可以获得更高的分割准确率。
- Effect of the number of skip connections: 跳跃连接数量的影响
- 我们的Swin-Unet的跳跃连接被添加在了1/4,1/8.1/16分辨率尺寸的位置,通过改变跳跃连接的数量分别为0,1,2,3,我们探索了所提方法的不同跳跃连接在分割性能上的影响
- 结果如表4,我们可以看到提出模型的分割性能随着跳跃连接数量的增加而增加,因此为了使得模型更加鲁棒,跳跃连接的数量在本文中设置为3。

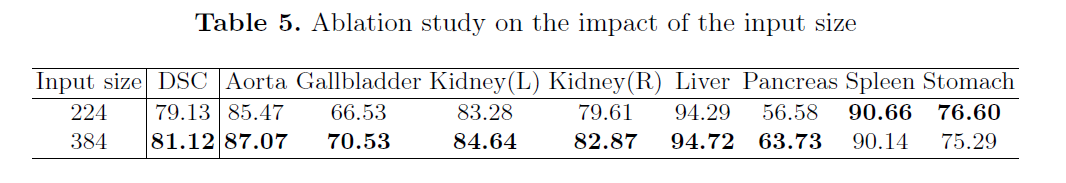
- Effect of input size: 输入大小的影响
- 如表5所示,是提出的Swin-Unet的输入分辨率为224x224,384x384的测试结果。当输入大小从224x224增加到384x384 ,块大小保持一致为4时候,输入Transformer的token序列的会变大,这会提升模型的分割性能。然而,虽然模型的分割准确率有轻微的提升,但是整个网络的计算量也显著增加。
- 为了确保算法的运行性能,本文中实验是基于224*224分辨率的尺度作为输入。
- Effect of model scale: 模型尺度的影响
- 与前人工作类似,我们探讨了网络深度对模型性能的影响,如表6所示,增加模型的尺度难以提升模型的性能。但是会增加整个网络的计算消耗。考虑到准确和速度的平衡,我们采用小模型执行医学图像分割。

- 与前人工作类似,我们探讨了网络深度对模型性能的影响,如表6所示,增加模型的尺度难以提升模型的性能。但是会增加整个网络的计算消耗。考虑到准确和速度的平衡,我们采用小模型执行医学图像分割。
4.6 Discussion
- 探讨一些为解决或者存在特殊意义的东西
- 正如我们所知道的是,基于Transformer模型的性能受到预训练模型的影响较大,在本文中,我们直接使用Swin Transformer在ImageNet上的训练权重来初始化网络的编码器和解码器,这或许是一个次优的方案。
- 这种初始化方法过于单一(简单),后面我们将探索预训练的端到端的Transformer进行医学图像分割,因为本文中的输入图像为2D,然而大部分的医学图像数据是3D的,我们将在下一步探索Swin-Unet用于3D医学图像分割。
5 Conclusion
- 总结本文提出了什么,解决了什么
- 本问,我们引入了一个基于纯Transformer的U形编码解码器执行医学图像分割
- 为了利用Transformer的能力,我们使用了Swin-Transfomer 块作为基础单元,学习特征表示和长程语义信息的相互关系。
- 在多器官和心脏上的分割任务的实验证明了提出的Swin-Unet具有极佳的性能和泛化能力。