前言
在做单细胞相关聚类分析的过程中,为了研究异质性,发现了一种专门用于单细胞转录组的聚类分析R包——SC3(single cell consensus clustering,单细胞一致性聚类)。该聚类算法发表于Nature Methods(2017),能够让单细胞RNA-seq基于转录组特征对细胞类型进行定量表征。这是一种用户友好的无监督聚类工具,它通过一致方法将多个聚类解决方案组合在一起,从而得到高精度和鲁棒性的分群结果。SC3包的官方具体使用教程见:http://www.bioconductor.org/packages/release/bioc/vignettes/SC3/inst/doc/SC3.html
SC3包源代码见Github:https://github.com/hemberg-lab/SC3
原文的SC3聚类流程如图所示:
(a)使用SC3框架进行聚类的概述(参见方法)。用Treutlein数据举例说明了一致步骤。
(b)用于设置SC3参数的已发布数据集。N是数据集中的细胞数;k为作者最初确定的簇数;单位:RPKM是每千碱基每百万次读的转录本,RPM是每百万次读的转录本,FPKM是每千碱基每百万次读的转录本片段,TPM是每百万次读的转录本片段。
( c)ARI>处d值的直方图。金标准数据集达到95。黑色竖线表示细胞总数N的d = 4-7%,分类准确率高。
(d) (b)中所示数据集的SC3聚类的100个实现。条对应点的中位数。红色和灰色分别对应有一致步长和无一致步长时的聚类。这条黑线对应的ARI=0。8。黑色虚线分隔了金和银标准数据集。
笔者主要想通过该包对单细胞转录组进行聚类数目的估计。在网上没有直接写SC3关于这个功能的帖子,故我去原文,官方教程以及代码研究了一下,该包主要通过随机矩阵理论(Random Matrix Theory,RMT) 确定聚类的数目。
安装SC3包
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("SC3")
同时还需要安装如下包:
library(SingleCellExperiment)#安装方法和SC3一样
library(SC3)
library(ggplot2)
library(scater)
利用SC3实现聚类数目的估计
首先看一下源代码:
if (k_estimator) {
object <- sc3_estimate_k(object)
# Do not override cluster if user has set a k
if (is.null(ks))
{
ks <- metadata(object)$sc3$k_estimation
}
把该方法应用于自己的代码:
注意:在跑自己的数据之前,要先准备两个文件:(1)单细胞表达矩阵文件;(2)每个细胞的注释文件。
pbmc <- read.table("SC3/分组表达/exp_SCE_M.csv",stringsAsFactors=FALSE, header=TRUE, check.names=FALSE,row.names=1, sep=",")
ann<-read.table("SC3/ann/SCE_M.csv", header=T, row.names=1, sep=",")
#构建sce对象
sce <- SingleCellExperiment(
assays = list(
counts = as.matrix(pbmc),
logcounts = log2(as.matrix(pbmc) + 1)
),
colData=ann
)
# define feature names in feature_symbol column
rowData(sce)$feature_symbol <- rownames(sce)
# remove features with duplicated names
sce <- sce[!duplicated(rowData(sce)$feature_symbol), ]
# define spike-ins
#isSpike(sce, "ERCC") <- grepl("ERCC", rowData(sce)$feature_symbol)
#开始聚类(estimate k)
sce<-sc3_estimate_k(sce) #估计k值
ks<-metadata(sce)$sc3$k_estimation #调用k值
ks
a <- ks - 1
b <- ks + 1
sce <- sc3(sce, ks = a:b, biology = TRUE) #这一步会运行很久
#主成分分析PCA
sce <- runPCA(sce)
plotPCA(sce, colour_by = "cell_type")
#画降维后的分布
sc3_n_clusters <- paste0("sc3_", as.character(ks),"_clusters")
sc3_n_log2_outlier_score <- paste0("sc3_", as.character(ks), "_log2_outlier_score")
sc3_n_clusters
sc3_n_log2_outlier_score
plotPCA(
sce,
colour_by = sc3_n_clusters,
size_by = sc3_n_log2_outlier_score
)
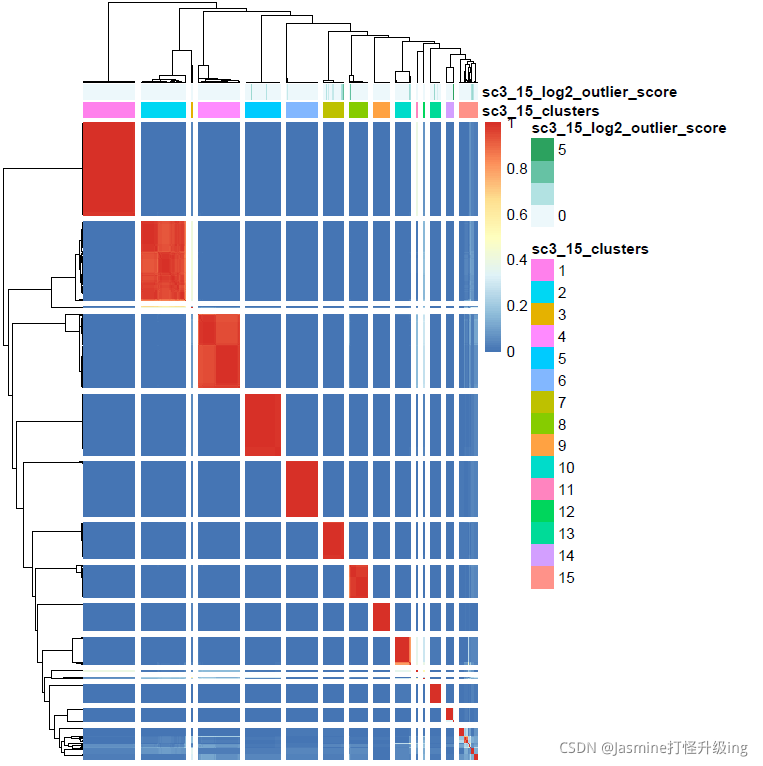
#计算一致性矩阵
sc3_plot_consensus(
sce, k = ks,
show_pdata = c(
#"cell_type",
#"log10_total_features",
sc3_n_clusters,
sc3_n_log2_outlier_score
)
)
#计算簇内稳定性
sc3_plot_cluster_stability(sce, k = ks)
绘制的PCA结果和一致性矩阵结果如下图:

SC3包后续还可以进行基因表达矩阵热图的绘制; 提供调整p值< 0.01的所有差异表达基因的列表,并绘制p值最低的50个基因的基因表达谱;以及找到标记基因。
参考:
代码:https://blog.csdn.net/weixin_42889479/article/details/108943720
https://www.jianshu.com/p/65094dfd6922
https://www.jianshu.com/p/24f65dd2c88f
https://www.jianshu.com/p/ea81c3635a8b