基于人工智能的多肽药物分析问题(三)
2021SC@SDUSC
1. Importing larger libraries
os.environ['TF_CPP_MIN_LOG_LEVEL'] = '3'
os.environ["CUDA_VISIBLE_DEVICES"] = str(args.gpu)
script_dir = os.path.dirname(__file__)
sys.path.insert(0, script_dir)
import pyErrorPred
num_process = 1
if args.process > 1:
num_process = args.process
其中os.environ[]表示的是一个字符串所对应环境的映像对象,使用os.environ.keys()可以查看当前运行环境的系统的各种信息,代码中首先将'TF_CPP_MIN_LOG_LEVEL'设置为3等级,即将log日志信息设置为忽略 “通知”,“警告”和“报错”级别的日志输出。易知log信息分为四个级别,按重要性递增为:INFO(通知)<WARNING(警告)<ERROR(错误)<FATAL(致命的);所以,该系统最多只会输出FATAL级别的日志。代码的作用基本上是忽略一些不必要的输出信息,便于得到清晰的结果。
"CUDA_VISIBLE_DEVICES"命令是选择将要使用的gpu,其中gpu编号信息从命令行参数获取。
之后,代码将script_dir的路径添加至sys.path路径。并将num_process默认设置为1。
2. Getting samples names
该部分主要是对输出文件的命名设置
if not args.pdb:
if args.prefix == None:
samples = [i[:-4] for i in os.listdir(args.infolder) if isfile(args.infolder+"/"+i) and i[-4:] == ".pdb" and i[0]!="."]
ignored = [i[:-4] for i in os.listdir(args.infolder) if not(isfile(args.infolder+"/"+i) and i[-4:] == ".pdb" and i[0]!=".")]
else:
samples = [i[:-4] for i in os.listdir(args.infolder) if isfile(args.infolder+"/"+i) and i[-4:] == ".pdb" and i[0]!="." and args.prefix in i]
ignored = [i[:-4] for i in os.listdir(args.infolder) if not(isfile(args.infolder+"/"+i) and i[-4:] == ".pdb" and i[0]!=".")]
if args.verbose:
print("# samples:", len(samples))
if len(ignored) > 0:
print("# files ignored:", len(ignored))
如本系列第二篇文章说到的 命令函参数的pdb命令表示的是运行在单个的pdb文件上,默认为FALSES,所以,在此处对文件做了命名的处理。
3. Featurization happens here
inputs = [join(args.infolder, s)+".pdb" for s in samples]
tmpoutputs = [join(args.outfolder, s)+".features.npz" for s in samples]
if not args.reprocess:
arguments = [(inputs[i], tmpoutputs[i], args.verbose) for i in range(len(inputs)) if not isfile(tmpoutputs[i])]
already_processed = [(inputs[i], tmpoutputs[i], args.verbose) for i in range(len(inputs)) if isfile(tmpoutputs[i])]
if args.verbose:
print("Featurizing", len(arguments), "samples.", len(already_processed), "are already processed.")
else:
arguments = [(inputs[i], tmpoutputs[i], args.verbose) for i in range(len(inputs))]
already_processed = [(inputs[i], tmpoutputs[i], args.verbose) for i in range(len(inputs)) if isfile(tmpoutputs[i])]
if args.verbose:
print("Featurizing", len(arguments), "samples.", len(already_processed), "are re-processed.")
if num_process == 1:
for a in arguments:
pyErrorPred.process(a)
else:
pool = multiprocessing.Pool(num_process)
out = pool.map(pyErrorPred.process, arguments)
print(modelpath)
# Exit if only featurization is needed
if args.featurize:
return 0
遍历sample列表,分别设置输入文件列表的名字及后缀,以及输出文件列表的名字和相应的后缀
当命令行参数中未定义reprocess参数,即未定义重新处理特征文件的话,对arguments 和already_processed两个变量进行遍历复制操作;如果参数定义需要输出详细参数,则通过print输出。
如果num_process的值为1,那么对于arguments列表的每一个元素,调用了process方法
4. process() 方法定义
def process(args):
filename, outfile, verbose = args
try:
start_time = time.time()
pose = Pose()
pose_from_file(pose, filename)
fa_scorefxn = get_fa_scorefxn()
score = fa_scorefxn(pose)
pdict = init_pose(pose)
maps = extract_multi_distance_map(pose)
_2df, aas = extract_EnergyDistM(pose, energy_terms)
_1df, _ = extractOneBodyTerms(pose)
prop = extract_AAs_properties_ver1(aas)
np.savez_compressed(outfile,
idx = pdict['idx'],
val = pdict['val'],
phi = pdict['phi'],
psi = pdict['psi'],
omega6d = pdict['omega6d'],
theta6d = pdict['theta6d'],
phi6d = pdict['phi6d'],
tbt = _2df,
obt = _1df,
prop = prop,
maps = maps)
if verbose: print("Processed "+filename+" (%0.2f seconds)" % (time.time() - start_time))
except Exception as inst:
print("While processing", outfile+":", inst)
该方法中,首先初始化一个Pose()对象,然后使用pose_from_file方法,从命令行提供的pdb文件名,提取出pdb文件中蛋白质的结构。
其中,Pose 类包括描述结构的各种类型的信息。一些核心组件包括 Energies、PDBInfo 和 Conformation
蛋白质数据库 (PDB) 是一种文本文件格式,用于描述 3D 分子结构和其他信息
使用get_fa_scorefxn传入pose对象,返回一个score评分
4.1 init——pose() 初始化pose
def init_pose(pose):
pdict = {}
pdict['pose'] = pose
pdict['nres'] = pyrosetta.rosetta.core.pose.nres_protein(pdict['pose'])
pdict['N'], pdict['Ca'], pdict['C'], pdict['Cb'] = get_coords(pdict['pose'])
set_lframe(pdict)
set_neighbors6D(pdict)
set_neighbors3D(pdict)
set_features1D(pdict)
return pdict
声明pdict为一个字典,pdict['pose'] = pose将传入的pose对象作为pose键的值,pdict['nres']的值调用了pyrosetta的方法nres_protein,返回pose对象记录的蛋白质的残基的数量,其中,虚残基、膜残基或包埋残基不被记录。
之后调用get_coords()方法:
4.1.1 get_coords()
def get_coords(p):
nres = pyrosetta.rosetta.core.pose.nres_protein(p)
# three anchor atoms to build local reference frame
N = np.stack([np.array(p.residue(i).atom('N').xyz()) for i in range(1,nres+1)])
Ca = np.stack([np.array(p.residue(i).atom('CA').xyz()) for i in range(1,nres+1)])
C = np.stack([np.array(p.residue(i).atom('C').xyz()) for i in range(1,nres+1)])
# recreate Cb given N,Ca,C
ca = -0.58273431
cb = 0.56802827
cc = -0.54067466
b = Ca - N
c = C - Ca
a = np.cross(b, c)
Cb = ca * a + cb * b + cc * c
return N, Ca, C, Ca+Cb
get_coords传入的参数为先前声明的pose实例对象,残基数保存在nres变量中,使用三个锚原子来建立局部参考系,其中:
N变量:对于p变量中每一个残基,p.residue(i)方法查看p这个pose实例的第i个残基,返回值是三个大写字母组成的字符串,如ARG,再通过.xyz()方法以xyzVector形式返回原子坐标,并保存至变量N,
np.stack()方法
numpy.stack(arrays, axis=0)
# 用于沿着指定的轴连接数组的序列,axis参数指定新轴在结果尺寸中的索引,axis参数默认为0,如果为0那么他将会是第一个维度。
即返回的数组比输入的数组多一个维度。
Ca,C变量同理,保存了一个和N变量同维度的数组
之后声明三个局部变量ca,cb,cc,令
b = Ca - N
c = C - Ca
a = np.cross(b, c) # 计算b,c向量的叉乘结果
Cb = ca * a + cb * b + cc * c
最后将四个变量值返回给pdict变量的四个键。
4.1.2 set_lframe()
def set_lframe(pdict):
# local frame
z = pdict['Cb'] - pdict['Ca']
z /= np.linalg.norm(z, axis=-1)[:,None]
x = np.cross(pdict['Ca']-pdict['N'], z)
x /= np.linalg.norm(x, axis=-1)[:,None]
y = np.cross(z, x)
y /= np.linalg.norm(y, axis=-1)[:,None]
xyz = np.stack([x,y,z])
pdict['lfr'] = np.transpose(xyz, [1,0,2])
将pdict字典变量传递给set_lframe方法
主要是用于设置pdict的lfr的值:
其中:np.linalg.norm用于求矩阵的范数
该方法有4个参数x, ord=None, axis=None, keepdims=False
-
x传入要求解的矩阵变量
-
ord表示求解的范数类型
-
axis表示处理的类型,为1时表示按行向量处理,求多个行向量的范数,为0时表示按列向量处理,求多个列向量的范数,默认情况则表示矩阵范数。
-
keepdims表示是否保持矩阵的二维特性
矩阵的范数:
ord=1:列和的最大值
ord=2:|λE-ATA|=0,求特征值,然后求最大特征值得算术平方根
ord=∞:行和的最大值
向量的范数:
np.transpose函数的作用就是调换数组的行列值的索引值,类似于求矩阵的转置:
其中,用从0开始的整数表示数组的维度:0,1,2,3
而transpose的axes参数即第二个参数表示的时调换的规则
np.transpose(xyz, [1,0,2]) 将xyz三个维度的坐标进行了转化,根据1,0,2的参数可知将x和y两个维度的坐标进行了互换,并返回
4.1.3 set_neighbors6D()
def set_neighbors6D(pdict):
N = pdict['N']
Ca = pdict['Ca']
Cb = pdict['Cb']
nres = pdict['nres']
dmax = 20.0
# fast neighbors search
kdCb = scipy.spatial.cKDTree(Cb)
indices = kdCb.query_ball_tree(kdCb, dmax)
# indices of contacting residues
idx = np.array([[i,j] for i in range(len(indices)) for j in indices[i] if i != j]).T
idx0 = idx[0]
idx1 = idx[1]
# Cb-Cb distance matrix
dist6d = np.zeros((nres, nres))
dist6d[idx0,idx1] = np.linalg.norm(Cb[idx1]-Cb[idx0], axis=-1)
# matrix of Ca-Cb-Cb-Ca dihedrals
omega6d = np.zeros((nres, nres))
omega6d[idx0,idx1] = get_dihedrals(Ca[idx0], Cb[idx0], Cb[idx1], Ca[idx1])
# matrix of polar coord theta
theta6d = np.zeros((nres, nres))
theta6d[idx0,idx1] = get_dihedrals(N[idx0], Ca[idx0], Cb[idx0], Cb[idx1])
# matrix of polar coord phi
phi6d = np.zeros((nres, nres))
phi6d[idx0,idx1] = get_angles(Ca[idx0], Cb[idx0], Cb[idx1])
pdict['dist6d'] = dist6d
pdict['omega6d'] = omega6d
pdict['theta6d'] = theta6d
pdict['phi6d'] = phi6d
使用cKDTree进行快速邻居搜索
- 调用
scipy.spatial.cKDTree生成二维样本数据点,然后调用query_ball_tree方法进行快速查找邻点,求自身与他人之间距离不超过r的所有点对,dmax为context中定义的20.0
示意图:
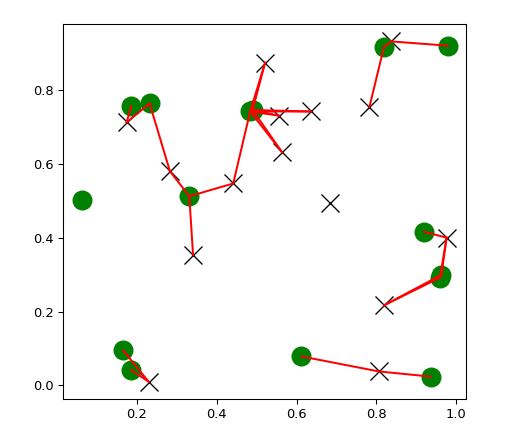
- 之后根据得到的点对计算联系残差指数
[[i,j] for i in range(len(indices)) for j in indices[i] if i != j]生成之后转置再保存至idx变量
- Cb-Cb距离矩阵
利用 np.zeros生成nres行,nres列的零矩阵,将Cb[idx1]-Cb[idx0]的范数保存至零矩阵的(idx0,idx1)元素中
5. 总结
本周主要工作是对特征实例化部分进行分析,之后争取进一步了解数学原理。