基于人工智能的多肽药物分析问题(七)
2021SC@SDUSC
前言:下文所需两种模式预测,预计可以在几篇文章之内结束分析
1. 两种预测模式
原程序提供了pyrosetta/e2e两种预测模式,根据Baek等的报道pyrosetta方法要比e2e更精确一些。 运行e2e的“主攻”是pytorch,需要RoseTTAFold 环境;pyrosetta的“主攻”包括pytorch、tensorflow、pyRosetta,需要RoseTTAFold和folding环境,而且pyRosetta需要在folding环境额外安装。tensorflow-gpu、pytorch需要特定版本的cudatoolkit、cudnn、python依赖包,所有的依赖包都可以在以下两个文件种找到:
RoseTTAFold-linux.yml
folding-linux.yml
由于硬件,限制了可以探索的模型的大小,替代的架构或损耗公式,或者更密集地使用网络进行推理。DeepMind报告表明,数天内使用多个GPU进行单独预测,该预测是通过网络以与服务器相同的方式进行的;在序列和模板搜索(约1.5小时)后,e2e版本RoseTTAFold需要在RTX2080 GPU上约10分钟才能为少于400个残基的蛋白质生成骨架坐标,pyRosetta版本在单个RTX2080 GPU上进行网络计算需要5分钟,在15个CPU核上生成所有原子结构需要1小时。
蛋白质复合物的预测
![[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-2bxh6aWl-1636811486494)(C:\Users\你好\AppData\Roaming\Typora\typora-user-images\image-20211113200718508.png)]](https://img-blog.csdnimg.cn/62ee4cde083049a8b354f3c64ef1b6e6.png?x-oss-process=image/watermark,type_ZHJvaWRzYW5zZmFsbGJhY2s,shadow_50,text_Q1NETiBA5oOz5Y-Y5oiQ5aSn5L2s55qE5bCP55m9,size_11,color_FFFFFF,t_70,g_se,x_16)
这边我们能看到,rosettaflod 除了提供上述两种预测模式pyrosetta和e2e外,还能够预测复合物的结构。
2. pyrosetta模式预测过程代码分析
2.1 参数准备
def get_args():
import argparse
parser = argparse.ArgumentParser()
parser.add_argument("-m", dest="model_dir", default="%s/weights"%(script_dir),
help="Path to pre-trained network weights [%s/weights]"%script_dir)
parser.add_argument("-i", dest="a3m_fn", required=True,
help="Input multiple sequence alignments (in a3m format)")
parser.add_argument("-o", dest="out_prefix", required=True,
help="Prefix for output file. The output file will be [out_prefix].npz")
parser.add_argument("--hhr", default=None,
help="HHsearch output file (HHsearch). If not provided, zero matrices will be given as templates")
parser.add_argument("--atab", default=None,
help="HHsearch output file (atab file)")
parser.add_argument("--db", default="%s/pdb100_2021Mar03/pdb100_2021Mar03"%script_dir,
help="Path to template database [%s/pdb100_2021Mar03]"%script_dir)
parser.add_argument("--cpu", dest='use_cpu', default=False, action='store_true')
args = parser.parse_args()
return args
这里主要还是对命令行参数做了一些解析,具体的作用可以参考本系列的第二篇文章,里面有较为详细的解释,此处不再赘述,仅介绍一下各个参数的用法:
-m :预先训练的网络权重的路径 (训练过程在本系列前面的文章已经解析过)
-i : 输入多个对比序列 ,以a3m的格式输入
-o : 输入文件名,预测系统会在文件名后自动补上后缀名 .npz
–hhr : HHsearch的输出文件 默认为0矩阵
–db: 模板数据库路径
–cpu: cpu数量
2.2 生成具名元组
args = get_args()
FFDB=args.db
FFindexDB = namedtuple("FFindexDB", "index, data")
ffdb = FFindexDB(read_index(FFDB+'_pdb.ffindex'),read_data(FFDB+'_pdb.ffdata'))
这里指定了元组名称为FFindexDB,元素名称为"index, data",FFDB为数据库路径
因为元组的局限性:不能为元组内部的数据进行命名,所以往往我们并不知道一个元组所要表达的意义,所以在这里引入了 namedtuple这个工厂函数,来构造一个带字段名的元组。具名元组的实例和普通元组消耗的内存一样多,因为字段名都被存在对应的类里面。这个类跟普通的对象实例比起来也要小一些,因为 Python 不会用 dict 来存放这些实例的属性。
返回一个具名元组子类 typename,其中参数的意义如下:
- typename:元组名称
- field_names: 元组中元素的名称
- rename: 如果元素名称中含有 python 的关键字,则必须设置为 rename=True
- verbose: 默认就好
利用生成的具名元组实例化了一个FFindexDB对象,该对象的index参数和data参数用两个方法来指定
def read_index(ffindex_filename):
entries = []
fh = open(ffindex_filename)
for line in fh:
tokens = line.split("\t")
entries.append(FFindexEntry(tokens[0], int(tokens[1]), int(tokens[2])))
fh.close()
return entries
def read_data(ffdata_filename):
fh = open(ffdata_filename, "rb")
data = mmap.mmap(fh.fileno(), 0, prot=mmap.PROT_READ)
fh.close()
return data
根据传入的文件名,解析相关数据的文件,存储在list中返回。
2.3 预测过程
if not os.path.exists("%s.npz"%args.out_prefix):
pred = Predictor(model_dir=args.model_dir, use_cpu=args.use_cpu)
pred.predict(args.a3m_fn, args.out_prefix, args.hhr, args.atab)
首先实例化了Predictor类,该类的成员变量model_dir也就是训练完毕的模型文件的路径,定义模型的名字为RoseTTAFold
def __init__(self, model_dir=None, use_cpu=False):
if model_dir == None:
self.model_dir = "%s/models"%(os.path.dirname(os.path.realpath(__file__)))
else:
self.model_dir = model_dir
self.model_name = "RoseTTAFold"
if torch.cuda.is_available() and (not use_cpu):
self.device = torch.device("cuda")
else:
self.device = torch.device("cpu")
self.active_fn = nn.Softmax(dim=1)
self.model = RoseTTAFoldModule(**MODEL_PARAM).to(self.device)
could_load = self.load_model(self.model_name)
if not could_load:
print ("ERROR: failed to load model")
sys.exit()
在Predictor类的构造函数中,对硬件和模型配置作了一些处理:
- 自动检测电脑的cuda是否能使用,若能够使用并且命令行未指定使用cpu跑模型,那么则利用cuda跑模型。
- 设置了模型的激活函数active function 为Softmax,维度为1
- 加载自定义的RoseTTAFoldModule模型
实例化RoseTTAFoldModule模型的参数
关于RoseTTAFoldModule模型见下一小节
![[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-TyyPeKi9-1636811486497)(C:\Users\你好\AppData\Roaming\Typora\typora-user-images\image-20211113213520020.png)]](https://img-blog.csdnimg.cn/2272d05b4e3940a2a9db1f6d94868585.png?x-oss-process=image/watermark,type_ZHJvaWRzYW5zZmFsbGJhY2s,shadow_50,text_Q1NETiBA5oOz5Y-Y5oiQ5aSn5L2s55qE5bCP55m9,size_9,color_FFFFFF,t_70,g_se,x_16)
之后调用load_model方法检测相关文件路径能否正确加载数据,是则继续运行,否则结束运行。
def load_model(self, model_name, suffix='pyrosetta'):
chk_fn = "%s/%s_%s.pt"%(self.model_dir, model_name, suffix)
if not os.path.exists(chk_fn):
return False
checkpoint = torch.load(chk_fn, map_location=self.device)
self.model.load_state_dict(checkpoint['model_state_dict'], strict=False)
return True
2.4 RoseTTAFoldModule模型
由于之前未学习过pytorch相关,再结合之前所学tensorflow相关知识,现补充pytorch知识点:
pytorch中对于一般的序列模型,直接使用torch.nn.Sequential类及可以实现,这点类似于tensorflow 实现的keras,但是更多的时候面对复杂的模型,比如:多输入多输出、多分支模型、跨层连接模型、带有自定义层的模型等,就需要自己来定义一个模型了。本文将详细说明如何让使用Mudule类来自定义一个模型,本次预测所用的到rosettafold即需要自定义。
二者区别:
keras更常见的操作是通过继承Layer类来实现自定义层,不推荐去继承Model类定义模型,详细原因可以参见官方文档
pytorch中其实一般没有特别明显的Layer和Module的区别,不管是自定义层、自定义块、自定义模型,都是通过继承Module类完成的,这一点很重要。其实Sequential类也是继承自Module类的。
注意:我们当然也可以直接通过继承torch.autograd.Function类来自定义一个层。
总结:pytorch里面一切自定义操作基本上都是继承nn.Module类来实现的
自定义网络的必要部分:
我们在定义自已的网络的时候,需要继承nn.Module类,并重新实现构造函数__init__构造函数和forward这两个方法。但有一些注意技巧:
(1)一般把网络中具有可学习参数的层(如全连接层、卷积层等)放在构造函数__init__()中,当然我也可以把不具有参数的层也放在里面;(2)一般把不具有可学习参数的层(如ReLU、dropout、BatchNormanation层)可放在构造函数中,也可不放在构造函数中,如果不放在构造函数__init__里面,则在forward方法里面可以使用nn.functional来代替
(3)forward方法是必须要重写的,它是实现模型的功能,实现各个层之间的连接关系的核心。
2.4.1 构造方法
在实例化RoseTTAFoldModule模型的构造方法中,定义了一些网络结构:MSA_emb,Templ_emb,Pair_emb_w_templ,IterativeFeatureExtractor,DistanceNetwork等层次
2.4.2 Embedding层
class MSA_emb(nn.Module):
def __init__(self, d_model=64, d_msa=21, p_drop=0.1, max_len=5000):
super(MSA_emb, self).__init__()
self.emb = nn.Embedding(d_msa, d_model)
self.pos = PositionalEncoding(d_model, p_drop=p_drop, max_len=max_len)
self.pos_q = QueryEncoding(d_model)
def forward(self, msa, idx):
B, N, L = msa.shape
out = self.emb(msa)
out = self.pos(out, idx)
return self.pos_q(out)
-
定义了嵌入层Embedding,指定词嵌入字典大小为21,每个词嵌入向量大小为64(关于嵌入层的作用,会在之后的文章进行分享)
-
生成坐标数据层,为Embedding层提供位置信息
class PositionalEncoding(nn.Module):
def __init__(self, d_model, p_drop=0.1, max_len=5000):
super(PositionalEncoding, self).__init__()
self.drop = nn.Dropout(p_drop,inplace=True)
pe = torch.zeros(max_len, d_model)
position = torch.arange(0, max_len).unsqueeze(1)
div_term = torch.exp(torch.arange(0, d_model, 2) *
-(math.log(10000.0) / d_model))
pe[:, 0::2] = torch.sin(position * div_term)
pe[:, 1::2] = torch.cos(position * div_term)
pe = pe.unsqueeze(0)
self.register_buffer('pe', pe) # (1, max_len, d_model)
def forward(self, x, idx_s):
pe = list()
for idx in idx_s:
pe.append(self.pe[:,idx,:])
pe = torch.stack(pe)
x = x + torch.autograd.Variable(pe, requires_grad=False)
return self.drop(x)
在该模型中,定义了drpout,作用是:在训练过程的前向传播中,让每个神经元以一定概率p处于不激活的状态。以达到减少过拟合的效果。然后对position的第二维度新增了一个维度,生成了64个元素的二维数组。之后进行了部分数学运算:
![[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-KyFr7LOm-1636860019165)(file:///C:/Temp/msohtmlclip1/01/clip_image002.png)]](https://img-blog.csdnimg.cn/b420419b68654264b5803f312f6b44a7.png)
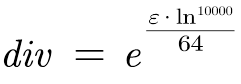
然后对position和div的乘积取sin值和cos值,交替插入到pe变量中,再将pe提升至三维变量。
然后调用了register_buffer,register_buffer主要用于当希望模型中的某些参数参数不更新(从开始到结束均保持不变),但又希望参数保存下来(model.state_dict() ),这样,该部分参数不会随着权重参数一起更新。
- 建立查询嵌入层
class QueryEncoding(nn.Module):
def __init__(self, d_model):
super(QueryEncoding, self).__init__()
self.pe = nn.Embedding(2, d_model) # (0 for query, 1 for others)
def forward(self, x):
B, N, L, K = x.shape
idx = torch.ones((B, N, L), device=x.device).long()
idx[:,0,:] = 0 # first sequence is the query
x = x + self.pe(idx)
return x
该模型中仅建立了一层Embedding层,用于分辨查询任务和其他任务。
到此建立好了Predictor类中的msa_emb模型
2.4.3 template Embedding层
class Templ_emb(nn.Module):
def __init__(self, d_t1d=3, d_t2d=10, d_templ=64, n_att_head=4, r_ff=4,
performer_opts=None, p_drop=0.1, max_len=5000):
super(Templ_emb, self).__init__()
self.proj = nn.Linear(d_t1d * 2 + d_t2d + 1, d_templ)
self.pos = PositionalEncoding2D(d_templ, p_drop=p_drop)
# attention along L
enc_layer_L = AxialEncoderLayer(d_templ, d_templ * r_ff, n_att_head, p_drop=p_drop,
performer_opts=performer_opts)
self.encoder_L = Encoder(enc_layer_L, 1)
self.norm = LayerNorm(d_templ)
self.to_attn = nn.Linear(d_templ, 1)
在该模型中,建立了一个线性层,位置关系编码层,以及像素编码层等等,下篇文章再作具体分析。
(好复杂的结构!!)
3. 总结
本篇文章主要讲述了前半部分的神经网络结构,由于涉及到pytorch具体知识,所以后续文章会额外补充相关知识点。