写在前面的话
本文是对nnUNet如何在2D数据上进行训练以及预测,是对几篇博文参考之后的总结,由于本人对nnUNet使用也是刚开始不久,所以对于nnUNet的理解肯定还停留在表层,所以原理上的东西就不多介绍了,更侧重于介绍其在2D数据上的使用流程。官方也有nnUNet训练2D数据的例子,一切以官方为准,本文仅作参考。
本人的系统环境为:Ubuntu18.04,pytorch为:1.7.1+cu110。其余环境没有测试过。
参考文章:
- 保姆级教程:nnUnet在2维图像的训练和测试
- 四:2020.07.28)nnUNet最舒服的使用教程(让我的奶奶也会用nnUNet)
- 不用写代码神器!教你用4行命令轻松使用nnUNet训练自己的医学图像分割模型
一、 nnUNet简介
nnUNet是一种基于2D and 3D 原始U-Nets 自适应框架,该框架能根据给定数据集的属性自动调整所有超参数,整个过程无需人工干预。仅仅依赖于朴素的U-Net结构(就是原始U-Net)和鲁棒的训练方案,nnU-Net在六个得到公认的分割挑战中实现了最先进的性能。
github仓库:https://github.com/MIC-DKFZ/nnUNet
paper:《Automated Design of Deep Learning Methods for Biomedical Image Segmentation》
nnUNet是德国癌症研究中心的工程师编写的框架,迄今为止依旧在维护和更新。虽然整个过程实现自动化,但是对GPU资源要求比较高,虽然官方说For training nnU-Net models the GPU should have at least 10 GB,但是实测后发现,如果按默认配置,需要19G显存。如果显存不足可以考虑降低batch size大小。
二、nnUNet安装
由于本人避免python环境冲突,用conda创建一个虚拟环境nnUNet,并激活虚拟环境
conda create -n nnUNet python=3.8
source activate nnUnet
由于是面向初学者的使用教程,初学者请务必按照我的做法,等你熟练掌握以后再考虑新的姿势(有些文件夹的创建时多余的,但是你还是跟着我这样做最好)。
第一步:在某个目录下(比如说/data)创建nnUNetFrame目录,在nnUNet的终端环境下cd到这个nnUNetFrame目录,然后git clone https://github.com/MIC-DKFZ/nnUNet.git
第二步:cd nnUNet
第三步:pip install -e .(兄弟们和集美们别忘了加 . )
当你安装完成这些以后,你的每一次对nnUNet的操作,都会在命令行里以nnUNet_开头,代表着你的nnUNet开始工作的指令。
接下来是创建数据保存目录。
- 进入你之前创建的nnUNetFrame文件夹里面,创建一个名为DATASET的文件夹,现在你的nnUNetFrame文件夹下有两个文件夹,nnUNet是代码源,另一个DATASET就是我们接下来用来放数据的地方;
- 进入创建好的DATASET文件夹下面,创建下面三个文件夹分别是nnUNet_preprocessed、nnUNet_raw、nnUNet_trained_models
第一个用来存放原始数据预处理之后的数据,第二个用来存放原始的你要训练的数据,第三个用来存放训练的结果。 - 进入上面第二个文件夹nnUNet_raw,创建下面两个文件夹,分别是nnUNet_cropped_data、nnUNet_cropped_data,右边为原始数据,左边为crop以后的数据。
- 进入右边文件夹nnUNet_raw_data,创建一个名为Task001_BloodVessel的文件夹(解释:这nnUnet的数据格式是固定的,Task001_BloodVessel由Task+ID+数据名组成,你可以对这个任务的数字ID进行任意的命名,比如你要分割心脏,你可以起名为Task001_Heart,比如你要分割肾脏,你可以起名为Task002_Kidney,前提是必须按照这种格式)
三、 数据准备
3.1 2D数据转3D数据
假定我们的数据长这样,图片和mask分别存放在img和mask的文件夹下,两者同名。
由于这是2D图片数据,要将其转为3D数据,其实就是z轴为1的3维数据。
所以随意新建一个项目,创建一个2DDataProcessTo3D.py脚本
import os
import random
from tqdm import tqdm
import SimpleITK as sitk
import cv2
import numpy as np
def SplitDataset(img_path, train_percent=0.9):
data = os.listdir(img_path)
train_images = []
test_images = []
num = len(data)
train_num = int(num * train_percent)
indexes = list(range(num))
train = random.sample(indexes, train_num)
for i in indexes:
if i in train:
train_images.append(data[i])
else:
test_images.append(data[i])
return train_images, test_images
def conver(img_path, save_dir, mask_path=None, select_condition=None, mode="trian"):
os.makedirs(save_dir, exist_ok=True)
if mode == "train":
savepath_img = os.path.join(save_dir, 'imagesTr')
savepath_mask = os.path.join(save_dir, 'labelsTr')
elif mode == "test":
savepath_img = os.path.join(save_dir, 'imagesTs')
savepath_mask = os.path.join(save_dir, 'labelsTs')
os.makedirs(savepath_img, exist_ok=True)
if mask_path is not None:
os.makedirs(savepath_mask, exist_ok=True)
ImgList = os.listdir(img_path)
with tqdm(ImgList, desc="conver") as pbar:
for name in pbar:
if select_condition is not None and name not in select_condition:
continue
Img = cv2.imread(os.path.join(img_path, name))
if mask_path is not None:
Mask = cv2.imread(os.path.join(mask_path, name), 0)
Mask = (Mask / 255).astype(np.uint8)
if Img.shape[:2] != Mask.shape:
Mask = cv2.resize(Mask, (Img.shape[1], Img.shape[0]))
Img_Transposed = np.transpose(Img, (2, 0, 1))
Img_0 = Img_Transposed[0].reshape(1, Img_Transposed[0].shape[0], Img_Transposed[0].shape[1])
Img_1 = Img_Transposed[1].reshape(1, Img_Transposed[1].shape[0], Img_Transposed[1].shape[1])
Img_2 = Img_Transposed[2].reshape(1, Img_Transposed[2].shape[0], Img_Transposed[2].shape[1])
if mask_path is not None:
Mask = Mask.reshape(1, Mask.shape[0], Mask.shape[1])
Img_0_name = name.split('.')[0] + '_0000.nii.gz'
Img_1_name = name.split('.')[0] + '_0001.nii.gz'
Img_2_name = name.split('.')[0] + '_0002.nii.gz'
if mask_path is not None:
Mask_name = name.split('.')[0] + '.nii.gz'
Img_0_nii = sitk.GetImageFromArray(Img_0)
Img_1_nii = sitk.GetImageFromArray(Img_1)
Img_2_nii = sitk.GetImageFromArray(Img_2)
if mask_path is not None:
Mask_nii = sitk.GetImageFromArray(Mask)
sitk.WriteImage(Img_0_nii, os.path.join(savepath_img, Img_0_name))
sitk.WriteImage(Img_1_nii, os.path.join(savepath_img, Img_1_name))
sitk.WriteImage(Img_2_nii, os.path.join(savepath_img, Img_2_name))
if mask_path is not None:
sitk.WriteImage(Mask_nii, os.path.join(savepath_mask, Mask_name))
if __name__ == "__main__":
train_percent = 0.9
img_path = r".../img"
mask_path = r".../mask"
output_folder = r"./dataset"
os.makedirs(output_folder, exist_ok=True)
train_images, test_images = SplitDataset(img_path, train_percent)
conver(img_path, output_folder, mask_path, train_images, mode="train")
conver(img_path, output_folder, mask_path, test_images, mode="test")
一些参数解释:
train_percent:训练集和验证集划分比例
img_path:图片路径
mask_path:mask路径
output_folder:保存路径
几个注意的点:
imagesTr是训练数据,imagesTs是测试数据,labelsTr是训练数据的标签,labelsTs是测试数据的标签(这个可能没什么用),数据样本la_003_0000.nii.gz由case样本名字+模态标志+.nii.gz组成,不同的模态用0000/0001/0002/0003区分。
示例树结构:
nnUNet_raw_data_base/nnUNet_raw_data/Task001_BloodVessel
├── dataset.json
├── imagesTr
│ ├── la_003_0000.nii.gz
│ ├── la_004_0000.nii.gz
│ ├── ...
├── imagesTs
│ ├── la_001_0000.nii.gz
│ ├── la_002_0000.nii.gz
│ ├── ...
└── labelsTr
├── la_003.nii.gz
├── la_004.nii.gz
├── ...
我们的原始2维数据是RGB三通道的,我们可以把RGB三通道的数据看成3个模态,分别提取不同通道的数据,形状转换成(1,width, height),采用SimpleITK保存为3维数据。
imagesTr:
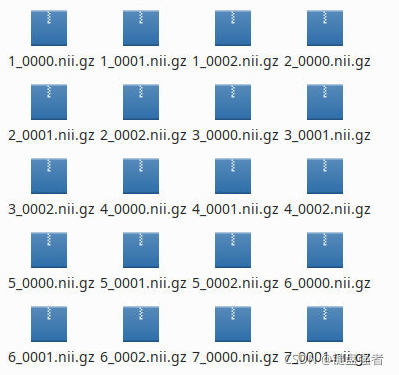
labelsTr:
3.2 制作dataset.json
dataset.json的文件内容是包含数据集的元数据, 如任务名字,模态,标签含义,训练集包含的图像地址。
这里讲一下,dataset.json这个文件怎么弄。一个合格的文件应该包含如下信息:
name: 数据集名字
dexcription: 对数据集的描述
modality: 模态,0表示CT数据,1表示MR数据。nnU-Net会根据不同模态进行不同的预处理
labels: label中,不同的数值代表的类别
numTraining: 训练集数量
numTest: 测试集数量
training: 训练集的image 和 label 地址对
test: 只包含测试集的image. 这里跟Training不一样

最简单的方法就是copy别人的代码,在前人的基础上修改一下。如: dataset_conversion
最终效果如图(图片来源:保姆级教程:nnUnet在2维图像的训练和测试):
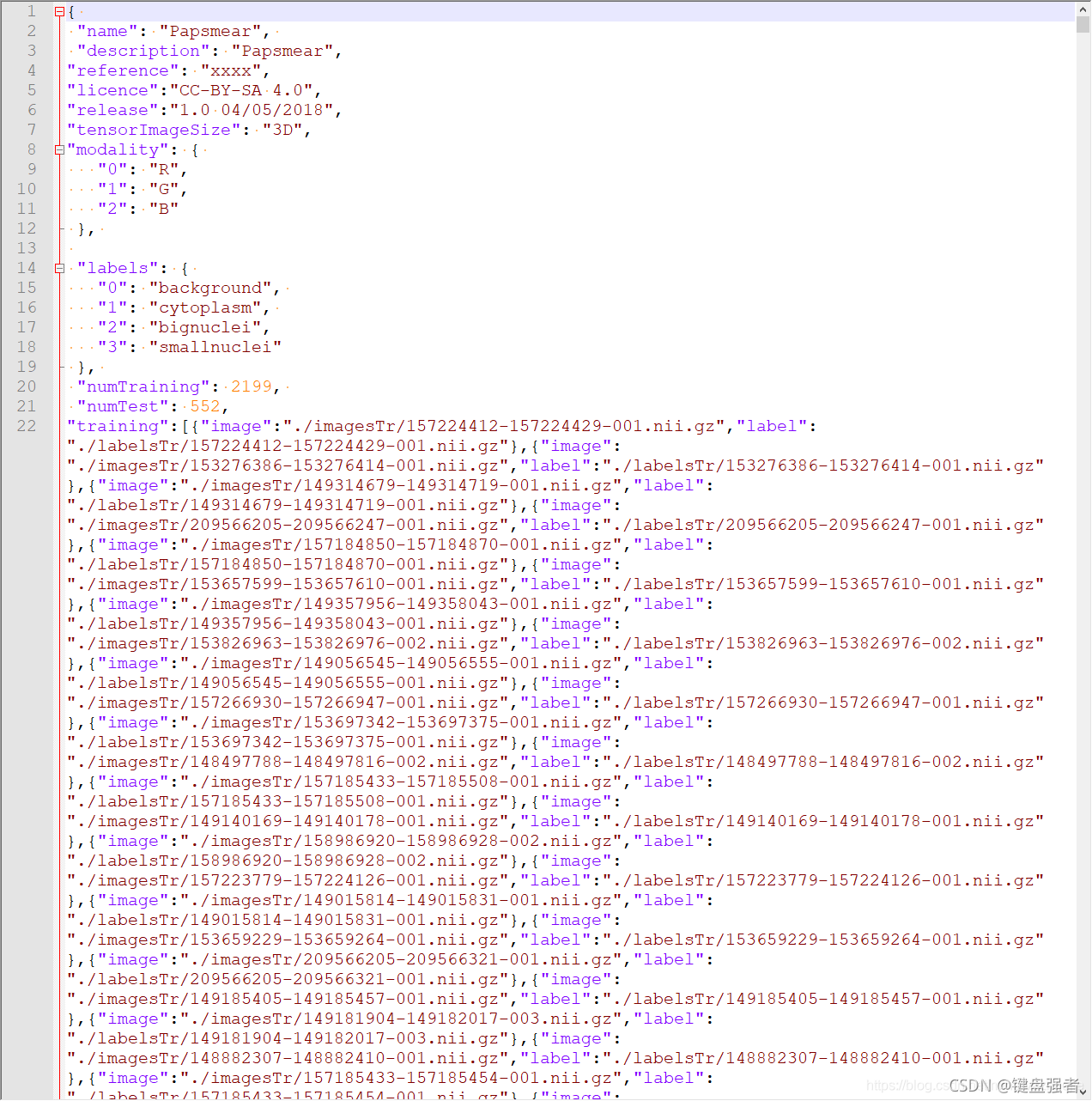
dataset.json里的数据名去掉了模态标志,所以每个数据看起来有三个重复名字,在后面读图的时候会自动添加模态标志。还有name这一行是你任务数据集的名字,比如说,你在nnUNet_raw_data创建一个名为Task001_BloodVessel,那么这个name就是BloodVessel。
3.3 设置数据集环境变量
首先需要将2DDataProcessTo3D.py脚本中output_folder目录下的,imagesTr、imagesTs、labelsTr、labelsTs和制作好的dataset.json复制到nnUNet_raw_data/Task001_BloodVessel(假定你任务的名字就是这个,当然还可以命名成其他,具体请参照上面)。
在终端中运行导出命令,设置环境变量:
export nnUNet_raw_data_base="/data/nnUnet/Data/nnUNet_raw"
export nnUNet_preprocessed="/data/nnUnet/Data/nnUNet_preprocessed"
export RESULTS_FOLDER="/data/nnUnet/Data/nnUNet_trained_models"
假定你的目录是创建在/data下的。
四、数据预处理
nnUNet_plan_and_preprocess -t 100 -data23d 2 --verify_dataset_integrity
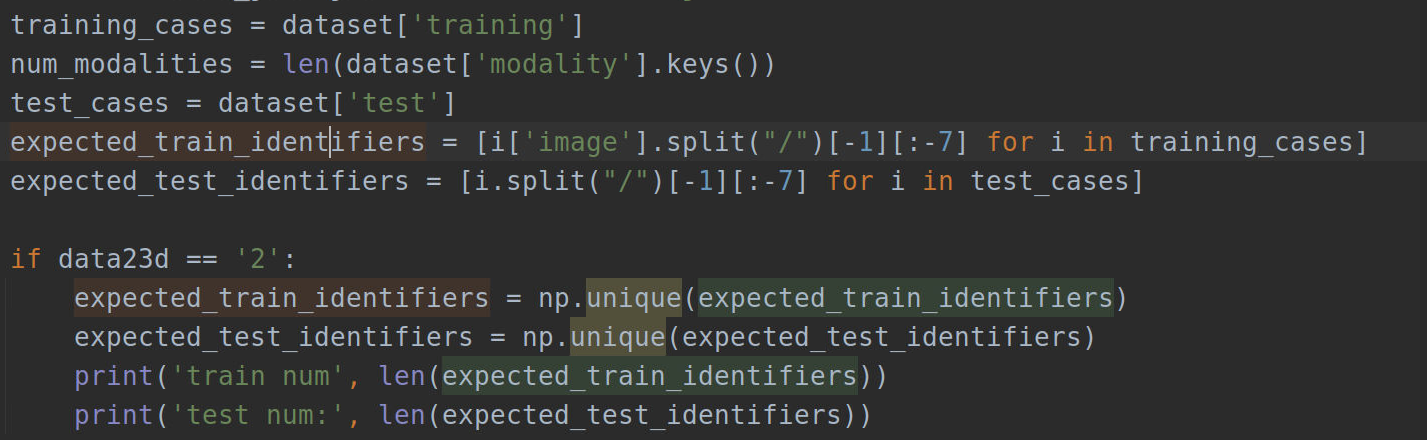
这里根据保姆级教程:nnUnet在2维图像的训练和测试所说增加了一个设置参数data23d用来指定要处理的是2维数据还是3维数据,在nnunet/preprocessing/sanity_checks.py中的verify_dataset_integrity函数中增加做如下设置:
if data23d == '2':
expected_train_identifiers = np.unique(expected_train_identifiers)
expected_test_identifiers = np.unique(expected_test_identifiers)
print('train num', len(expected_train_identifiers))
print('test num:', len(expected_test_identifiers))
具体位置为:
此外,由于新增了一个参数,还需要在nnunet/experiment_planning/nnUNet_plan_and_preprocess.py中添加
efault=3, help="the data is 2d or 3d")
并且在传参一行要加入
if args.verify_dataset_integrity:
verify_dataset_integrity(join(nnUNet_raw_data, task_name), args.data23d)
五、训练
训练时逐条运行下面命令
CUDA_VISIBLE_DEVICES=1 nnUNet_train 2d nnUNetTrainerV2 Task001_BloodVessel 0 --npz
CUDA_VISIBLE_DEVICES=1 nnUNet_train 2d nnUNetTrainerV2 Task001_BloodVessel 1 --npz
CUDA_VISIBLE_DEVICES=1 nnUNet_train 2d nnUNetTrainerV2 Task001_BloodVessel 2 --npz
CUDA_VISIBLE_DEVICES=1 nnUNet_train 2d nnUNetTrainerV2 Task001_BloodVessel 3 --npz
CUDA_VISIBLE_DEVICES=1 nnUNet_train 2d nnUNetTrainerV2 Task001_BloodVessel 4 --npz
六、预测
6.1 模型预测
运行完5折交叉验证后,可以确定最佳的配置,下面的008是Task的ID,我们这个任务是008
nnUNet_find_best_configuration -m 2d -t 008 –strict
运行后会在路径nnUNet_trained_models/nnUNet/ensembles/Task001_BloodVessel下生成
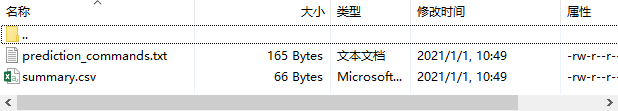
然后打开上面的txt文件,里面会生成预测方法:
nnUNet_predict -i FOLDER_WITH_TEST_CASES -o OUTPUT_FOLDER_MODEL1 -tr nnUNetTrainerV2 -ctr nnUNetTrainerV2CascadeFullRes -m 2d -p nnUNetPlansv2.1 -t Task001_BloodVessel
INPUT_FOLDER: 测试数据地址
OUTPUT_FOLDER: 分割数据存放地址
CONFIGURATION: 使用的什么架构,2d or 3d_fullres or 3d_cascade_fullres等
所以最终的预测命令就是:
nnUNet_predict -i /data/nnUnet/Data/nnUNet_raw/nnUNet_raw_data/Task001_BloodVessel/imagesTs/ -o /data/nnUnet/Data/nnUNet_raw/nnUNet_raw_data/Task001_BloodVessel/imagesTsPred/ -tr nnUNetTrainerV2 -ctr nnUNetTrainerV2CascadeFullRes -m 2d -p nnUNetPlansv2.1 -t Task08_HepaticVessel
就会在imagesTsPred生成五折交叉验证的结果。
6.2 还原结果
因为模型预测的结果时nii.gz文件,所以需要将其转成2D的图像数据
所以新建一个3DDataProcessTo2D.py的脚本,并计算dice
import os
from tqdm import tqdm
import SimpleITK as sitk
import cv2
import numpy as np
from sklearn.metrics import f1_score
def conver(img_dir, output_dir):
os.makedirs(output_dir, exist_ok=True)
img_list = [i for i in os.listdir(img_dir) if ".nii.gz" in i]
with tqdm(img_list, desc="conver") as pbar:
for name in pbar:
image = sitk.ReadImage(os.path.join(img_dir, name))
image = sitk.GetArrayFromImage(image)[0]
image = (image * 255).astype(np.uint8)
cv2.imwrite(os.path.join(output_dir, name.split(".")[0]+".png"), image)
def computer_dice(mask_dir, label_dir):
dice_list = []
name_list = os.listdir(mask_dir)
with tqdm(name_list, desc="dice") as pbar:
for name in pbar:
mask = cv2.imread(os.path.join(mask_dir, name), cv2.IMREAD_GRAYSCALE)
label = cv2.imread(os.path.join(label_dir, name), cv2.IMREAD_GRAYSCALE)
if mask.shape != label.shape:
mask = cv2.resize(mask, (label.shape[1], label.shape[0]))
mask = (mask / 255).ravel().astype(np.int)
label = (label / 255).ravel().astype(np.int)
dice = f1_score(y_true=label, y_pred=mask)
dice_list.append(dice)
print(sum(dice_list)*1.0/len(dice_list))
if __name__ == "__main__":
img_dir = "/data/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BloodVessel/imagesTsPred"
output_dir = "/data/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BloodVessel/imagesTsPredMask"
conver(img_dir, output_dir)
label_dir = "..../train_dataset/mask"
computer_dice(output_dir, label_dir)