说明:这是一个机器学习实战项目(附带数据+代码+文档+代码讲解),如需数据+代码+文档+代码讲解可以直接到文章最后获取。
?
?
1.项目背景
? ? ? ?gcForest(多粒度级联森林)是一种深度森林结构。近年来,深度神经网络在图像和声音处理领域取得了很大的进展。关于深度神经网络,我们可以把它简单的理解为多层非线性函数的堆叠,当我们人工很难或者不想去寻找两个目标之间的非线性映射关系,我们就多堆叠几层,让机器自己去学习它们之间的关系,这就是深度学习最初的想法。既然神经网络可以堆叠为深度神经网络,那我们可以考虑,是不是可以将其他的学习模型堆叠起来,以获取更好的表示性能,gcForest就是基于这种想法提出来的一种深度结构。gcForest通过级联的方式堆叠多层随机森林,以获得更好的特征表示和学习性能。
2.数据获取
本次建模数据来源于网络(本项目撰写人整理而成),数据项统计如下:
数据详情如下(部分展示):?
3.数据预处理
3.1 用Pandas工具查看数据
使用Pandas工具的head()方法查看前五行数据:
?
关键代码:
?
3.2查看数据集摘要
使用Pandas工具的info()方法查看数据集的摘要信息:
<class 'pandas.core.frame.DataFrame'> RangeIndex: 718 entries, 0 to 717 Data columns (total 29 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 age 718 non-null int64 1 gender 718 non-null int64 2 body_mass_index 718 non-null float64 3 heart_failure 718 non-null int64 4 hypertension 718 non-null int64 5 chronic_obstructic_pulmonary_disease 718 non-null int64 6 chronic_liver_disease 718 non-null int64 7 diabetes_mellitus 718 non-null int64 8 chroinc_kidney_disease 718 non-null int64 9 charlson 718 non-null int64 10 emergency 718 non-null int64 11 surgery 718 non-null int64 12 APSIII 718 non-null int64 13 SAPSII 718 non-null int64 14 non_renal_sofa-1 718 non-null int64 15 non_renal_sofa-3 718 non-null int64 16 non_renal_sofa 718 non-null int64 17 aki_stage 718 non-null int64 18 creatinine_baseline 718 non-null float64 19 creatinine-1 718 non-null float64 20 creatinine-3 718 non-null float64 21 creatinine 718 non-null float64 22 urine_output-1 718 non-null float64 23 urine_output-3 718 non-null float64 24 urine_output 718 non-null float64 25 diuretic 718 non-null int64 26 mechanical_ventalition 718 non-null int64 27 renal_toxic_drug 718 non-null int64 28 acute_kidney_disease 718 non-null int64 dtypes: float64(8), int64(21) memory usage: 162.8 KB从上表可以看到,总共有718条数据,29个数据项,所有数据中没有缺失值。
关键代码:
4.探索性数据分析
4.1检查目标变量的分布
用Pandas工具的value_counts()方法进行统计,输出结果如下:
?
图形化展示如下:
?
从上面两个图中可以看到,分类为1的有352条/分类为0的有366条,数据偏差不大。另外,可以看到这是一个二分类的任务。
关键代码:
4.2 相关性分析
用Pandas工具的corr()方法 matplotlib seaborn进行相关性分析,结果如下:
?
?
通过上图可以看到,数据项之间正值是正相关/负值是负相关,数值越大 相关性越强。另外通过上面两个图的颜色也可以直观地看出,第二张图的数据项之间的相关性更强。
5.特征工程
5.1 建立特征数据和标签数据
acute_kidney_disease为标签数据,除 acute_kidney_disease之外的为特征数据。关键代码如下:
?
5.2数据集拆分
训练集拆分,分为训练集和验证集,80%训练集和20%验证集。关键代码如下:
?
6.构建GCForest建模 ?
6.1建模
模型参数如下:
?关键代码如下:
7.模型评估
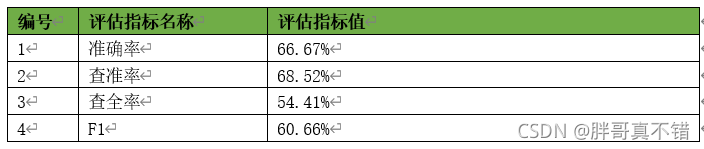
7.1评估指标及结果
评估指标主要采用准确率、查准率、查全率、F1分值。
通过上述表格可以看出,准确率为66.67%,F1分值为60.66%;大家可以进一步优化;如果替换成其它数据集效果会更好,因为我提供的这个数据集里面有很多分类的变量未进行进一步的预处理。
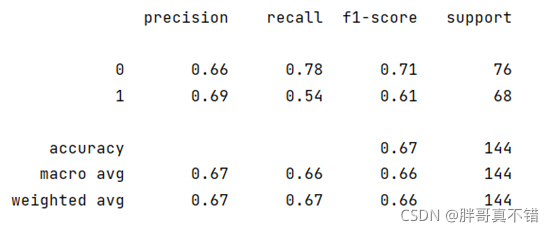
?7.2 分类报告
通过上图可以看到,分类为0的F1分数为0.71,分类为1的F1分数为0.61,准确率为67%。
7.3 ROC曲线
?
通过上图可以看到,GCForest模型的AUC值为0.72,说明整体效果还是很不错的,如果把数据集在进行预处理一下,AUC的值会更高。
8.结论与展望
根据测试集的特征数据,来预测这些患者是否会有相关疾病;根据预测结果:针对将来可能会患有此种疾病的人员,提前进行预防。
注意事项:
GCForest.py这个是实现多粒度级联森林模型的源代码,用的时候和其它代码放在同一个目录,避免报错:找不到GCForest模块。
本次机器学习项目实战所需的资料,项目资源如下:
项目说明:
链接:https://pan.baidu.com/s/13r3-mTcCRBfwWRtbpnFUpw?
提取码:s2wn网盘如果失效,可以添加博主微信:zy10178083