创新实训-生物大分子序列分析平台09
2021SC@SDUSC
在生物信息学中,一些药物分子和蛋白质结构经常用图结构进行表示,因此本周了解了一些常见的图神经网络结构。
图注意力神经网络代码
pytorch代码链接
所谓注意力,就是指对信息重要性的分配,在图中,一个点的邻接节点对它的重要性是不同的。
i和j是两个邻接节点,α
i
j
ij
ij为节点i对节点j的注意力分数。
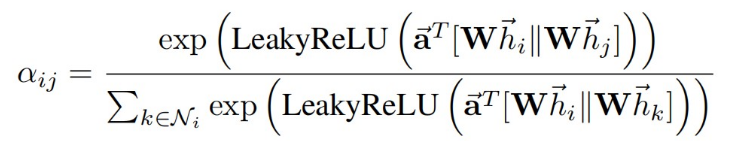
节点特征向量与矩阵相乘,即进行线性变换得到Wh。_prepare_attentional_mechanism_input函数求算的就是上面公式中的
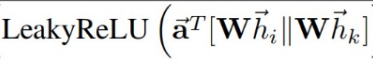
相当于将每个节点向量与其它节点向量拼接,再乘上a。
torch.where(condition, x, y):condition是条件,x 和 y 是同shape 的矩阵, 针对矩阵中的某个位置的元素, 满足条件就返回x,不满足就返回y。判断adj是否大于0,可只计算邻接节点。
最后做一个softmax进行归一化和dropout操作,然后乘上特征矩阵输出。
class GraphAttentionLayer(nn.Module):
"""
Simple GAT layer, similar to https://arxiv.org/abs/1710.10903
"""
def __init__(self, in_features, out_features, dropout, alpha, concat=True):
super(GraphAttentionLayer, self).__init__()
self.dropout = dropout
self.in_features = in_features
self.out_features = out_features
self.alpha = alpha
self.concat = concat
self.W = nn.Parameter(torch.empty(size=(in_features, out_features)))
nn.init.xavier_uniform_(self.W.data, gain=1.414)
self.a = nn.Parameter(torch.empty(size=(2*out_features, 1)))
nn.init.xavier_uniform_(self.a.data, gain=1.414)
self.leakyrelu = nn.LeakyReLU(self.alpha)
def forward(self, h, adj):
Wh = torch.mm(h, self.W) # h.shape: (N, in_features), Wh.shape: (N, out_features)
e = self._prepare_attentional_mechanism_input(Wh)
zero_vec = -9e15*torch.ones_like(e)
attention = torch.where(adj > 0, e, zero_vec)
attention = F.softmax(attention, dim=1)
attention = F.dropout(attention, self.dropout, training=self.training)
h_prime = torch.matmul(attention, Wh)
if self.concat:
return F.elu(h_prime)
else:
return h_prime
def _prepare_attentional_mechanism_input(self, Wh):
# Wh.shape (N, out_feature)
# self.a.shape (2 * out_feature, 1)
# Wh1&2.shape (N, 1)
# e.shape (N, N)
Wh1 = torch.matmul(Wh, self.a[:self.out_features, :])
Wh2 = torch.matmul(Wh, self.a[self.out_features:, :])
# broadcast add
e = Wh1 + Wh2.T
return self.leakyrelu(e)
def __repr__(self):
return self.__class__.__name__ + ' (' + str(self.in_features) + ' -> ' + str(self.out_features) + ')'