arrays 掩码
将要完成的事:
使用Numpy 里的 arrays掩码模块分析COVID-19的数据并处理丢失的数据
可以学到的内容
- 理解什么是arrays掩码,并且是怎么创建出来的
- 如何访问和修改掩码数组里的数据
- 学会在何时使用arrays掩码是最合适的
什么是arrays 掩码
思考以下问题:你有个数据集里面有部分数据丢失或者是无效的。而你想对这些数据进行处理,并且对不想要的数据跳过或者进行标记而不是直接删除他们,你可能使用条件语句过滤数据。
numpy.ma模块提供了一些Numoy ndarrays相同的功能,b并为数据添加了一种使得无效条目不会参与计算的结构。
掩码数组是标准 numpy.ndarray 和掩码的组合。掩码要么是 nomask,表示关联数组的任何值都无效,要么是一个布尔数组,用于确定关联数组的每个元素的值是否有效。当掩码的某个元素为 False 时,关联数组的对应元素是有效的,称为未掩码。当掩码的元素为 True 时,关联数组的对应元素被称为被掩码(无效)。
可将MaskedArray视为以下的组合:
- data,任何形状或数量类型的常规numpy.ndarray;
- boolean mask,与数据形状相同的布尔掩码;
- fill_value,一个可用于替换无效条目以返回标准numpy.ndarray的值。
实例:使用arrays 掩码查看COVID-19的数据
import numpy as np
import os
# os.getcwd() function returns the current folder;you can change
# the filepath variable to point to the folder where you saved the .csv file
filepath = os.getcwd()
filename = os.path.join(filepath,"file_name")
'''
该数据文件包含不同类型的数据,其组织方式如下:
1.第一行是标题行,主要描述了各行中每列的数据,从第四列开始标明了观察日期;
2.第二行到第六行包含了将要检查数据类型不同的汇总数据,因此需要将其从将使用的数据中排除;
3.我们希望处理的数值数据从第四列第七行开始,并从那里延伸到最右侧的列和最下方的行。
'''
# skip_header and usecols to read only portions **of the data file** into each variable.
# read just the datas for columns 4-18 from the first row
dates = np.genfromtxt(
filename,
dtype = np.unicode_,
delimiter=',',
max_rows=1,
usecols=range(4,18),
encoding="utf-8-sig",
)
#read the names of the geographic locations from the first two colums,skipping the first six rows
locations = np.genfromtxt(
filename,
dtype=np.unicode_,
delimiter=",",
skip_header=6,
usecols=(0,1),
encoding="utf-8-sig",
)
#read the numeric data from just the first 14 days
nbcases = np.genfromtxt(
filename,
dtype=np.int_,
delimiter=",",
skip_header=6,
usecols=range(4,18),
encoding="utf-8-sig",
)
Exploring the data
import matplotlib.pyplot as plt
select_dates = [0, 3, 11, 13]
plt.plot(dates,nbcase.T,"--")
plt.xticks(select_dates,dates[select_dates])
plt.title("COVID-19 cumulative cases from Jan 21 to Feb 3 2020")
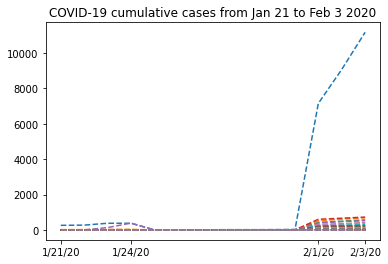
该图从1.24到2.1日具有奇怪的形状,我们提取的location数据包含了地区,国家两列。
接下来将所有来自China的数据分为一行,为此将从nbcase中仅选择位置数据对应于中国的行。接下来使用numpy.sum函数对所有选定的行(axis = 0)求和
china_total = nbcase[locations[:,1]=="China"].sum(axis=0)#numpy axis 详解:https://blog.csdn.net/sky_kkk/article/details/79725646
'''
输出
array([ 247, 288, 556, 817, -22, -22, -15, -10, -9,
-7, -4, 11820, 14410, 17237])
发现了异常出现负值了
'''
Missing data 丢失的数据
nbcases
#输出
array([[ 258, 270, 375, ..., 7153, 9074, 11177],
[ 14, 17, 26, ..., 520, 604, 683],
[ -1, 1, 1, ..., 422, 493, 566],
...,
[ -1, -1, -1, ..., -1, -1, -1],
[ -1, -1, -1, ..., -1, -1, -1],
[ -1, -1, -1, ..., -1, -1, -1]])
'''
当numpy.genfromtxt试图从csv文件中读取丢失的数据时就会以-1来标记,显然我们不能让-1也参与计算
导入numpy.ma模块后,我们将创建一个新数组,直接屏蔽无效值。
'''
from numpy import ma
nbcases_ma = ma.masked_values(nbcases,-1)
nbcases_ma
#输出
masked_array(
data=[[258, 270, 375, ..., 7153, 9074, 11177],
[14, 17, 26, ..., 520, 604, 683],
[--, 1, 1, ..., 422, 493, 566],
...,
[--, --, --, ..., --, --, --],
[--, --, --, ..., --, --, --],
[--, --, --, ..., --, --, --]],
mask=[[False, False, False, ..., False, False, False],
[False, False, False, ..., False, False, False],
[ True, False, False, ..., False, False, False],
...,
[ True, True, True, ..., True, True, True],
[ True, True, True, ..., True, True, True],
[ True, True, True, ..., True, True, True]],
fill_value=-1)
'''
It has three attributes(data,mask and fill_value)
keep in mind that the mask attribute has a True value for elements correspoding
to invalid data(represented by two dashes in the data attribute)
'''
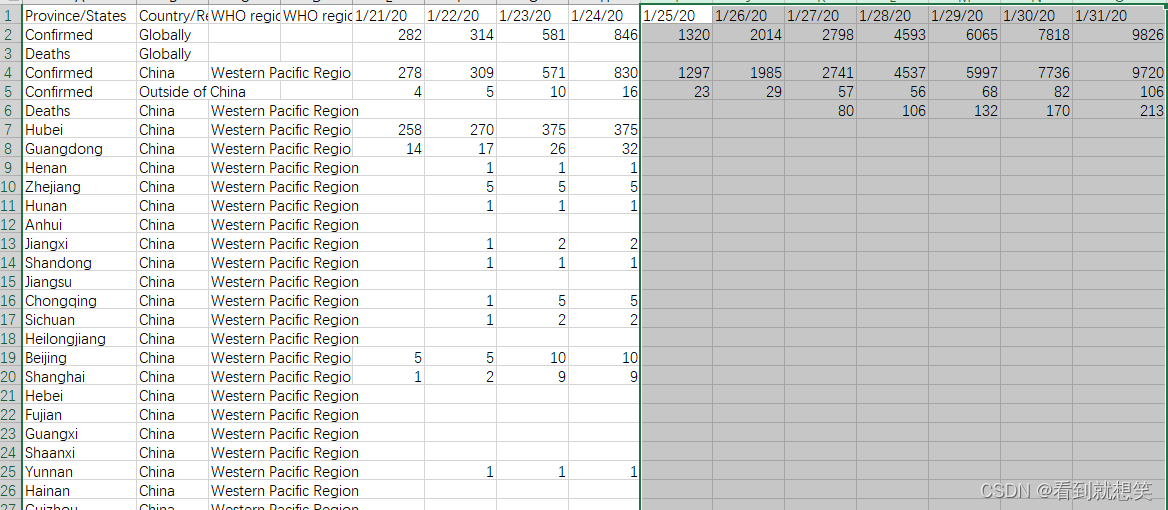
plt.plot(dates, nbcases_ma[1:].T, "--")
plt.xticks(selected_dates, dates[selected_dates])
plt.title("COVID-19 cumulative cases from Jan 21 to Feb 3 2020")
china_masked = nbcases_ma[locations[:, 1] == "China"].sum(axis=0)
china_masked
#输出
masked_array(data=[278, 309, 574, 835, 10, 10, 17, 22, 23, 25, 28, 11821,
14411, 17238],
mask=[False, False, False, False, False, False, False, False,
False, False, False, False, False, False],
fill_value=999999)
china_total = china_masked.data
china_total
#输出
array([ 278, 309, 574, 835, 10, 10, 17, 22, 23,
25, 28, 11821, 14411, 17238])
'''
首先将负值全部处理了,但是(835,10)这两个数字之间明显不符合sum的定义,说明还是有一些数据缺失了,
在中国大陆数据缺失的时候,香港台湾澳门等省和其他“未指定”区域的数据是有效的。
我们可以将这样的数据去除,以便更好地了解数据。
'''
#首先,确定中国大陆地区的位置索引:
china_mask = (
(locations[:, 1] == "China")
& (locations[:, 0] != "Hong Kong")
& (locations[:, 0] != "Taiwan")
& (locations[:, 0] != "Macau")
& (locations[:, 0] != "Unspecified*")
)
china_mask.nonzero()
#输出
(array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16,
17, 18, 19, 20, 21, 22, 23, 25, 26, 27, 28, 29, 31, 33]),)
china_total = nbcases_ma[china_mask].sum(axis=0)
china_total
#输出
masked_array(data=[278, 308, 440, 446, --, --, --, --, --, --, --, 11791,
14380, 17205],
mask=[False, False, False, False, True, True, True, True,
True, True, True, False, False, False],
fill_value=999999)
Fitting Data 拟合数据
#使用.mask来查看masked(invalid entries)的值
china_total.mask
invalid = china_total[china_total.mask]
invalid
#输出
masked_array(data=[--, --, --, --, --, --, --],
mask=[ True, True, True, True, True, True, True],
fill_value=999999,
dtype=int64)
#也可以使用logical negation 来查看valid entries
valid = china_total[~china_total.mask]
valid
#输出
masked_array(data=[278, 308, 440, 446, 11791, 14380, 17205],
mask=[False, False, False, False, False, False, False],
fill_value=999999)
# select the dates for which the data is valid
data[~china_total.mask]
#输出
array(['1/21/20', '1/22/20', '1/23/20', '1/24/20', '2/1/20', '2/2/20',
'2/3/20'], dtype='<U7')
# we can use the numpy.polyfit and numpy.polyval functions to create
# a cubic polynomial(三次多项式) that fits the data as best as possible:
t = np.arange(len(china_total))
params = np.polyfit(t[~china_total.mask], valid, 3)
cubic_fit = np.polyval(params, t)
plt.plot(t, china_total)
plt.plot(t, cubic_fit, "--")

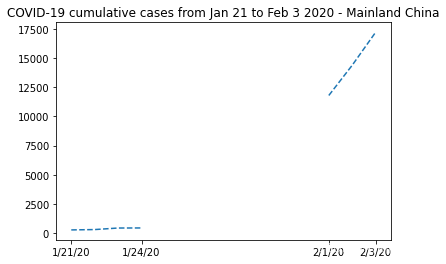
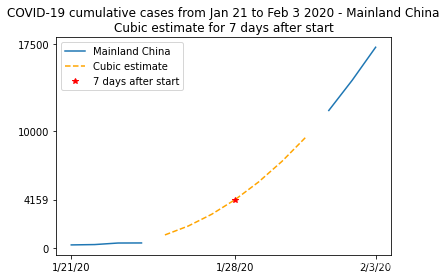
这个plot不太可读,因为线条似乎相互重叠,所以用一个更详细的情节来总结。我们将在可用时绘制真实数据,并显示不可用数据的三次拟合,使用此拟合计算对 2020 年 1 月 28 日(记录开始后 7 天)观察到的病例数的估计:
plt.plot(t, china_total)
plt.plot(t[china_total.mask], cubic_fit[china_total.mask], "--", color="orange")
plt.plot(7, np.polyval(params, 7), "r*")
plt.xticks([0, 7, 13], dates[[0, 7, 13]])
plt.yticks([0, np.polyval(params, 7), 10000, 17500])
plt.legend(["Mainland China", "Cubic estimate", "7 days after start"])
plt.title(
"COVID-19 cumulative cases from Jan 21 to Feb 3 2020 - Mainland China\n"
"Cubic estimate for 7 days after start"
)
参考网站:https://numpy.org/numpy-tutorials/content/tutorial-ma.html#
https://www.kaggle.com/atilamadai/covid19