目录
针对单一输入的sequence,在UniRef100数据库中进行搜索相似的序列
sequence对应的输出文件:(格式是.a3m)下图只显示了一部分
?
?1、安装和下载一些包
import os
import sys
# IPython的交互式shell
from IPython.utils import io
from google.colab import files
if not os.path.isdir("RoseTTAFold"):
# 抑制函数命令行的输出
with io.capture_output() as captured:
# 下载额外的函数colabfold.py,-qnc会覆盖之前下载的文件
%shell wget -qnc https://raw.githubusercontent.com/sokrypton/ColabFold/main/beta/colabfold.py
# 下载训练好的模型
# 下载官方提供的代码,也可以直接下载
%shell git clone https://github.com/RosettaCommons/RoseTTAFold.git
# 下载补丁
%shell wget -qnc https://raw.githubusercontent.com/sokrypton/ColabFold/main/beta/RoseTTAFold__network__Refine_module.patch
# 安装补丁
# 参考:https://www.jb51.net/article/98185.htm
%shell patch -u RoseTTAFold/network/Refine_module.py -i RoseTTAFold__network__Refine_module.patch
# 下载模型的权重参数,-qnc会覆盖之前下载的文件
%shell wget -qnc https://files.ipd.uw.edu/pub/RoseTTAFold/weights.tar.gz
# 解压
# 参考:https://www.cnblogs.com/zl520/p/12888092.html?ivk_sa=1024320u
%shell tar -xf weights.tar.gz
# 删除压缩文件
%shell rm weights.tar.gz
# download scwrl4:主要用于添加侧翼序列
# http://dunbrack.fccc.edu/SCWRL3.php
%shell wget -qnc https://files.ipd.uw.edu/krypton/TrRosetta/scwrl4.zip
%shell unzip -qqo scwrl4.zip
%shell rm scwrl4.zip # 也可删除
# 安装一些包
# 离线GPU版本
%shell pip install -q dgl-cu113 -f https://data.dgl.ai/wheels/repo.html
# 下载torch-geometric和所需的torch-scatter\torch-sparse
# 好像在windows里安装比较麻烦
%shell pip install -q torch-scatter -f https://pytorch-geometric.com/whl/torch-1.10.0+cu113.html
%shell pip install -q torch-sparse -f https://pytorch-geometric.com/whl/torch-1.10.0+cu113.html
%shell pip install -q torch-geometric
# py3Dmol用于绘制蛋白质的三维结构的
%shell pip install -q py3Dmol
with io.capture_output() as captured:
# 添加环境
sys.path.append('/content/RoseTTAFold/network')
# 预测端到端模型
import predict_e2e
# parse_a3m:主要是A3M文件,并将字母转换为0~20之间的数字
from parsers import parse_a3m2、所需的一些函数
# colabfold里面包含很多函数
import colabfold as cf
import py3Dmol
# subprocess 模块允许我们启动一个新进程
import subprocess
import numpy as np
import matplotlib.pyplot as plt
def get_bfactor(pdb_filename):
bfac = []
# 读取pdb文件,当是ATOM标签时,将对应的数据添加到bfac中
# 具体还不知道获取的是什么信息,后续再补?????
for line in open(pdb_filename,"r"):
if line[:4] == "ATOM":
bfac.append(float(line[60:66]))
return np.array(bfac)
# 接收get_factor中获取的bfac
def set_bfactor(pdb_filename, bfac):
I = open(pdb_filename,"r").readlines()
# 在同一个pdb文件中进行操作,根据bfac进行替换
O = open(pdb_filename,"w")
for line in I:
if line[0:6] == "ATOM ":
seq_id = int(line[22:26].strip()) - 1
O.write(f"{line[:60]}{bfac[seq_id]:6.2f}{line[66:]}")
O.close()
def do_scwrl(inputs, outputs, exe="./scwrl4/Scwrl4"):
"""
inputs
outputs
"""
subprocess.run([exe,"-i",inputs,"-o",outputs,"-h"],stdout=subprocess.DEVNULL, stderr=subprocess.DEVNULL)
bfact = get_bfactor(inputs)
set_bfactor(outputs, bfact)
return bfact3、序列输入
# 输入的序列
sequence = "PIAQIHILEGRSDEQKETLIREVSEAISRSLDAPLTSVRVIITEMAKGHFGIGGELASK" #@param {type:"string"}
# 剔除字符串中所有的非字母情况并保证最终的输出为大写字母
sequence = sequence.translate(str.maketrans('', '', ' \n\t')).upper()
print(f"{sequence}")
jobname = "test"
# 函数get_hash根据输入序列获取hash码
import hashlib
def get_hash(x):
return hashlib.sha1(x.encode()).hexdigest()
jobname = jobname+"_" + get_hash(sequence)[:6]
print(f'{jobname}')针对单一输入的sequence,在UniRef100数据库中进行搜索相似的序列
# 搜索基因数据库
msa_method = "mmseqs2" # ["mmseqs2","single_sequence","custom_a3m"]
# `mmseqs2` - FAST method from [ColabFold](https://github.com/sokrypton/ColabFold)
# `single_sequence` 单序列输入,但是这种不推荐使用
# `custom_a3m` 上传定制的a3m文件
# 临时目录
prefix = get_hash(sequence)
# 创建文件夹
os.makedirs('tmp', exist_ok=True)
prefix = os.path.join('tmp',prefix[:5])
os.makedirs(jobname, exist_ok=True)
# 选择采用的类型
# msa.a3m是表示input序列,最开始在uniref数据库中搜索到的文件
if msa_method == "mmseqs2":
a3m_lines = cf.run_mmseqs2(sequence, prefix, filter=True)
with open(f"{jobname}/msa.a3m","w") as a3m:
a3m.write(a3m_lines)
elif msa_method == "single_sequence":
with open(f"{jobname}/msa.a3m","w") as a3m:
# 单序列的输出格式
a3m.write(f">{jobname}\n{sequence}\n")
elif msa_method == "custom_a3m":
print("upload custom a3m")
# 这里需要自己手动上传,或者你直接加载指定的a3m文件也可以
msa_dict = files.upload()
lines = msa_dict[list(msa_dict.keys())[0]].decode().splitlines()
a3m_lines = []
for line in lines:
line = line.replace("\x00","")
if len(line) > 0 and not line.startswith('#'):
a3m_lines.append(line)
with open(f"{jobname}/msa.a3m","w") as a3m:
a3m.write("\n".join(a3m_lines))
# 对搜索到的a3m文件进行解析,保证只有大写字母,没有别的符号
msa_all = parse_a3m(f"{jobname}/msa.a3m")
# 去重操作
msa_arr = np.unique(msa_all,axis=0)
# 总共在uniref中搜索到多少条序列
total_msa_size = len(msa_arr)
print("总计搜索到:",total_msa_size)
if msa_method == "mmseqs2":
print(f'\n{total_msa_size} Sequences Found in Total (after filtering)\n')
else:
print(f'\n{total_msa_size} Sequences Found in Total\n')
if total_msa_size > 1:
plt.figure(figsize=(8,5),dpi=100)
plt.title("Sequence coverage")
# 按行求均值
print("msa_all:\n",msa_all)
seqid = (msa_all[0] == msa_arr).mean(-1)
print("seqid:\n",seqid)
# 从小到大进行排序,并返回索引值
seqid_sort = seqid.argsort()
non_gaps = (msa_arr != 20).astype(float)
non_gaps[non_gaps == 0] = np.nan
print("non_gaps:\n",non_gaps)
plt.imshow(non_gaps[seqid_sort]*seqid[seqid_sort,None],
interpolation='nearest', aspect='auto',
cmap="rainbow_r", vmin=0, vmax=1, origin='lower',
extent=(0, msa_arr.shape[1], 0, msa_arr.shape[0]))
# 图中红色部分表示对应每个残基上的值不等于20的数量
plt.plot((msa_arr != 20).sum(0), color='red')
plt.xlim(0,msa_arr.shape[1])
plt.ylim(0,msa_arr.shape[0])
plt.colorbar(label="Sequence identity to query",)
plt.xlabel("Positions")
plt.ylabel("Sequences")
plt.savefig(f"{jobname}/msa_coverage.png", bbox_inches = 'tight')
plt.show()?输出结果:
总计搜索到: 7377
7377 Sequences Found in Total (after filtering)
msa_all:
[[14 9 0 ... 0 15 11]
[14 12 9 ... 0 15 20]
[14 10 19 ... 15 15 6]
...
[14 10 19 ... 1 15 11]
[20 9 19 ... 20 20 20]
[20 20 20 ... 19 20 20]]
seqid:
[0.23728814 0.27118644 0.33898305 ... 0.13559322 0.25423729 0.13559322]
non_gaps:
[[ 1. 1. 1. ... nan nan nan]
[ 1. 1. 1. ... nan nan nan]
[ 1. 1. 1. ... 1. 1. 1.]
...
[nan nan nan ... 1. 1. nan]
[nan nan nan ... nan nan nan]
[nan nan nan ... 1. 1. 1.]] ?
?
?上图的横坐标表示序列每个残基的位置,本例中是59,纵坐标表示针对单序列输入搜索到的序列数量,这里是7377条;图中红色线条表示每个残基位置上值不等于20的数量;右侧表示搜索到的序列相对query的置信度,颜色越深相似度越高;
sequence对应的输出文件:(格式是.a3m)下图只显示了一部分

?4、预测模块
这里只是采用了RoseTTAFold的一个end-2-end版本,采用官方提供训练好的模型,并根据在uniref100中针对单条序列得到msa.a3m进行预测,得到一个pred.pdb,然后再将这个pred文件通过scwrl4工具进行打包,然后输出对应的plddt;
注:
SCWRL4 是一个用于预测给定蛋白质骨架的侧链构象的程序。
它作为控制台或命令行应用程序运行,并使用配置文件进行参数化。
数据在通过命令行参数指定的文件中传输。如何快速使用?
============
Quick start:
============
The following command will predict side-chain conformations built around
a protein backbone:
Scwrl4 -i abcd_IN.pdb -o abcd_OUT.pdb
This command will read the backbone coordinates from abcd_IN.pdb and write the
predicted side-chain coordinates into the file abcd_OUT.pdb. A test input file
called abcd_IN.pdb resides in the SCWRL4 directory.
You are free to specify relative or absolute paths to the input and output
files. If file locations include spaces, you should place the filenames in
double quotes.
For more usage details please refer to Readme.txt file in SCWRL4 directory or
just type Scwrl4.exe without input arguments.
Georgii Krivov, Maxim Shapovalov and Roland Dunbrack
Fox Chase Cancer Center
333 Cottman Ave
Philadelphia PA 19111
http://dunbrack.fccc.edu/scwrl4# 为主链运行 RoseTTAFold,为侧链预测运行Scrwl4
# load model
if "rosettafold" not in dir():
# 加载训练好的权重参数
rosettafold = predict_e2e.Predictor(model_dir="weights")
# make prediction using model
rosettafold.predict(f"{jobname}/msa.a3m",f"{jobname}/pred")
# 使用Scwrl4打包侧链信息
plddt = do_scwrl(f"{jobname}/pred.pdb",f"{jobname}/pred.scwrl.pdb")
print(plddt)
print(f"Predicted LDDT: {plddt.mean()}")
plt.figure(figsize=(8,5),dpi=100)
plt.plot(plddt)
plt.xlabel("positions")
plt.ylabel("plddt")
plt.ylim(0,1)
plt.savefig(f"{jobname}/plddt.png", bbox_inches = 'tight')
plt.show()
输出结果:
SE(3) iteration 0 [0.835]
SE(3) iteration 1 [0.8633]
SE(3) iteration 2 [0.841]
SE(3) iteration 3 [0.851]
SE(3) iteration 4 [0.854]
SE(3) iteration 5 [0.8984]
SE(3) iteration 6 [0.915]
SE(3) iteration 7 [0.928]
SE(3) iteration 8 [0.939]
SE(3) iteration 9 [0.943]
SE(3) iteration 10 [0.944]
SE(3) iteration 11 [0.943]
SE(3) iteration 12 [0.9424]
SE(3) iteration 13 [0.9424]
SE(3) iteration 14 [0.942]
SE(3) iteration 15 [0.942]
SE(3) iteration 16 [0.942]
SE(3) iteration 17 [0.942]
SE(3) iteration 18 [0.9414]
SE(3) iteration 19 [0.9414]
SE(3) iteration 20 [0.9414]
SE(3) iteration 21 [0.9414]
SE(3) iteration 0 [0.7915]
SE(3) iteration 1 [0.8657]
SE(3) iteration 2 [0.921]
SE(3) iteration 3 [0.9355]
SE(3) iteration 4 [0.942]
SE(3) iteration 5 [0.944]
SE(3) iteration 6 [0.9443]
SE(3) iteration 7 [0.9443]
SE(3) iteration 8 [0.944]
SE(3) iteration 9 [0.944]
SE(3) iteration 10 [0.944]
SE(3) iteration 11 [0.9434]
SE(3) iteration 12 [0.9434]
SE(3) iteration 13 [0.9434]
SE(3) iteration 14 [0.9434]
SE(3) iteration 15 [0.9424]
SE(3) iteration 16 [0.9424]
SE(3) iteration 17 [0.942]
[0.98 0.98 0.98 0.98 0.96 0.96 0.96 0.96 1. 1. 1. 1. 0.99 0.99
0.99 0.99 1. 1. 1. 1. 0.97 0.97 0.97 0.97 0.97 0.97 0.97 0.97
0.94 0.94 0.94 0.94 0.93 0.93 0.93 0.93 0.91 0.91 0.91 0.91 0.96 0.96
0.96 0.96 0.97 0.97 0.97 0.97 0.98 0.98 0.98 0.98 0.97 0.97 0.97 0.97
0.95 0.95 0.95 0.95 0.96 0.96 0.96 0.96 0.97 0.97 0.97 0.97 0.96 0.96
0.96 0.96 0.97 0.97 0.97 0.97 0.96 0.96 0.96 0.96 0.98 0.98 0.98 0.98
0.97 0.97 0.97 0.97 0.99 0.99 0.99 0.99 0.97 0.97 0.97 0.97 0.99 0.99
0.99 0.99 0.98 0.98 0.98 0.98 0.98 0.98 0.98 0.98 0.96 0.96 0.96 0.96
0.92 0.92 0.92 0.92 0.92 0.92 0.92 0.92 0.91 0.91 0.91 0.91 0.9 0.9
0.9 0.9 0.94 0.94 0.94 0.94 0.95 0.95 0.95 0.95 0.93 0.93 0.93 0.93
0.93 0.93 0.93 0.93 0.97 0.97 0.97 0.97 0.98 0.98 0.98 0.98 1. 1.
1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1.
1. 1. 1. 1. 0.99 0.99 0.99 0.99 0.95 0.95 0.95 0.95 0.93 0.93
0.93 0.93 0.96 0.96 0.96 0.96 0.92 0.92 0.92 0.92 0.9 0.9 0.9 0.9
0.94 0.94 0.94 0.94 0.89 0.89 0.89 0.89 0.83 0.83 0.83 0.83 0.77 0.77
0.77 0.77 0.82 0.82 0.82 0.82 0.85 0.85 0.85 0.85 0.88 0.88 0.88 0.88
0.88 0.88 0.88 0.88 0.9 0.9 0.9 0.9 0.82 0.82 0.82 0.82]
Predicted LDDT: 0.9440677966101694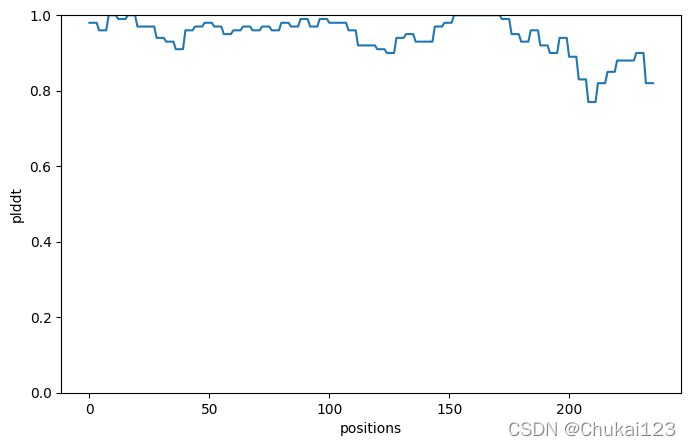
?5、展示3D结构
# 展示3D结构
color = "rainbow" # 有三种形式 ["chain", "lDDT", "rainbow"]
show_sidechains = True #{type:"boolean"}
show_mainchains = False #{type:"boolean"}
def read_pdb_renum(pdb_filename, Ls=None):
if Ls is not None:
L_init = 0
new_chain = {}
for L,c in zip(Ls, alphabet_list):
new_chain.update({i:c for i in range(L_init,L_init+L)})
L_init += L
n,pdb_out = 1,[]
resnum_,chain_ = 1,"A"
for line in open(pdb_filename,"r"):
if line[:4] == "ATOM":
chain = line[21:22]
resnum = int(line[22:22+5])
if resnum != resnum_ or chain != chain_:
resnum_,chain_ = resnum,chain
n += 1
if Ls is None: pdb_out.append("%s%4i%s" % (line[:22],n,line[26:]))
else: pdb_out.append("%s%s%4i%s" % (line[:21],new_chain[n-1],n,line[26:]))
return "".join(pdb_out)
def show_pdb(pred_output_path, show_sidechains=False, show_mainchains=False,
color="lDDT", chains=None, Ls=None, vmin=50, vmax=90,
color_HP=False, size=(800,480)):
if chains is None:
chains = 1 if Ls is None else len(Ls)
view = py3Dmol.view(js='https://3dmol.org/build/3Dmol.js', width=size[0], height=size[1])
view.addModel(read_pdb_renum(pred_output_path, Ls),'pdb')
if color == "lDDT":
view.setStyle({'cartoon': {'colorscheme': {'prop':'b','gradient': 'roygb','min':vmin,'max':vmax}}})
elif color == "rainbow":
view.setStyle({'cartoon': {'color':'spectrum'}})
elif color == "chain":
for n,chain,color in zip(range(chains),alphabet_list,pymol_color_list):
view.setStyle({'chain':chain},{'cartoon': {'color':color}})
if show_sidechains:
BB = ['C','O','N']
HP = ["ALA","GLY","VAL","ILE","LEU","PHE","MET","PRO","TRP","CYS","TYR"]
if color_HP:
view.addStyle({'and':[{'resn':HP},{'atom':BB,'invert':True}]},
{'stick':{'colorscheme':"yellowCarbon",'radius':0.3}})
view.addStyle({'and':[{'resn':HP,'invert':True},{'atom':BB,'invert':True}]},
{'stick':{'colorscheme':"whiteCarbon",'radius':0.3}})
view.addStyle({'and':[{'resn':"GLY"},{'atom':'CA'}]},
{'sphere':{'colorscheme':"yellowCarbon",'radius':0.3}})
view.addStyle({'and':[{'resn':"PRO"},{'atom':['C','O'],'invert':True}]},
{'stick':{'colorscheme':"yellowCarbon",'radius':0.3}})
else:
view.addStyle({'and':[{'resn':["GLY","PRO"],'invert':True},{'atom':BB,'invert':True}]},
{'stick':{'colorscheme':f"WhiteCarbon",'radius':0.3}})
view.addStyle({'and':[{'resn':"GLY"},{'atom':'CA'}]},
{'sphere':{'colorscheme':f"WhiteCarbon",'radius':0.3}})
view.addStyle({'and':[{'resn':"PRO"},{'atom':['C','O'],'invert':True}]},
{'stick':{'colorscheme':f"WhiteCarbon",'radius':0.3}})
if show_mainchains:
BB = ['C','O','N','CA']
view.addStyle({'atom':BB},{'stick':{'colorscheme':f"WhiteCarbon",'radius':0.3}})
view.zoomTo()
return view
show_pdb(f"{jobname}/pred.scwrl.pdb", show_sidechains, show_mainchains, color, chains=1, vmin=0.5, vmax=0.9).show()
def plot_plddt_legend(dpi=100):
thresh = ['plDDT:','Very low (<50)','Low (60)','OK (70)','Confident (80)','Very high (>90)']
plt.figure(figsize=(1,0.1),dpi=dpi)
########################################
for c in ["#FFFFFF","#FF0000","#FFFF00","#00FF00","#00FFFF","#0000FF"]:
plt.bar(0, 0, color=c)
plt.legend(thresh, frameon=False,
loc='center', ncol=6,
handletextpad=1,
columnspacing=1,
markerscale=0.5,)
plt.axis(False)
return plt
if color == "lDDT":
plot_plddt_legend().show() 只显示主链:
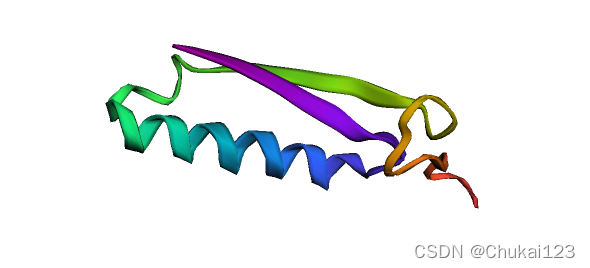
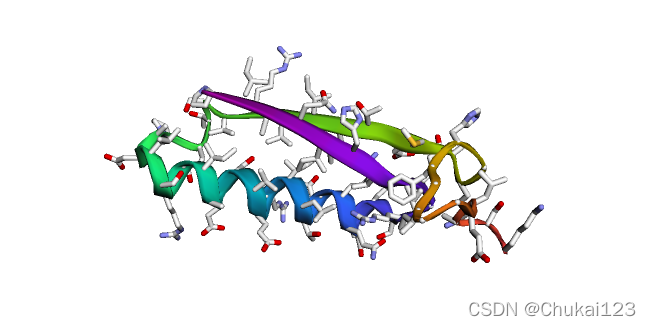
?显示对应的侧链:
?6、下载对应的预测文件
# 下载预测的文件
# 添加配置文件
settings_path = f"{jobname}/settings.txt"
with open(settings_path, "w") as text_file:
text_file.write(f"method=RoseTTAFold\n")
text_file.write(f"sequence={sequence}\n")
text_file.write(f"msa_method={msa_method}\n")
text_file.write(f"use_templates=False\n")
!zip -q -r {jobname}.zip {jobname}
files.download(f'{jobname}.zip')参考文献:
- Mirdita M, Schütze K, Moriwaki Y, Heo L, Ovchinnikov S and Steinegger M. ColabFold - Making protein folding accessible to all.
bioRxiv (2021) doi:?10.1101/2021.08.15.456425 - If you’re using?AlphaFold, please also cite:
Jumper et al. "Highly accurate protein structure prediction with AlphaFold."
Nature (2021) doi:?10.1038/s41586-021-03819-2 - If you’re using?AlphaFold-multimer, please also cite:
Evans et al. "Protein complex prediction with AlphaFold-Multimer."
biorxiv (2021) doi:?10.1101/2021.10.04.463034v1 - If you are using?RoseTTAFold, please also cite:
Minkyung et al. "Accurate prediction of protein structures and interactions using a three-track neural network."
Science (2021) doi:?10.1126/science.abj8754
参考GitHub:
?https://github.com/chukai123/ColabFold![]() https://github.com/chukai123/ColabFold
https://github.com/chukai123/ColabFold