《BERT6mA: prediction of DNA N6-methyladenine site using deep learning-based approaches》
- Sho Tsukiyama , Md Mehedi Hasan , Hong-Wen Deng and Hiroyuki Kurata
- 日本 九州理工学院生物科学和生物信息学系
- 2022 年 2 月 28 日
- Briefings in Bioinformatics, 2022, 1–15
- https://doi.org/10.1093/bib/bbac053
摘要
N6-甲基腺嘌呤(6mA)在DNA复制、DNA修复、转录和基因表达调控中发挥重要作用。几种实验方法被用来鉴定DNA修饰。然而,这些实验方法既昂贵又耗时。为了检测6mA并弥补实验方法的这些不足,我们提出了一种新的深度学习方法,称为BERT6mA。为了将BERT6mA与其他深度学习方法进行比较,我们使用了包括11个物种在内的基准数据集。在独立测试中,BERT6mA在八个物种中表现出最高的AUC。此外,BERT6mA在一些样本量较小的物种中表现不佳,为了克服这个问题,对BERT6mA进行了两个物种之间的预训练和微调。对特定物种的预训练和微调模型表现出比其他模型更高的性能,即使对于样本量较小的物种也是如此。除了预测之外,我们还分析了BERT6mA产生的注意权重,以揭示BERT6mA模型如何提取负责6mA预测的关键特征。为了促进生物科学的发展,BERT6mA在线网络服务器及其源代码可以在https://github.com/kuratahiroyuki/BERT6mA.git获得。
关键词: 6mA修改预测、BERT、word2vec、GRU、LSTM、CNN
Introduction
N6-甲基腺嘌呤(6mA)是一种重要的表观遗传修饰,在DNA复制、DNA修复、转录和基因表达调控中发挥重要作用。例如,在DNA错配修复中,新合成的链和模板通过甲基化或不通过甲基化来区分,而甲基化作为一种标记,在限制修饰系统中区分自我DNA和外来DNA。此外,之前的一项研究报告称,6mA与癌症等多种疾病有关。因此,6mA的鉴定对于理解表观遗传修饰过程和揭示与疾病相关的表观遗传调控至关重要。
高通量实验方法,包括单分子实时(SMRT)测序技术,甲基DNA免疫沉淀和液相色谱串联质谱,已被用于识别DNA甲基化位点。它们提供了一种以单核苷酸分辨率检测DNA甲基化的有效方法。然而,它们只覆盖了基因组DNA的一部分,没有在整个基因组中检测到6mA位点,其中一些存在测序质量和信号音调比问题。此外,实验方法既昂贵又耗时。因此,开发计算预测模型以降低实验成本并弥补实验方法的不足是有价值的。
已经开发了几种基于机器学习和深度学习的模型来预测DNA 6mA修饰位点。最近,Lv等人提出了iDNA MS,使用随机森林(RF)模型,结合单核苷酸二元编码(MNBE)和核苷酸化学性质和核苷酸频率(NCPNF)编码方法,预测了11个物种的6mA位点。这样的机器学习方法采用基于成分和基于化学性质的编码方法预测4mC和5mC的修饰位点。
另一方面,通过使用最新的深度学习,上述方法仍有待改进。因此,有必要开发一种有效的DNA预测器,该预测器可以学习多个物种中隐藏在6MA和非6MA之间的特征,从而成功地应用于识别特征模式。除了这些编码方法外,还使用了自然语言处理中的嵌入技术。尤其是,word2vec被认为是最好的嵌入方法之一,用于生物信息学分类中的各种分类和预测。
基于深度学习的模型通过从输入序列中捕捉与6mA显著相关的特征,实现了稳健而准确的预测。Wahab等人利用一维卷积神经网络(1D-CNN)记录了水稻和小家鼠体内6mA位点,并在两个物种中都获得了ROC曲线下面积(AUC)大于0.9。基于长期和短期记忆(LSTM)的模型和基于门控递归单元(GRU)的模型通过提取有关记忆机制中核苷酸顺序的信息,表现出稳定的性能。最近,Li等人推出了一种CNN和LSTM的组合模型,其性能优于之前的最先进模型。此外,transformers 双向编码器表示(BERT)是最强大的预测模型之一,它可以实现更快的推理,因为BERT不需要在基于递归神经网络(RNN)的模型(如LSTM和GRU)上执行的连续递归计算。Devlin等人首先提出了一个BERT模型,并对其模型进行了预训练和微调训练,以通过使用较少的数据构建具有较高泛化性能的模型。Zhang等人使用基于BERT的模型,在大肠杆菌和智人中识别出了高性能的6mA位点。Yu等人开发了iDNA ABT,使用BERT和自适应嵌入方法预测6MA,并将其与之前的模型(包括SNNRice6mA和DeepT orrent)进行了比较。
在这项研究中,我们提出了BERT6mA(BERT with word2vec),这是一种新的深度学习方法,可以识别6mA位点,如图1所示。我们将包括DNA序列组成、核苷酸化学性质和word2vec在内的七种编码方法与八种深度学习模型相结合,以比较Lv等人的iDNA MS和Yu等人的iDNA ABT的性能。此外,我们还产生了两种新的编码方案,即背景核苷酸化学性质和核苷酸频率(C-NCPNF)和背景单核苷酸二元编码(C-MNBE)。对于许多物种,BERT6mA优于iDNA MS、iDNAABT和其他基于深度学习的模型,但对于6mA数据较少的物种,BERT6mA表现不佳。为了用较少的6mA数据构建BERT,我们采用了预训练和微调方法。除了预测之外,我们还挑战了深度学习的黑匣子问题。我们分析了BERT产生的注意权重,以确定与6mA修饰密切相关的一些核苷酸分布。BERT6mA不仅可用于预测6mA,还可用于揭示BERT6mA区分6mA和非6mA的机制。据我们所知,这是首次采用多种深度学习算法构建6mA预测学习框架的研究,这可能有助于DNA表观遗传学研究。

本研究分析的总体框架。我们的工作包括特征编码、基于深度学习的预测、评估、跨物种验证、预训练和微调、6mA左右核苷酸的观察、注意力权重分析和web服务器实现。
Materials and methods
Datasets
为了在不同数据量的多物种中比较深度学习和机器学习的性能,基准数据集取自iDNA MS最近发表的文章。6mA位点的数据集来自多个已发表的参考文献和数据库,包括MethSMRT数据库、MDR数据库、GEO数据库和NCBI基因组数据库。它们包含了11个物种的6mA和非6mA数据,包括拟南芥(31873 6mA和非6mA)、秀丽隐杆线虫(7961 6mA和非6mA)、木麻黄(6066 6mA和非6mA)、黑腹果蝇(11191 6mA和非6mA)、弗雷加菌(3102 6mA和非6mA)、智人(18335 6mA和非6mA),中国蔷薇(599 6mAs和非6mAs)、酿酒酵母(3786 6mAs和非6mAs)、嗜热热菌(107600 6mAs和非6mAs)、链霉菌SUP5–1(3379 6mAs和非6mAs)和Xoc。BLS256(172156MA和非6MA)。所用11个物种的6mA样本主要通过SMRT以单核苷酸分辨率进行测量。序列窗口的长度设置为41 bp,这在之前的6mA预测研究中显示了最高的性能。甲基化腺嘌呤在6mA样本中位于中心位置,而非6mA样本中的甲基化腺嘌呤未经实验证实。每个物种的数据集按1:1的比例分为训练数据和独立测试数据。策划的数据集可以从Lv等人开发的web应用程序下载。
Feature encoding methods
特征编码与预测模型的性能有关。我们通过五种单一编码方法(NCPNF、MNBE、C-NCPNF、C-MNBE和word2vec)和两种组合编码方法将DNA序列转化为特征矩阵。
Nucleotide chemical property and nucleotide frequency
NCPNF特征矩阵由每个核苷酸的化学性质和密度生成。DNA序列中第i个核苷酸的化学性质是
由三种不同特征的组合表示(xi,yi,zi), xi, yi 和 zi是环结构、氢键和化学官能团的组合来表示。根据环结构,核苷酸分为嘌呤和嘧啶。就氢键而言,它们是根据形成强氢键还是弱氢键来分类的。就化学功能而言,它们分为氨基和酮基。根据上述分类,每个特征由以下各项表示:

其中A、T、G和C的化学特征向量分别对应于(1,1,1)、(0,1,0)、(1,0,0)和(0,0,1),第i个核苷酸的密度通过以下公式计算:

其中,L、
N
J
N_J
NJ?和q分别是DNA序列的长度、序列中第i个前缀字符串的长度和相关核苷酸的长度。通过如下排列化学性质和密度,生成第i个核苷酸的特征向量:
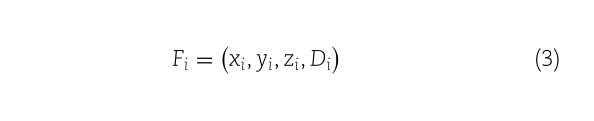
最后,将DNA序列中的特征向量串联起来,每个DNA序列由41×4矩阵表示.
单核苷酸二进制编码
MNBE将给定序列的确切核苷酸位置编码为二元载体,如下所示:
将DNA序列中每个核苷酸的载体连接起来,每个DNA序列由4 1×4矩阵表示。
语境性NCPNF与语境性MNBE
此外,我们生成了两种新的编码方法C-NCPNF和C-MNBE来预测6MA。如图S1所示,我们将基于NCPNF和MNBE的特征矩阵的连续指节节点向量排列成一行,并将它们连接起来,以创建基于NCPNF和MNBE的上下文特征矩阵。因此,矩阵中的每个载体都包含连续多个核苷酸的信息。在本研究中,由于Cekis设置为25,序列窗口由17×100矩阵表示。

图S1。使用上下文-NCPNF和- mnbe (C- NCPNF和C- mnbe)生成特征矩阵。基于C-NCPNF和c - mnbe的特征矩阵是通过连接基于NCPNF和基于mnbe的特征矩阵的k-mer向量产生的
Word2vec
Word2vec是自然语言处理领域中功能强大的嵌入方法之一。它通过学习单词上下文将不同的语言规则和模式编码成分布式表示。有两种学习单词上下文的方法:连续单词袋模型(CBOW)和连续跳过语法模型(Skip-Gram)。在学习过程中,CBOW根据上下文预测当前单词,而Skip Gram根据当前单词预测上下文。我们使用Skip-Gram是因为Skip-Gram比CBOW表现出更好的性能。
在使用word2vec对DNA序列进行编码时,连续的4-mers被视为单词,如图S2所示,word2vec模型根据所有物种的训练数据进行训练,每4-mer产生100维向量。特征矩阵通过连接它们生成,因此每个核苷酸序列窗口由38×100矩阵表示。

图S2。由word2vec编码。利用word2vec模型将DNA序列编码到特征矩阵中。连续的4-mers DNA序列被转换成100维的载体。
Deep learning models
构建了八个深度学习模型,包括LSTM、双向LSTM(Bi LSTM)、GRU、双向GRU(Bi GRU)、BERT、1D-CNN、带Bi LSTM的BERT和带Bi LSTM的1D-CNN。我们使用python包的pytorch来构建深度学习模型。每个深度学习架构如下所述。有关网络结构的所有参数汇总在表S1中。
 训练参数和网络架构
训练参数和网络架构
LSTM and Bi-LSTM
RNN对于预测特征之间具有相互依赖性的数据(如时间序列数据)非常有用。然而,由于梯度消失和爆炸,RNN无法学习长期依赖关系。LSTM引入了门结构和存储单元来解决这个问题。LSTM的优势促成了各种分类和预测。Bi-LSTM在两个方向上扩展LSTM单元。如图S3所示,在每一步将涉及一个或多个核苷酸的特征向量输入到LSTM单元。最后一步来自LSTM单元的输出向量应用于完全连接的层,以生成最终输出。在Bi LSTM中,在正向和反向的最后一步,LSTM单元的输出被连接起来,最终输出是通过将连接的向量应用到完全连接的层来生成的(图S4)。为了获得一个介于0和1之间的值作为最终输出,在完全连接的层上使用sigmoid函数作为激活函数。在本研究中,LSTM单元的隐藏大小设置为128。

图S3。LSTM和GRU模型的结构。在每一步中,特征矩阵中的每个向量都被输入到LSTM或GRU单元。最后的输出是通过将LSTM单元最后一步的输出应用到全连接层得到的。

图S4。Bi-LSTM和Bi-GRU模型的结构。在每一步中,特征矩阵中的每个向量都被输入到LSTM或GRU单元。在最后一步,正向和反向LSTM单元的输出被连接起来,并应用到一个完全连接的层,以产生最终的输出。
来自 transformers的双向编码器表示
近年来,BERT因其最先进的技术而备受关注,该技术适用于各个领域的广泛任务。它由Vaswani等人在2017年开发的 transformers的多个编码器层组成,不仅学习单向依赖性,还学习双向依赖性。编码器部分包括多头注意和前馈网络。如图S5所示,将特征矩阵输入到BERT中。来自BERT网络的输出被连接起来,并通过一个sigmoid函数传输到一个完全连接的层,以产生最终输出。与之前的研究一样,层次数、注意头数和隐藏大小分别设置为3、4和100。

图S5。BERT模型的结构。将特征矩阵输入BERT网络。BERT的输出向量被连接起来,然后应用到一个完全连接的层上。
One-dimensional convolutional neural network
CNN主要由两种类型的层组成:卷积层和池层。在卷积层,通过使用滤波器来提取预测所需的重要特征。池层提供了针对模式修改的健壮预测,并通过压缩信息来抑制过度拟合。我们使用Wahab等人开发的CNN模型。该CNN由两个卷积层和两个最大池层组成(图S6)。使用ReLU函数的每个卷积层的输出被发送到最大池层。每个最大池层的输出以0.4的dropout。.第二个最大池层的输出被展平并发送到完全连接的层以生成最终输出。一个S形函数被用作激活函数。以与Wahab等人相同的方式,卷积层中的滤波器数量、滤波器大小和步幅分别设置为32、5和1,最大池层的池大小和步幅分别为2和2。

图S6。一维cnn模型的结构。
特征矩阵由两个卷积层和一个全连通层处理。卷积层的输出向量应用于最大池化层和dropout层。最后的输出是通过连接来自1D-CNN的输出并将它们应用到一个完全连接的层来生成的。
Hybrid models
我们构建了混合模型,包括BERT和Bi LSTM(图S7)和带有Bi-LSTM的1D-CNN(图S8)。在这两种混合模型中,特征矩阵分别由BERT和1D-CNN=处理。然后,在每一步,分别将来自BERT中的第三编码器层和1D-CNN中的第二最大池层的中间向量输入Bi-LSTM单元。在最后一步,来自正向和反向LSTM单元的输出被串联,并且组合的输出被应用到具有sigmoid函数的完全连接层,以产生最终输出。BERT与Bi LSTM的混合模型是在之前的研究中提出的,用于预测苦味肽。为了将BERT和Bi LSTM与BERT和Bi LSTM进行比较,将BERT和Bi LSTM的参数(包括层数、注意头和隐藏大小)设置为与BERT和Bi LSTM相同的值。带有BiLSTM的1D-CNN的参数设置为与1D-CNN和Bi LSTM相同的值。

图S7。基于Bi-LSTM模型的BERT结构。
该网络由BERT、Bi-LSTM和全连接层组成。BERT的输出向量在每一步输入到Bi-LSTM单元。
Training of deep learning models
我们以与Lv等人相同的方式评估了深度学习方法的预测性能。对训练数据集进行五倍交叉验证,并对训练后的模型进行独立测试。在5倍交叉验证中,训练数据被分为五个子集。然后,使用四个子集作为训练模型;另一个用于验证。优化由Adam优化器执行,学习率为1.0 × 10?5.将小批量设置为128,并通过二元交叉熵损失函数计算损失。为了防止过度学习,当验证数据中的最小损失连续20个epoch未更新时,培训过程终止。培训的所有参数汇总在表S1中。
跨物种验证
为了验证特定物种的训练模型是否能有效预测其他物种的6mA位点,跨物种验证的方式与Lv等人的研究相同。根据一个物种的全部数据训练的BERT6mA模型由其他物种的全部数据进行检验。
Pretraining and fine-tuning
一般来说,用较少的数据训练深度学习模型是困难的。为了克服这个问题,进行了预训练和微调。详细地说,作为预训练模型,基于目标物种以外的物种的整体数据训练具有wrod2vec的BERT模型。通过对目标物种的训练数据进行5倍交叉验证,对预训练模型进行了微调。根据目标物种的测试数据对微调模型进行了评估。“跨物种验证”部分中的训练模型被用作预训练模型。
Evaluation
我们通过五个统计指标来评估预测模型的性能:敏感性、特异性、准确性、马修斯相关系数(MCC)和AUC。除AUC外的其他衡量标准如下所示:

核苷酸分布模式
pLogo(概率标志)常用于检查核苷酸的分布模式,并显示特定核苷酸的出现频率或偏好。在pLogo中,每个核苷酸的高度表示其出现频率或偏好的统计显著性,这是通过使用阴性样本作为背景序列的二项检验(Bonferroni校正的P值<0.01)确定的。
为了研究阳性样本中的共现核苷酸,核苷酸对的条件概率如下所示: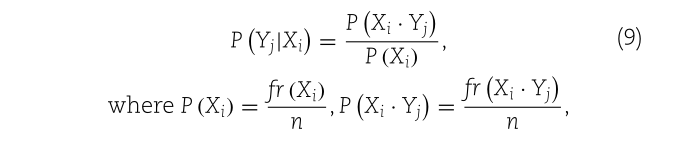
fr(Ai)是核苷酸A出现在位置i的阳性样本数,fr(Ai·Bj)是两个核苷酸A和B同时出现在位置i和j的阳性样本数,n是阳性样本数。
Attention weight of BERT6mA
BERT由多个双向Transformer编码器组成,其核心机制称为多头注意。在构成多头注意的自我注意中,word2vec生成的第i个k-mer位置的特征向量分别通过线性函数q(·)、k(·)和v(·)转换为查询、键和值向量q(xi)、k(xi)和v(xi),[ineq25]]然后,注意权重αi,jis计算为q(xi)和k(xj)的softmax-normalized点积: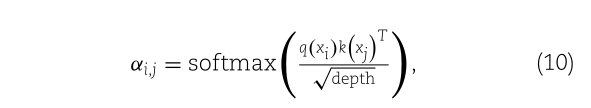
其中depth对应于q(xi)的维数。自我注意力的输出值为该值的加权和: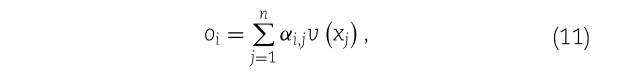
其中n是特征矩阵中特征向量的数量。为了获得多头注意,多个注意的输出被称为“头”,用Wo作为并行计算、串联和线性变换,如下所示:

式中,h是头的总数。自然语言处理研究使用注意权重来解释预测并探索关键特征。
在这项研究中,我们分析了在五倍交叉验证模型的独立测试中产生的注意权重。在每个样本中,在每个位置对所有层和头部的注意力权重进行平均。我们将平均注意力称为注意力图(图S9)。然后,在每个位置对所有样本的注意力图进行平均。为了描述注意,计算了注意图中每个位置的值与注意图中所有位置的平均值之间的偏差。此外,为了确定负责预测6mA的关键k-mer位置,将位置权重定义为每个关键位置处查询方向上的平均注意图(图S10)。由于word2vec编码每个连续4-mer的核苷酸序列,因此41个核苷酸位点被转化为38个位置。

图S9。注意力地图的生成和可视化
通过对5个交叉验证模型中所有头部和层的注意力权重进行平均,生成每个样本的注意力地图。然后,对所有样本的注意图在每个位置取平均。为了表征不同条件下的注意图,计算了注意图与平均注意图的偏差。

图S10。位置权向量的生成与分析
为了确定负责6mA预测的关键序列位点,通过对查询方向上的注意图平均生成位置权重向量。在正样本和负样本上平均每个位置的权重。然后计算每个位置上正样本和负样本平均位置权重向量的差值。
效应大小
为了研究效应大小,即含有倾向性核苷酸的样本与不含倾向性核苷酸的样本之间位置权重的差异,我们计算了每个位置的效应大小(Cohen’s d),如下所示:

n
W
i
t
h
n_{With}
nWith?和
n
W
i
t
h
o
u
t
n_{Without}
nWithout?分别是包含和不包含样本的倾向性核苷酸的数量,
x
w
i
t
h
x_{with}
xwith?和
x
w
i
t
h
o
u
t
,
x_{without},
xwithout?,分别是包含和不包含样本的首选核苷酸在位置p处的位置权重的平均值;
s
w
i
t
h
s_{with}
swith?、p和
s
w
i
t
h
o
u
t
s_{without}
swithout?分别比较p位置位置权重的标准偏差。
Results and discussion
深度学习和机器学习方法的比较.
为了比较基于深度学习的预测和基于机器学习的预测,我们使用了Lv等人提供的数据集。我们预测序列中心的腺嘌呤是否甲基化。我们使用了word2vec、NCPNF、CNCPNF、MNBE、C-MNBE、NCPNF和MNBE组合、C-NCPNF和C-MNBE组合的7种编码方法,以及LSTM、GRU、BILSTM、BiGRU、BERT、1D-CNN、BERT和BILSTM的8种深度学习模型。在这两种组合编码方法中,我们在每个位置连接了它们的特征向量。本研究共构建了56个(=7×8)深度学习模型。采用编码方法将DNA序列转化为特征矩阵后,对深度学习模型进行训练,并对训练数据进行5倍交叉验证。经过训练的五个模型的预测性能在独立测试中取平均值。在56个深度学习模型中,我们绘制了前五名深度学习模型的AUC和图2中独立测试中的最先进模型。所有采用的深度学习方法的预测性能见表S2–S12。

图2。深度学习和机器学习方法在独立测试中的性能比较。条形图显示了五种基于深度学习的方法的AUC,它们的曲线下面积更大(红色条),以及11个物种的先前模型(蓝色条),包括拟南芥(A)、秀丽隐杆线虫(B)、木麻黄(C)、黑腹果蝇(D)、草莓(E)、智人(F)、月季(G)、酿酒酵母(H),T嗜热菌(I),T.SUP5–1(J)和 Xoc。BLS256(K)。W2V、C-BE、C-NC、BE、NC、BE-NC和C-BE C-NC分别对应于word2vec、上下文二进制编码、上下文NCPNF、二进制编码、NCPNF、二进制编码和NCPNF的组合,以及上下文二进制编码和上下文NCPNF的组合。BERT、LSTM、GRU、BLSTM、BGRU、CNN、BERT BLSTM、CNN BLSTM对应于BERT、LSTM、GRU、Bi-LSTM、BiGRU、1D-CNN、带Bi LSTM的BERT和带Bi LSTM的1D-CNN。
在我们提出的深度学习模型中,对于大多数物种,BERT模型表现出更高的性能。具体来说,使用word2vec方法的BERT(名为BERT6mA)在八个物种集显示出最高的AUC,除中华绒螯蟹、中华绒螯蟹SUP5-1和Xoc以外的物种。BLS256。这意味着,与其他任何深度学习方法相比,BERT更有效地学习word2vec表示的DNA上下文模式。其次是BERT,1D CNN和Bi-GRU对一些物种表现出更高的性能,如拟南芥、秀丽隐杆线虫和智人。在许多物种中,Bi-GRU和GRU模型的性能高于Bi-LSTM和LSTM模型。在许多情况下,1D-CNN与BiLSTM的混合深度学习模型和BERT与BiLSTM的混合深度学习模型的学习效果并不优于BERT和CNN的单一模型。这些混合模型可能过于复杂,无法预测6mA位点,多个超参数(如层数和隐藏向量的维数)可能仍有待调整。
对于F以外的物种,BERT6mA比之前的模型表现出更高的AUC,F . vesca,R. chinensis,T . thermophile and Ts. SUP5-1。重要的是,对于R以外的物种,我们的BERT6mA优于基于BERT的iDNA ABT。SUP5-1,表明采用BERT模型的word2vec的有效性。另一方面,BERT6mA在F方面的性能与iDNA MS(Lv等人的射频型号)相当,但有点低。分别为胡荽和红景天。我们认为,低性能是由于中华绒螯蟹的数据量少。最后,为了验证序列窗口长度为41,我们研究了不同长度对预测性能(AUC)的影响。中心含有腺嘌呤的窗口长度分别为41、31和21。就长度而言,几乎没有观察到性能上的显著差异(表S13)。
编码方法的比较
为了描述编码方法,通过独立测试对11个物种在不同深度学习模型下的AUC进行了平均。如图3和表S14所示,CMNBE和C-NCPNF的AUC分别高于MNBE和NCPNF。这可能是因为C-MNBE和C-NCPNF编码涉及多个核苷酸的信息,而MNBE和NCPNF只编码一个核苷酸信息。尤其是在基于RNN的模型中,C-MNBE和C-NCPNF的AUC显著高于MNBE和NCPNF。

图3 不同编码方法的性能比较。对于具有不同编码方法的深度学习模型,在独立测试中,对所有物种的曲线下面积(AUC)进行平均,并计算其方差。W2V、C-BE、C-NC、BE、NC、BE-NC和C-BE C-NC分别对应于word2vec、上下文二进制编码、上下文NCPNF、二进制编码、NCPNF、二进制编码和NCPNF的组合以及上下文二进制编码和上下文NCPNF的组合。采用单侧配对样本t检验比较MNBE和C-MNBE之间的AUC,以及NCPNF和C-NCPNF之间的AUC。
上下文编码方法(C-MNBE和C-NCPNF)比基于RNN模型的非上下文(MNBE和NCPNF)预测性能提高,表明考虑上下文核苷酸模式是有效的。有趣的是,1D-CNN和BERT即使在使用非上下文编码方法时也表现出很高的预测性能。这表明,与基于RNN的模型相比,1D-CNN能够通过过滤器学习连续的核苷酸模式,并且BERT能够很好地学习核苷酸之间的上下文信息或依赖性。在许多深度学习模型中,组合NCPNF和BE方法以及C-NCPNF和C-BE方法比单一编码方法表现出更高的性能。
当使用BERT和1D-CNN时,word2vec表现出非常高的性能。我们认为,BERT和1D-CNN分别捕获了word2vec表示上下文信息的依赖关系和模式。这些结果表明,编码方法的有效性取决于深度学习的体系结构。
最先进模型的比较
最近,基于深度学习的模型,如Deep6mA和6mA Pred,已经展示了最先进的性能。Deep6mA通过CNN和Bi-LSTM提取序列特征,学习核苷酸之间的依赖信息。它的表现优于SNNRice6mA和MM-6mAPred。6mA Pred通过LSTM提取6mA的相关特征,并通过注意机制捕捉6mA与非6mA的序列差异。它的性能优于SNNRice6mA和iDNA6mA rice。为了描述BERT6mA的性能,我们使用Lv等人的数据集将BERT6mA与Deep6mA和6mA Pred进行了比较。这些深度学习模型在训练数据集上进行了5倍交叉验证训练,并在独立数据集上进行了测试。Deep6mA和6mA Pred的源代码与默认参数值一起使用。
如图4所示,与木麻黄中的Deep6mA和6mA Pred相比,BERT6mA表现出了较高的性能。维斯卡、酿酒酵母和苏普特5-1。在其他物种中,BERT6mA表现出类似的表现。尽管6mA Pred在T。嗜热性,这表明其他物种的性能较低。由于这两种机制,BERT6mA实现了对不同物种和数据量的稳健预测。一种是,4-mer word2vec方法编码的特征矩阵包含与6mA相关的上下文信息,因为word2vec将4-mer氨基酸的规则性嵌入为单个单词。另一个是BERT6mA考虑序列上下文中所有四个词之间的依赖关系。

图4。BERT6mA与Deep6mA和6mA Pred的比较。在独立试验中,对拟南芥(A)、秀丽隐杆线虫(B)、木麻黄(C)、黑腹果蝇(D)、红腹果蝇(E)、智人(F)、月季(G)、酿酒酵母(H)、T等11个物种绘制了ROC曲线。嗜热菌(I),T.SUP5–1(J)和 Xoc。BLS256(K)。使用了在5倍交叉验证中显示曲线下最高面积的模型。
跨物种验证
为了研究哪些物种对具有6mA特异性特征或序列相似性,进行了跨物种验证。如图5所示,这三种植物分别是拟南芥、中华绒螯蟹和Fvesca和两种动物H.sapiens和D.melanogaster的AUC较高,表明它们具有相似的核苷酸分布模式。另一方面,在T嗜热菌、线虫和Xoc。BLS256较低,表明这些物种的核苷酸分布存在显著差异。AUCs的变化将由物种的差异引起,因为所用物种的6mA数据主要通过SMRT测量。这些结果与Lv等人提出的基于机器学习的跨物种验证结果一致。有趣的是,一些植物和动物在6mA左右拥有非常相似的核苷酸模式。

图5 跨物种验证的曲线下面积(AUC)性能。根据一个物种的整体数据训练的带有word2vec的BERT模型,根据另一个物种的整体数据进行评估。
通过预训练和微调构建BERT6mA
就R.chinensis而言,与Lv等人的机器学习模型相比,BERT6mA表现出较低的性能,可能是因为它的数据不足。为了提高预测性能,采用了预训练和微调。利用中华绒螯蟹的训练数据,通过5倍交叉验证对每个物种的预训练模型进行微调。根据中华绒螯蟹的试验数据对微调模型进行了评估。通过对F vesca的数据进行预训练,独立测试中的AUC值得到改善、D.melanogaster、H.sapiens、A.thaliana、C.equisetifpolia、Ts.SUP5-1和S.cerevisiae。具体来说,F上的预训练。在独立测试中,vesca极大地提高了微调模型的预测性能(图6和表S15)。预训练模型的AUC高于非预训练模型和之前的模型。预训练和微调在提高预测性能方面非常有效。

图6 在独立试验中通过预训练和微调提高月季的性能。BERT6mA模型由除中国红豆杉之外的所有物种数据集预训练。通过5倍交叉验证,对训练前的模型进行了微调。根据中华绒螯蟹的试验数据对微调后的模型进行了评估。条形图显示了预训练模型(红色)、非预训练模型(绿色)和先前模型(蓝色)的曲线下面积(AUCs)。
核苷酸偏好分析
pLogo被应用于=-=四个物种的阳性样本,包括R. chinensis,F . vesca,Xoc BLS256和秀丽隐杆线虫用于可视化和检查6mA左右核苷酸的偏好模式。如图7A所示,中华绒螯蟹和F vesca在6mA左右相似,但与Xoc不同、BLS256。这表明== R chinensis和F vesca的AUCs 源自6mA左右的类似核苷酸模式。相反,中华绒螯蟹和秀丽隐杆线虫之间的AUC较低,F . vesca和秀丽隐杆线虫,在chinensis vesca和Xoc之间。BLS256和F之间。维斯卡和Xoc。跨物种验证中的BLS256可能是由不同的模式引起的。通过同样的机制,中华绒螯蟹在F vesca在秀丽隐杆线虫和Xoc上的性能高于chinensis\红杉微调、预训练模型。BLS256。从这些结果来看,对其他在6mA左右具有类似核苷酸模式的物种进行预训练对于提高预测性能至关重要==。
如图7B所示,在R.chinensis中,G在位置22的条件概率很高。此外,相对于出现在6mA左右的任何核苷酸而言,位点25和28处A的条件概率都很高,这表明这些A对6mA至关重要。26位G和27位C的条件概率较高,表明这两个核苷酸同时发生。在pLogo的16到20个地点观察到了多个As(图7A),但它们的条件概率并不高。多重As将出现在相对广泛的范围内,从16到20,而不是在特定地点。


图7 核苷酸分布在6mA左右。(A) 为了确定6mA左右的关键核苷酸,将pLogo应用于月季(顶部)、草莓(第二排)、Xoc的阳性数据。BLS256(第三排)和秀丽隐杆线虫(底部)。红色标记和黑色标记的位置表示显著的临界位置,Bonferroni校正的P值<0。01和提供特定核苷酸出现频率>75%的位置。(B) 在中国红豆杉6mA序列中,显示了核苷酸对(X和Y)的条件概率。呈现的核苷酸对在阳性样本中出现的频率高于阴性样本。轴上的数字和字母分别表示位置和核苷酸。
注意力和位置权重分析
为了确定BERT6mA关注的核苷酸分布,我们通过5倍交叉验证在五个训练模型的独立测试中可视化了注意图。图8A显示了微调后的中华绒螯蟹的平均注意力图F vesca预训练模型和中华绒螯蟹未经任何预训练的训练模型。注意,该位置被定义为word2vec转化的核苷酸位点。一个位置包含核苷酸序列的4-mer位点的信息,因为连续的4-mer氨基酸被编码为单个单词。例如,位置 i拥有从i到i 3的站点信息。预训练模型中的平均注意力在查询中键的某些特定位置呈现高值或低值(图8A)。特定位置的值加权良好,在预训练模型中比在非预训练模型中观察到的更清楚。这一结果表明,预训练模型注意特定位置的关键特征,提高预测性能。
为了进一步确定哪些关键位置在6mA和非6mA之间产生不同的注意权重,我们分析了每个位置正样本和负样本之间的位置权重差异,如图8B所示。预训练模型比未预训练模型提供了更高的峰值。在预训练模型中,在pLogo中出现具有统计意义的核苷酸的位置,位置权重的差异更大。这表明BERT6mA关注阳性和阴性样本之间可区分的核苷酸位置。在预训练模型中,位置权重的影响大小为:位置16至20处的AA包括和AA不包括样品,位置22处的G包括和不包括样品,位置25处的A包括和不包括样品,位置26和27处的GC包括和不包括样品,在A包括和不包括位置28处的样品之间观察到峰值(图9),这表明位置权重反映了6mA相关核苷酸的存在。此外,预训练模型中的峰值分别大于非预训练模型中的峰值。这表明,预训练的BERT6mA模型使用注意权重来关注负责6mA预测的核苷酸偏好位点,如pLogo所示(图7A)。

图8 可视化注意图中的偏差以及正样本和负样本之间平均位置权重的差异。(A) 平均注意力图中偏差的热图。位置(i,j)处的注意力权重是查询的第i个位置向量和键的第j个位置向量的softmax归一化点积。在对月季精调、佛手柑预训练模型(左)和未经任何预训练的月季训练模型(右)进行独立测试时,计算平均注意力。水平条表示平均注意力图的偏差。(B) 计算了两个模型的阳性和阴性样本之间的平均位置权重差异:中国红杉微调,F vesca预训练模型(红线)和未经任何预训练的中华绒螯蟹训练模型(蓝线)。
这些结果表明,BERT6mA产生注意权重,以了解哪些核苷酸受到注意。它们有助于提取序列模式的关键,解决深度学习中固有的黑盒问题。DNA模式或基序的注意力权重对于生物学家发现DNA模式及其相关生物功能之间的关系非常有用。
Web服务器构造
为了方便社区,我们使用apache(2.4.18)和flask(1.1.2)构建了BERT6mA的web服务器应用程序。用户可以从http://kurata35访问服务器。生物6mA预测,需要输入或上传41 bp长的DNA序列,腺嘌呤以FASTA格式位于中心。除了预测标签和分数外,还提供了注意权重。有关其他概述,请参阅网站的帮助。web
Conclusions
我们将7种编码方法与8种深度学习模型相结合,构建了56种深度学习模型,用于11个物种的6mA位点预测。在这些深度学习模型中,在一项独立测试中,BERT6mA在八个物种中表现最好。另一方面,对于样本量较小的物种,BERT6mA并没有优于机器学习模型。为了解决这个问题,我们成功地将预训练和微调方法应用于BERT6mA。请注意,当微调所使用的样本序列与预训练所使用的样本序列显示出较低的相似性时,这是无效的。重要的是选择两个物种的数据集,这两个物种的序列相似性约为6MA。最后,BERT6mA与最先进的深度学习模型相比表现出更高或可比性。除了预测之外,BERT6mA还分析了注意力权重,以提出6mA位点所需的关键核苷酸模式。
Funding
科学研究补助金(B)(19H04208);以及JSPS研究员资助金(19F19377);来自日本科学促进会(JSPS)。美国国立卫生研究院U19AG05537301和R01AR069055的拨款项目(给M.M.H.和H.W.D.)。