[NSF 2021]
运用图神经网络进行命名实体识别
原文链接:https://par.nsf.gov/biblio/10287109
1摘要
???????对一般领域的命名实体识别(NER)进行了很好的研究,最近的系统在识别普通实体类型方面达到了人类水平的表现。然而,对于那些往往具有复杂语境和行话实体类型的专业领域来说,NER的性能仍然是中等的。为了应对这些挑战,我们建议在全局核心推理关系和局部依赖关系的基础上明确连接实体提及,以建立更好的实体提及表示。在我们的实验中,我们通过图神经网络将实体提及关系纳入其中,并表明我们的系统在两个不同领域的数据集上明显地提高了NER的性能。我们进一步表明,即使只有少量的标记数据可用,我们提出的轻量级系统也能有效地将NER性能提升到一个更高的水平,这对于特定领域的NER来说是可取的。
1 引言
???????对一般领域的命名实体识别(NER)进行了很好的研究,最近的系统在识别少数常见的命名实体类型(如人和组织)方面取得了接近人类水平的性能,主要受益于神经网络模型的使用(Ma和Hovy,2016;Yang和Zhang,2018)和预训练的语言模型(LM)(Akbik等人,2018;Devlin等人,2019)。然而,对于专业领域来说,其性能仍然适中,这些领域往往具有多样化和复杂的背景,以及更丰富的语义相关实体类型(例如,生物医学领域的细胞、组织、器官等)。考虑到这些挑战,我们假设意识到同一实体以及语义相关实体的重复出现将导致特定领域的更好的NER性能。
???????因此,我们建议明确地将文档中具有核心关系或紧密语义关系的实体提法连接起来,以更好地学习实体提法的表征。确切地说,如图1所示,我们首先将同一实体的重复提及连接起来,即使它们相隔数句。例如,被命名的实体 "肿瘤血管 "同时出现在标题和S6句子中,但其语境却完全不同。连接文档中重复提到的内容可以整合上下文线索,并能对其实体类型进行一致的预测。
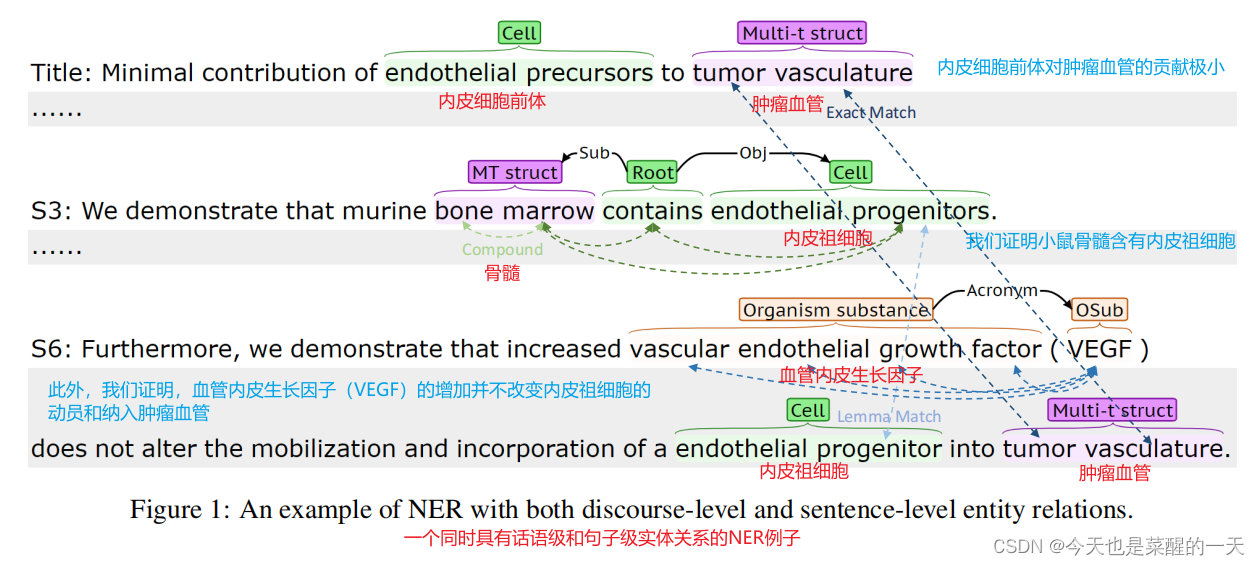
???????其次,我们还根据句子层面的依存关系将实体提及连接起来,以有效识别语义上相关的实体。例如,句子S3中的两个实体,即多组织结构类型的 "骨髓 "和细胞类型的 “内皮祖细胞”,在依赖树中分别是谓词 "包含 "的主语和宾语。如果系统能够可靠地预测一个实体的类型,我们就可以更容易地推断另一个实体的类型,因为我们知道它们在依赖树上是密切相关的。
???????我们通过使用图谱神经网络(GNNs)纳入这两种关系,具体而言,我们使用图谱注意力网络(GATs)(Velickovic等人,2018),该网络已被证明对一系列任务有效(Sui等人,2019;Linmei等人,2019)。经验结果表明,我们的轻量级方法可以为序列标签模型学习更好的词表征,并在两个数据集上进一步提高NER的性能,即来自生物医学领域的AnatEM(Pyysalo和Ananiadou,2014)数据集和来自行星科学领域的火星(Wagstaff等人,2018)数据集,比基于强大的LMs基线。此外,考虑到缺乏注释对特定领域NER的挑战,我们绘制了学习曲线,并表明在有限的注释可用时,利用实体提及之间的关系可以有效和持续地提高NER的性能。
2 相关工作
???????NER研究由来已久,最近的方法(Yang and Zhang, 2018; Jiang et al., 2019; Jie and Lu, 2019; Li et al., 2020)使用BiLSTM-CNN-CRF(Ma and Hovy, 2016)等神经网络模型和BERT(Devlin et al., 2019)和FLAIR(Akbik et al., 2018)等语境嵌入,将一般领域的NER性能提高到人类水平。然而,由于有限的注释和处理复杂的特定领域语境的挑战,特定领域的NER性能仍然是中等的。
???????我们旨在通过考虑实体提及之间的核心推理关系和语义关系来进一步提高NER的性能。这与通常认为NER是在核心推理解析或实体关系提取之前进行的上游任务的方式相反。这个想法与最近在多个信息提取任务之间进行联合推理的工作一致(Miwa和Bansal,2016;Li等人,2017;Bekoulis等人,2018;Luan等人,2019;Sui等人,2020;Yuan等人,2020),包括NER、核心推理和关系提取,通过挖掘提取之间的依赖关系。然而,联合推理方法需要对所有目标任务进行注释,并旨在提高所有任务的性能,而我们的轻量级方法旨在提高基本的NER任务的性能,不需要额外的注释(通常对特定领域不可用)。
???????我们的方法也与最近几种鼓励实体提及之间的标签依赖性的NER的神经方法有关。Pooled FLAIR模型(Akbik等人,2019)提出了一个全局池化机制来学习单词表征。Dai等人(2019)使用一个带有正则器的核心参考层来协调词的表征。与我们的工作密切相关的是,Qian等人(2019)使用图神经网络来捕捉同一单词的重复,但在一个更密集的图中,包括相邻单词之间的边缘,并且是为了完全覆盖低层编码层。记忆网络(Gui等人,2020;Luo等人,2020)也被用来存储和完善基础模型的预测,考虑到单词的重复或共同出现。此外,依赖关系通常被用来连接实体进行关系提取(Zhang等人,2018;Bunescu和Mooney,2005),但我们的目的是通过将一个实体与句子中其他密切相关的实体联系起来,更好地推断其类型。
3 模型结构
???????我们的实体关系图(EnRelG)系统主要包含5层,如图2所示:嵌入层、编码层、GNNs层、融合层和解码层。
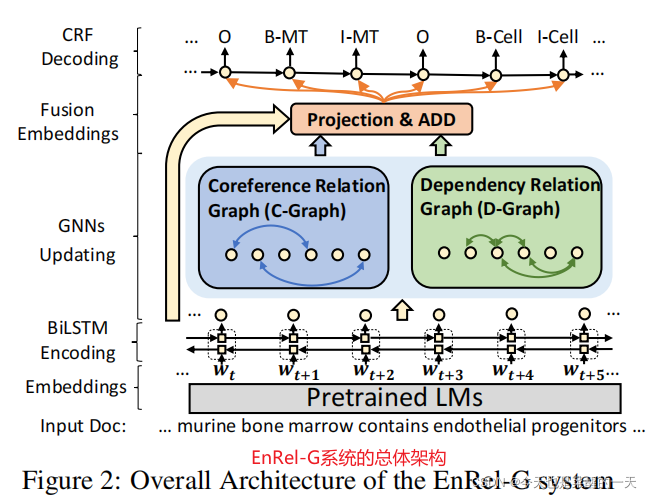
3.1 嵌入层
???????我们选择BERT-base LM作为我们的嵌入层。对于特定领域的数据集,我们在生物医学领域使用BioBERT(Lee等人,2020),在行星科学领域使用SciBERT(Beltagy等人,2019)。具体来说,对于一个有n个单词的输入文档 D = [ w 1 , w 2 , . . . , w n ] D=[w_1, w_2, ..., w_n] D=[w1?,w2?,...,wn?],BERT模型将输出一个上下文单词嵌入矩阵 E = [ w 1 , w 2 , . . . , w n ] ∈ R n × d 1 E=[w_1, w_2, ..., w_n]∈R^{n×d_1} E=[w1?,w2?,...,wn?]∈Rn×d1?,每个单词有一个 d 1 d_1 d1? 维向量。
3.2 编码层
???????为了捕捉连续的上下文信息,我们使用BiLSTM层对BERT模型的词嵌入进行编码。我们将前向和后向的LSTM隐藏状态连接起来作为编码的表示,然后得到嵌入矩阵 E l s t m = B i L S T M ( E ) ∈ R n × d 2 E^{lstm} = BiLSTM(E) ∈ R^{n×d_2} Elstm=BiLSTM(E)∈Rn×d2?,每个词有一个 d 2 d_2 d2? 维的向量。
3.3 图神经网络层
???????对于GNNs层,我们首先介绍如何利用实体之间的全局核心推理关系(coreference graph,C-graph)和局部依赖关系(dependency graph,D-graph)建立实体关系图,然后描述GNNs模型如何将其纳入词的表示。
???????推理关系图。对于每个文档,我们根据核心推理关系建立一个图
G
C
=
(
V
,
A
C
)
G^C=(V, A^C)
GC=(V,AC),其中
V
V
V 是表示文档中所有单词的节点集合,
A
C
A^C
AC 是相邻矩阵。具体来说,我们使用 3 种句法核心推理线索来近似实体核心推理关系,如图1:
???????(1)精确匹配,如果两个名词相同,则连接在一起,例如,标题和 S6 中的 “肿瘤血管”;
???????(2)词法匹配,如果两个名词有相同的词法,则连接在一起,例如。S3和S6中的 "progenitors "和 “progenitor”;
???????(3)缩略语匹配,缩略语词与所有全称表达词相连,如S6中的 "VEGF "和 “血管内皮生长因子”。
???????对于每个连接的节点对
(
i
,
j
)
(i,j)
(i,j),我们设置
A
i
,
j
C
=
1
A^C_{i, j} = 1
Ai,jC?=1。我们还为每个节点添加一个自我连接(
A
i
,
i
C
=
1
A^C_{i, i} = 1
Ai,iC?=1),以保持词的原始语义信息。
???????依赖关系图。我们根据句子层面的依赖关系,为每个文档建立一个依赖关系图
G
D
=
(
V
,
A
D
)
G^D=(V,A^D)
GD=(V,AD)。我们首先使用
s
c
i
s
p
a
C
y
2
scispaCy^2
scispaCy2 工具对每个句子进行解析,然后在依赖树中连接以下词对:
???????(1)主语头词和宾语头词及其谓语,我们把它们连接起来,以加强主语和宾语的实体之间的相互作用。例如,"marrow (骨髓)"和 "progenitors(祖先) "与S3中的谓语 “contains(包含)”。(2)复合词&头词,我们把复合词和它们的头词联系起来,因为它们经常同时存在于一个实体中。例如,S3中的 "骨 "和 “髓”。和以前一样,我们为每个连接对
(
i
,
j
)
(i, j)
(i,j)设置
A
i
,
j
D
=
1
A^D_{i,j} = 1
Ai,jD?=1,同时为每个节点添加自我连接(
A
i
,
i
D
=
1
A^D_{i,i} = 1
Ai,iD?=1)。
???????然后,我们用基于 GNN 的实体关系图更新编码后的词嵌入,特别是 GATs。由于节点代表文档中的词,我们将编码层中的图中的节点表示初始化为
E
l
s
t
m
=
[
w
1
l
s
t
m
,
w
2
l
s
t
m
,
.
.
.
,
w
n
l
s
t
m
]
E^{lstm} = [w^{lstm}_1, w_2^{lstm}, ..., w_n^{lstm}]
Elstm=[w1lstm?,w2lstm?,...,wnlstm?]。图注意力机制通过聚合其邻居的表示和其相应的归一化关注分数,将节点
w
i
l
s
t
m
w_i^{lstm}
wilstm? 的初始表示更新为
w
i
g
n
n
w_i^{gnn}
wignn?。

???????如公式(1),我们有 K 个关注头,并把它们串联起来(||)作为最终的表示。对于头 k,我们用
W
k
W^k
Wk 对所有的相邻节点(
N
i
N_i
Ni?,从相邻矩阵
A
A
A 中得到)进行加权,然后用注意力得分
α
i
j
k
α^k_{ij}
αijk? 进行汇总。 σ是激活函数LeakyReLU。注意力得分
α
i
j
k
α^k_{ij}
αijk? 的获得方法如下(
a
T
a^T
aT 是一个权重向量)。

???????对于两个关系图中的每一个,我们都使用一个独立的图注意层。两个GAT 的输出词表示被表示为: G C = [ w 1 g n n ( C ) , w 2 g n n ( C ) , . . . , w n g n n ( C ) ] ∈ R n × d 3 G^C = [w_1^{gnn(C)},w_2^{gnn(C)}, ..., w_n^{gnn(C)}] ∈ R^{n × d_3} GC=[w1gnn(C)?,w2gnn(C)?,...,wngnn(C)?]∈Rn×d3? 和 G D = [ w 1 g n n ( D ) , w 2 g n n ( D ) , . . . , w n g n n ( D ) ] ∈ R n × d 3 G^D = [w_1^{gnn(D)},w_2^{gnn(D)}, ..., w_n^{gnn(D)}] ∈ R^{n × d_3} GD=[w1gnn(D)?,w2gnn(D)?,...,wngnn(D)?]∈Rn×d3? ,每个词的维度为 d 3 d_3 d3?。
3.4 融合层
???????与Sui等人(2019)类似,我们也使用融合层来混合编码的词嵌入和GNNs更新的词嵌入。我们首先使用线性变换将这些嵌入投射到相同的隐藏空间,然后将它们相加,如 F = W N E l s t m + W C G C + W D G D F = W_NE^{lstm} + W_CG^C + W_DG^D F=WN?Elstm+WC?GC+WD?GD,其中 W N W_N WN? , W C W_C WC?, W D W_D WD? 是可训练的权重。然后,我们将有一个特征矩阵 F ∈ R n × d 4 F∈R^{n×d_4} F∈Rn×d4?,用于混合 n n n 个词的顺序语境信息和全局实体关系。
3.5 解码层
???????最后,一个条件随机场(CRF)(Lafferty等人,2001)层被用来将丰富的嵌入
F
=
[
f
1
,
f
2
,
.
.
.
,
f
n
]
F=[f_1, f_2, ..., f_n]
F=[f1?,f2?,...,fn?] 解码为一串标签
y
=
{
y
1
,
y
2
,
.
.
.
,
y
n
}
y=\{y_1, y_2, ..., y_n\}
y={y1?,y2?,...,yn?}。在训练短语中,我们通过最小化相对于金标签的负对数可能性损失来优化整个模型。
4 实验
???????我们在两个特定领域的数据集上测试我们的模型:来自生物医学领域的AnatEM(Pyysalo和Ananiadou,2014)和来自行星科学领域的Mars(Wagstaff等人,2018)。AnatEM在1212个文档中注释了12种类型的实体,有13701次实体提及;Mars有117个较长的文档,有4458次实体提及,包含3种类型。
4.1 基线模型
???????NCRF++(Yang and Zhang, 2018)是一个开源的神经序列标签工具包。我们使用BiLSTM-CNN-CRF结构作为基线。
???????FLAIR(Akbik等人,2018)是一个基于BiLSTM的字符级预训练LM,它已经被用于许多NER系统(Jiang等人,2019;Wang等人,2019)。我们使用它的嵌入与BiLSTM-CRF架构作为基线。
???????Pooled FLAIR(Akbik等人,2019年)是FLAIR模型的扩展版本,具有全局记忆和同一单词的池化机制,这有助于一致预测核心词的实体提及。我们也用它的嵌入与BiLSTM-CRF架构作为基线。
???????Tuning Bio/SciBERT 我们还使用具有BiLSTM-CRF架构的Bio/SciBERT作为AnatEM/Mars数据集的基线,与我们的系统相比,它没有GNNs层或融合层。
4.2 结果
???????为了缓解随机湍流,我们使用不同的随机种子对所有系统进行了五次训练,并使用相同的脚本5评估它们在测试集上的平均性能,如表1所示。
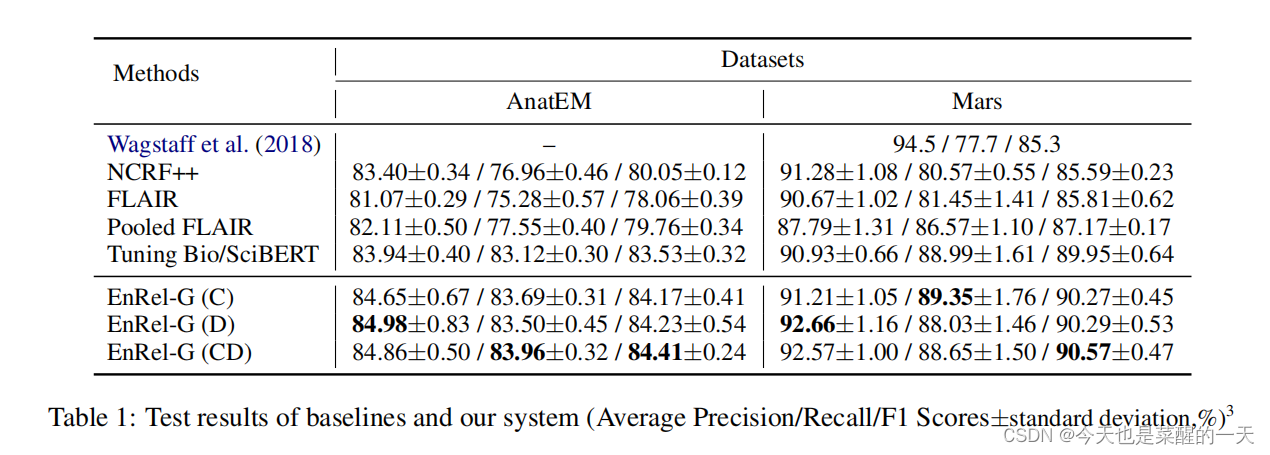
???????我们可以看到,在所有的系统中,我们的系统在全局实体核心推理和局部依赖关系方面的表现最好。与AnatEM上的BioBERT相比,它的平均F1得分提高了0.88分(84.41%对83.53%),与Mars上的SciBERT相比,提高了0.62分(90.57%对89.95%)。此外,核心推理和依赖关系都有助于提高NER的性能。具体来说,我们的模型与核心推理或依赖关系图在AnatEM数据集上的F1得分提高了0.64分或0.7分,在Mars数据集上提高了0.32分或0.34分。
4.3 学习曲线
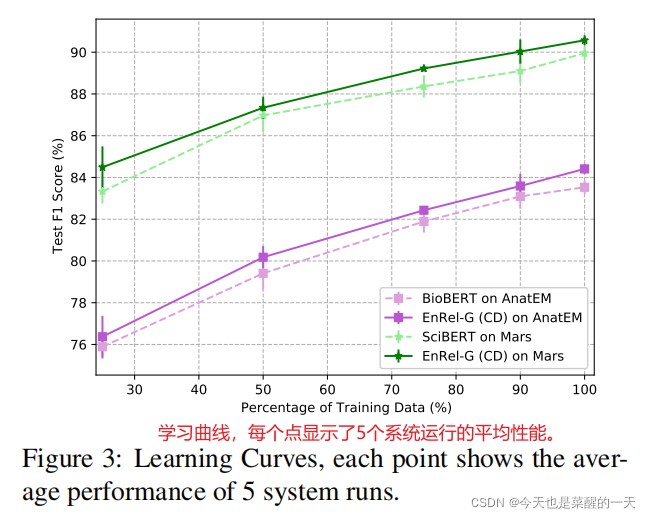
???????特定领域NER系统的一个主要限制是缺乏注释,因此,最好地利用标记数据是至关重要的。学习曲线(图3)显示,即使只有极少量的标注数据(训练数据的四分之一),利用实体提及之间的关系可以有效地将NER的性能提升到一个更高的水平,这在AnatEM数据集和Mars数据集上都是如此。
4.4 计算成本的分析
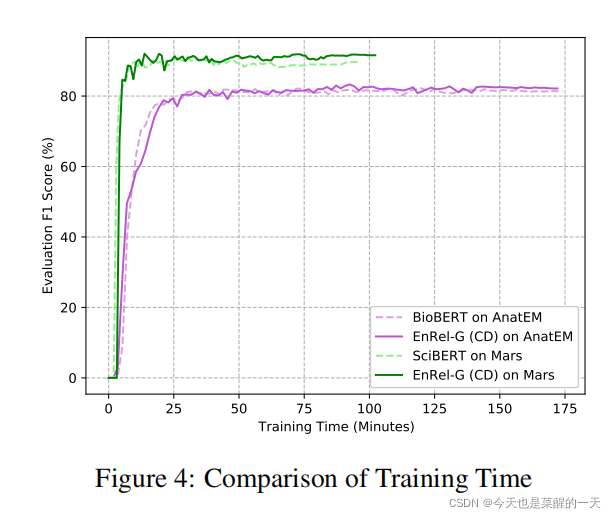
???????尽管微调预训练的LMs提高了许多NLP任务的性能,但一个限制是训练时间的增加。因此,在预训练的LMs基础上建立高效的计算模型是非常重要的。如图4所示,我们带有GNNs层的模型并没有增加微调BERT模型的时间成本。带或不带GNNs层的方法的训练时间是相似的。
4.5 依赖关系图中的边缘选择
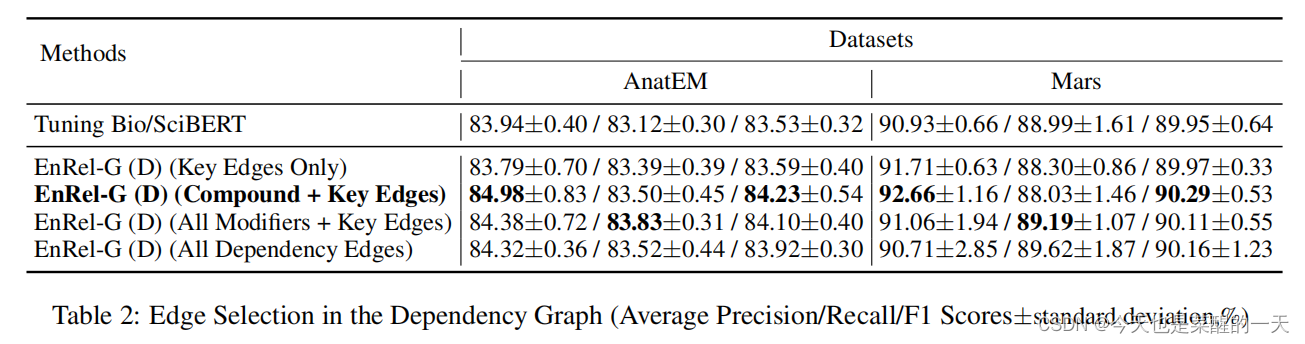
???????为了构建句子层面的依存关系图,我们只选择了两类依存关系:主语、宾语和它们的谓语之间(关键边)以及复合修饰语和它的头部词之间。如表2所示,我们也尝试将所有的修饰语与它们的头词连接起来,发现这样做产生的性能略差,原因可能是除化合物之外的许多修饰语本身不是实体。此外,包括所有的依存关系边缘也比使用这两种选定的依存关系产生更差的性能,可能是由于同样的原因,依存关系树中的许多节点不是实体提及的部分,许多依存关系并不直接有助于捕捉实体之间的关系。
# 5 结论 ???????在这项工作中,我们明确地捕获了实体提法之间的全局核心推理和局部依赖关系,并使用图神经网来纳入这些关系以改善特定领域的NER任务。在两个数据集上的实验结果显示了这种轻型方法的有效性。我们还发现,实体关系的选择对系统的性能很重要。未来的工作可能会考虑使用GNNs来纳入外部知识以提高性能。
如有错误,请及时联系我,谢谢