文献:Defining HLA-II Ligand Processing and Binding Rules with Mass Spectrometry Enhances Cancer Epitope Prediction
?为什么分享这一篇文献?
这一篇文章是来自Neon Therapeutics公司的,这家公司是肿瘤新生抗原领域的领导者;同时本文采用质谱数据定义了HLA-II型配体处理和结合规则增强了癌症表位预测!值得阅读!质谱数据是未来新生抗原的重要数据来源!
可以看一下这篇报道:Neon Therapeutics:融资5000万如何玩转“肿瘤疫苗”?
目录
????????3.1、MAPTAC可以实现单个HLA-II等位基因的可扩展性分析
????????3.2、MAPTAC 解析 HLA-II 肽结合基序
????????3.3、在MAPTAC数据上训练的算法预测免疫原性
????????3.4、专业 APC 是肿瘤微环境中的主要 HLA-II 呈现者
????????3.5、特定基因有权访问 HLA-II 呈递途径
????????3.6、一些基因区域被优先加工但缺乏明显的切割基序
????????3.7、集成表示规则极大地增强了 HLADR 配体组预测
????????3.8、模型可以预测由专业 APC 提供的肿瘤衍生 HLA-II 肽
前言:尽管它们在指导 T 细胞反应中发挥作用,但 HLA-II 表位仍然难以预测,阻碍了它们的治疗潜力。 解决不同的 HLA-II 基序并精确定位由专业 APC (抗原提呈细胞)呈现的肿瘤表位。 这些数据能够改进 HLA-II 表位预测和治疗靶向。
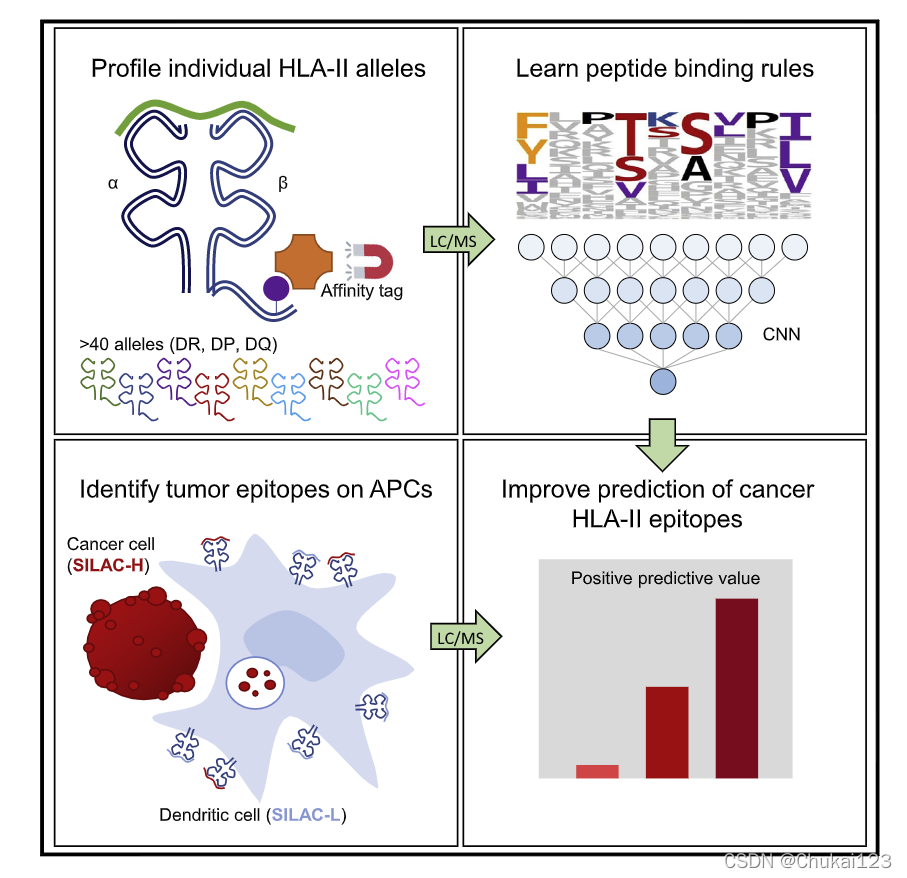
Highlists:
1、亲和力标记(affinity tag)协议使单个HLA-II等位基因的蛋白质组学分析能够实现;
2、即使在“热门”的肿瘤中,专业 APC(不是癌细胞)也会驱动 HLA-II 表达;
3、细胞定位影响哪些被吞噬的癌症蛋白被呈现;
4、用于结合和处理的机器学习模型改进了 HLA-II 预测;
1、Summary
越来越多的证据表明CD4+T细胞可以识别癌症特异性抗原并控制肿瘤生长。然而,预测人白细胞抗原类II分子(HLA-II)呈现的抗原仍然困难,阻碍以最佳方式针对它们进行治疗的努力。障碍包括HLA-II途径的不准确的肽结合预测和未解决的复杂性。
为了解决这些挑战,本文开发了一种用于发现HLA-II结合基序的改进技术,并对肿瘤配体组进行综合分析,以学习肿瘤微环境中相关的处理规则。并且分析了 >40 个 HLA-II 等位基因,并表明结合基序对 HLA-DM 高度敏感,HLA-DM 是一种肽加载伴侣。同时还证明肿瘤内HLA-II呈递由专业抗原呈递细胞(APC)而不是癌细胞主导。
综合这些观察结果,本文开发了准确预测 APC 配体组的算法,包括来自吞噬癌细胞的肽。 这些工具和生物学见解将有助于改进以 HLA-II 为导向的癌症疗法。
2、Introduction
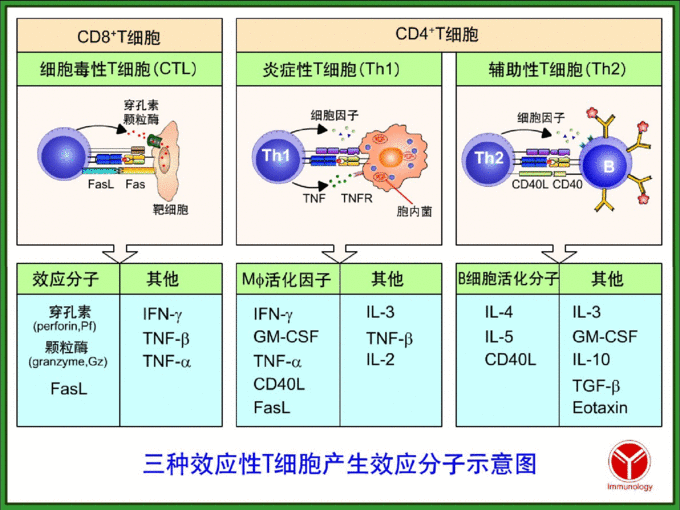
近期癌症免疫疗法的努力旨在诱导患有癌症抗原的T细胞反应,有组织突变序列称为Neoantigens。目前,这些努力集中于引发CD8+T细胞对人白细胞抗原等级I(HLA-I)呈现的配体的反应。然而,研究表明,CD4+T细胞还可以识别HLA-II所呈递的配体并有助于肿瘤对照。CD8+T细胞是针对MHC I型的,CD4+T细胞是针对MHC-II型。
理想情况下,癌症疫苗和其他免疫疗法将利用指导 CD4+ T 细胞反应,但到目前为止,这些反应主要是预测会引发 CD8+ T 细胞反应的表位附带的。
阻碍准确识别 HLA-II 癌症抗原的一个障碍是获得了解肽结合规则所需的全面、高质量的数据。 三个高度多态的规范 HLA-II 基因座 HLA-DR、-DP 和 -DQ 所需的数据,其中每个等位基因变体表现出不同的肽结合偏好。
一种广泛使用的定义肽结合基序的方法是一种生化测定法,该测定法在没有生理伴侣的情况下测量单个肽的亲和力。 测得的亲和力数据覆盖范围仅限于常见的欧洲 HLA-DR 等位基因,即使对于这些等位基因,预测准确性也落后于 HLA-I(Barra 等人,2018;Karosiene 等人,2013)。 基于质谱 (MS) 的配体组学应通过提供可扩展性和内源性肽加载条件来改进预测。
尽管如此,天然配体组是多等位基因的,隐藏了获得准确训练数据所需的肽到等位基因定位信息。 使用反卷积和单等位基因细胞系解决 HLA-I 的这个问题已经取得了进展(Abelin 等人,2017;Andreatta 等人,2017;Bassani-Sternberg 和 Gfeller,2016)。 虽然单等位基因 HLA-II 配体组数据集是使用低通量转基因小鼠模型生成的(Clement 等人,2016;Ooi 等人,2017),但 HLA-II 缺陷细胞系(á lvaro-Benito 等人) ., 2018) 或具有纯合 HLA-DR 等位基因的细胞系 (Nanaware et al., 2019),对于跨多个细胞背景的 HLA-II 等位基因特异性数据集生成仍然没有全面且可扩展的解决方案。
另一个挑战是肿瘤抗原最有可能进入 HLA-II 呈递途径的不确定性。 最近基于 MS 的研究调查了肿瘤样本的 HLA-II 配体组,但尚未解决专业抗原呈递细胞 (APC) 或癌细胞是否呈递治疗相关的 HLA-II 抗原。此外,我们目前不知道肿瘤抗原的 HLA-II 加工是否主要依赖于吞噬作用或自噬。 根据在相关细胞类型中哪个途径占主导地位,在哪些蛋白质更适合作为 HLA-II 配体来源方面可能存在巨大差异。 使问题更加复杂的是,没有系统的方法来确定蛋白质中的哪些区域最有可能产生 HLA-II 配体,尽管流行的理论认为蛋白质序列特征应该影响 HLA-II 加工潜力(Barra et al., 2018; Paul 等人,2018)。
为了研究可治疗靶向 HLA-II 抗原的加工和呈递规则,本文采用了双管齐下的方法:(1) 改进肽结合预测,?(2) 确定 HLA-II 配体如何在肿瘤微环境 (TME) 中加工和呈递 .
为了了解等位基因特异性肽结合规则,本文开发了一种可扩展的单等位基因 HLA 配体组分析工作流程,称为 MAPTAC(带有标记的等位基因构建体的单等位基因纯化)。 MAPTAC 使我们能够清楚地解析大于 40 个 HLA-II 等位基因的肽结合基序,并训练能够准确识别免疫原性病毒表位和新抗原的结合预测算法。
为了改进 HLA-II 加工预测,本文分析了肿瘤样本,建立了专业的 APC 作为肿瘤内 HLA-II 表达的主要来源,并定义了这些细胞优先加工的基因和基因区域集。 然后,证明了整合结合和处理特征的算法可以预测天然 APC 配体,更重要的是,可以预测来自内吞癌细胞的 HLA-II 配体子集。 这些在理解治疗相关 HLA-II 抗原的加工和呈递规则方面取得的进展将使旨在利用 CD4+ T 细胞反应的治疗成为可能。
生理伴侣?:一类在序列上没有相关性但有共同功能的蛋白质,它们在细胞内帮助其他含多肽的结构完成正确的组装,而且在组装完毕后与之分离,不构成这些蛋白质结构执行功能时的组份
?CD8+T细胞是针对MHC-I型的,CD4+T细胞是针对MHC-II型
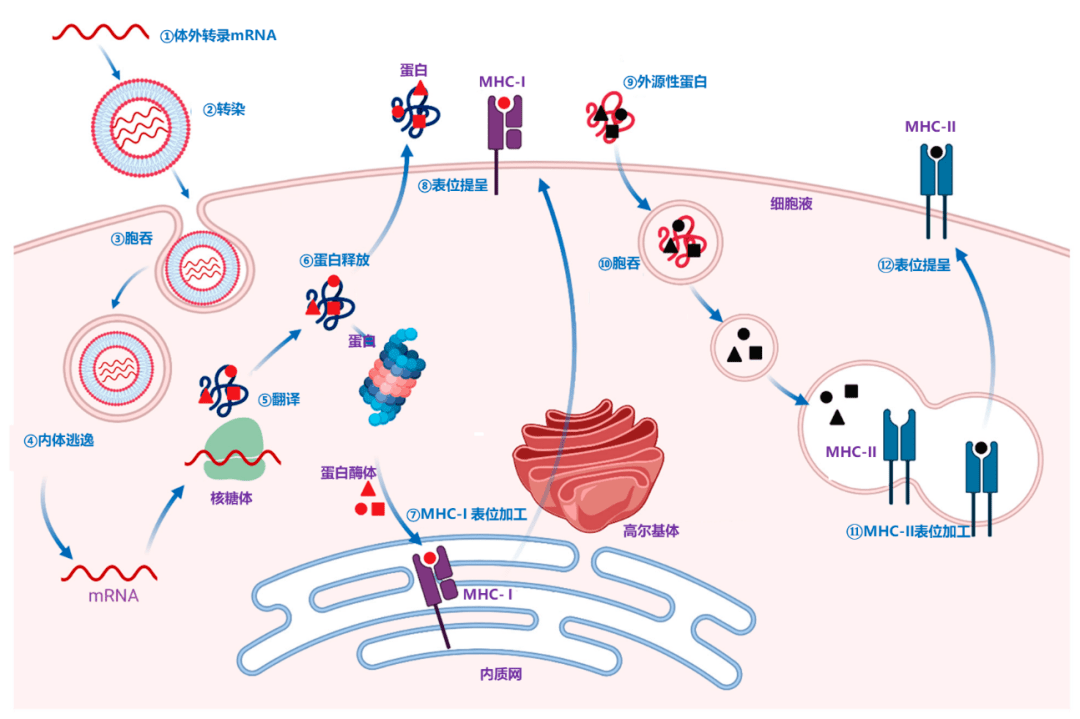
?下图是MHC-I型和MHC-II型表位呈递的过程
3、Results
3.1、MAPTAC可以实现单个HLA-II等位基因的可扩展性分析
目前对 HLA-II 结合基序的了解主要基于使用生化结合分析生成的数据(Justesen 等人,2009;Sidney 等人,2001,2013),这些数据汇编在免疫表位数据库(IEDB)中(Vita 等人) al., 2019) 并用于训练 HLA-II 预测算法,例如 NetMHCIIpan (Andreatta et al., 2015)。 五个最常见的欧洲 HLADRB1 等位基因在 IEDB 中得到了很好的支持(每个 3,326-8,967 个肽段),尽管其中只有约 29% 是强结合剂(亲和力 <100 nM),并且 85% 的 IEDB 肽段总体上是精确的 15 长度( 图 S1A 和 S1B)。 支持 HLA-DP、HLA-DQ 和非欧洲 HLA-DR 等位基因(例如,HLA-DRB1*15:02)的数据要少得多。
这一部分是在IEDB中保存的实验获得的数据,这一部分比较少,阳性比较少,而且85%都是15长度的
为了创建具有等位基因广度的高质量数据集,本文开发了 MAPTAC,该技术能够有效分离结合单个等位基因的 HLA-II 肽,用于基于 MS 的鉴定(图 1A)。 所选择的 HLA-II 异二聚体的 α?和 β链由基因构建体编码,该基因构建体在 β 链的 C 末端具有生物素受体肽 (BAP) 序列(Yang 等,2004)。 因为 HLA-DRA 在功能上是不变的,MAPTAC 产生了单等位基因 HLA-DR 结果,而不管外源 β链和内源 α 链之间的潜在配对。
下图A表示MAPTAC的整体流程:其中plasmid表示质粒
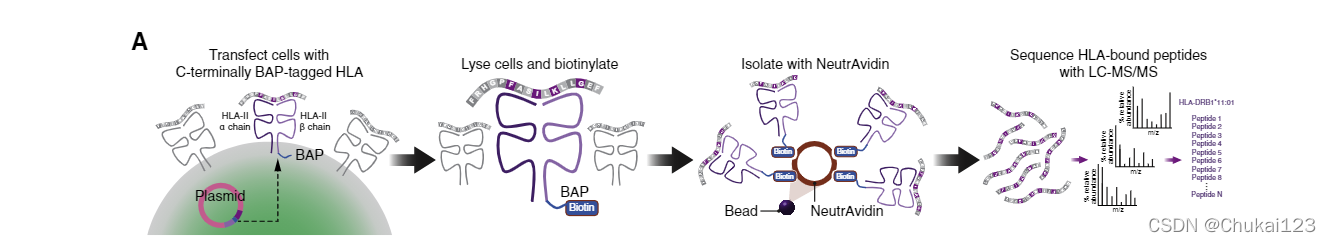
图1A.用 C 末端 BAP 标记的 HLA 转染细胞,并用裂解细胞和生物素进行处理,再用 NeutrAvidin 分离,最后使用 LC-MS/MS 对 HLA 结合肽进行测序;
注:Biotin表示生物素
?LC-MS/MS原理:
1906年Thomson发明了质谱技术,1989年第一台商业化液相色谱串联质谱仪问世。经过百年的发展,液相色谱串联质谱(LC-MS/MS)越来越广泛应用于药物、食品、环境、法医、临床等各个领域。液相色谱串联质谱中,液相色谱负责分离待测物与干扰物,质谱负责检测。样品进样后首先在流动相的携带下进入色谱柱,经过色谱柱分离后,进入质谱进行检测。质谱根据被测物的质荷比(m/z)进行检测,被测物在离子源转换成气相离子进入质谱,在三重四级杆中一级质谱扫描特定范围离子或允许特定离子进入碰撞室,在碰撞室内分子离子碰撞裂解,形成子离子进入二级质谱,二级质谱扫描特定范围离子或允许特定离子进入检测器。LC-MS/MS具有灵敏度高,选择性强,准确性好等特点,在临床检测上适用范围远远超过放射性免疫检测和化学检测范围,是其他方法无可比拟的。
此处参考链接:https://www.biomart.cn/specials/waters/article/91561经过质谱仪分析后:横坐标表示质荷比,纵坐标表示相对丰度(元素中同位素的百分比数)
下图1B表示生成的相关数据:
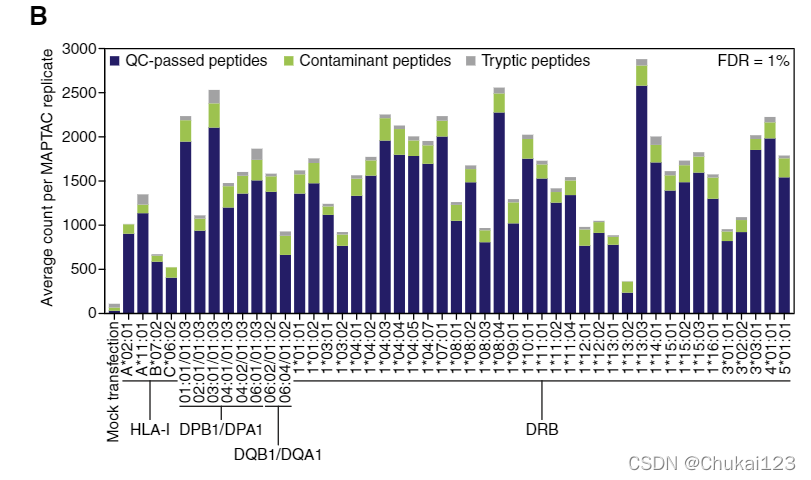
?图B的横坐标表示不同的分型allele,纵坐标表示每个 MAPTAC 复制的平均计数,其中包括QC-passed肽,污染肽和胰蛋白肽,采用的FDR=1%
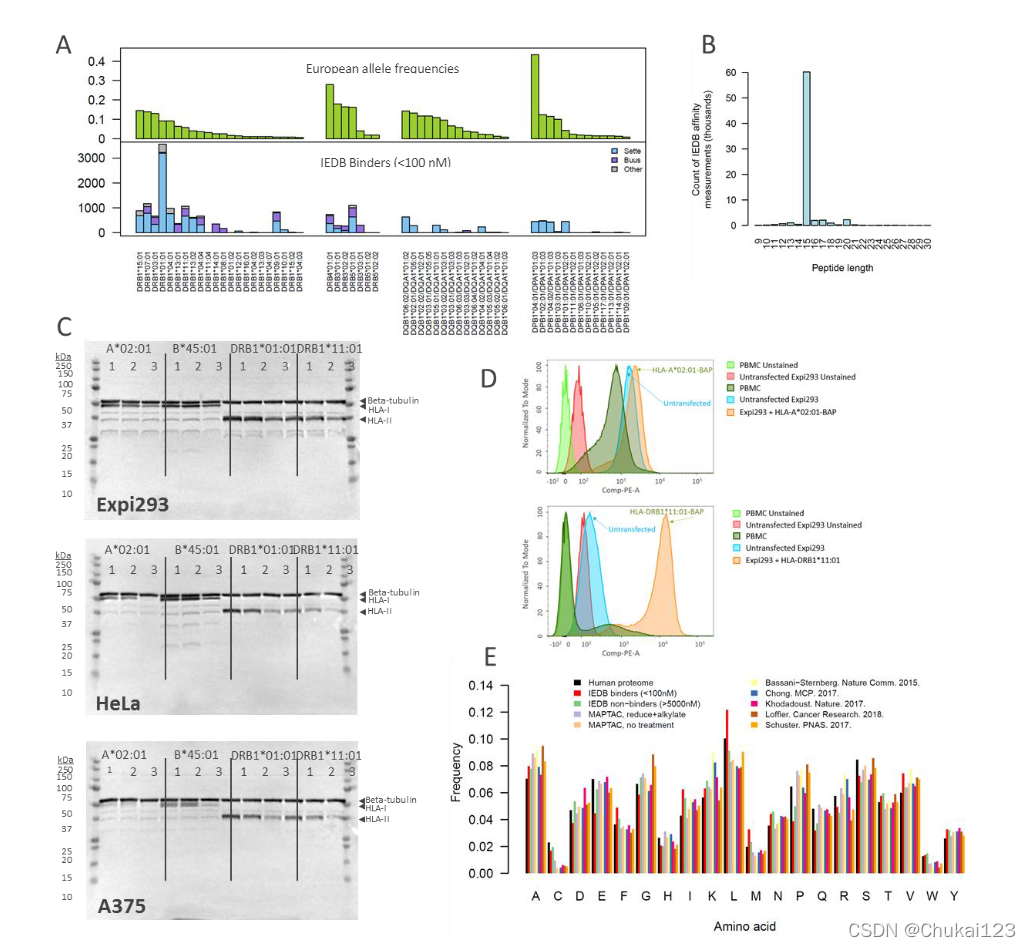
同样文中也提出了与IEDB的比较:(附件中的图)
图S1A表示IEDB中欧洲分型的频率分布,图S1B表示IEDB中HLA-II型中不同长度的peptide的亲和力数量,图S1C表示转染方案的比较,图S1D表示HLA细胞表面分析,图S1E表示为MAPTAC和IEDB观察等方案的氨基酸频率。
图S1 A-E
?图1C表示选择HLA-I和MAPTAC分布的HLA-II等位基因的肽长度分布。可以看到HLA-I型9长度居多,而MAPTAC生成的HLA-II型肽主要分布在15~20长度。
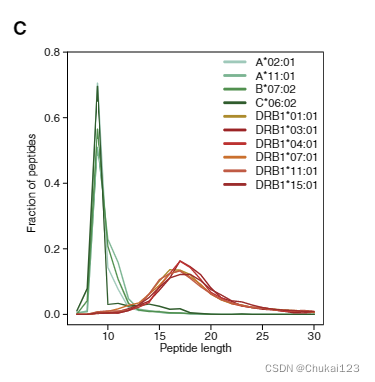
?经过48 小时转染实现了 MAPTAC 构建体(图 S1C)和细胞表面呈现(图 S1D)的稳健表达。这在 6 种不同的癌症和 APC 细胞系(Expi293、HEK293、A375、KG-1、K562 和 B721.221)和超过 40 个 HLA-II 等位基因中得到证实,为所有典型 HLA-I 基因座(HLA -A、-B 和 -C)和 HLA-II 基因座(HLA-DR;数据 S1A 和 S1B)。通过质量控制过滤器的每个重复(5000 万个细胞)的独特肽识别的平均数量范围为 236 到 2,580 个等位基因(图 1B),中位数为 1,319 个肽。采用了几种工艺变化来增加数据深度,包括 HLA-DM 过表达、肽还原和烷基化。只有一小部分 MS 命中对应于已知污染物、胰蛋白酶肽和模拟转染(空质粒;图 1B)。 MAPTAC HLA-I 和 HLA-II 肽的长度分布与之前使用基于抗体的下拉的 MS 研究中观察到的一致(图 1C;Bassani-Sternberg 等人,2016)。
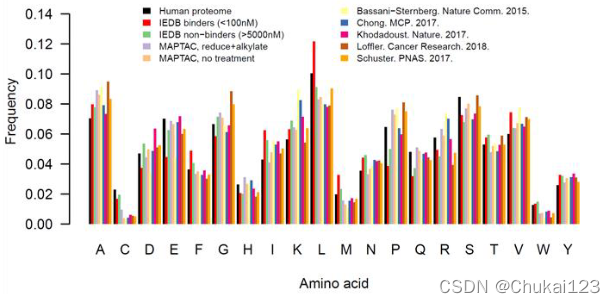
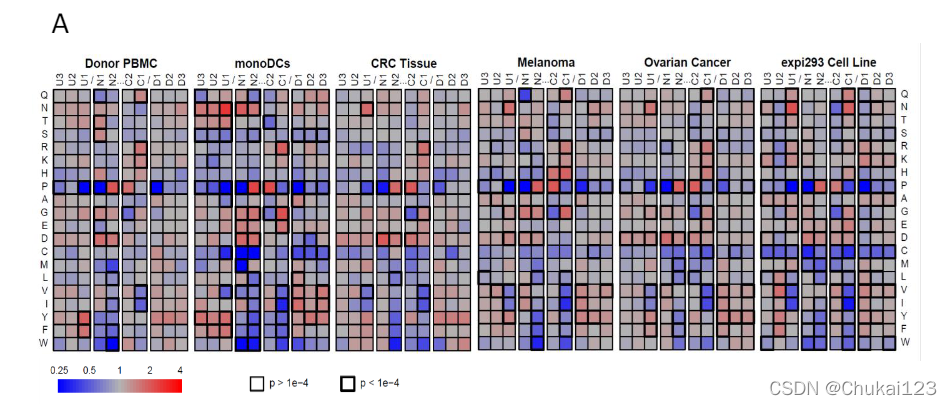
在 MAPTAC HLA-II 肽中,大多数氨基酸以与源蛋白质组频率一致的速率表示(图 S1E)。 例外情况包括 C、M 和 W,它们分别减少了 85%、34% 和 42%,这与之前基于 MS 的 HLA-II 肽研究一致(BassaniSternberg 等人,2016;Chong 等人, 2018 年;Khodadoust 等人,2017 年;Lo ? ffler 等人,2018 年;Schuster 等人,2017 年)。 HLA-II 肽的还原和烷基化(STAR 方法)几乎使 C 的频率增加了三倍,尽管它在蛋白质组方面的代表性仍然不足。 在来自 IEDB 的等位基因匹配的高亲和力肽 (<100 nM) 中未观察到 C、M 和 W 的消耗。 相反,与 IEDB 非粘合剂 (>5,000 nM) 相比,IEDB 粘合剂表现出 D (39%) 和 E (37%) 的消耗以及 M (+65%) 的富集。 因此,MAPTAC 表现出与其他技术观察到的偏差一致的明确偏差。
3.2、MAPTAC 解析 HLA-II 肽结合基序
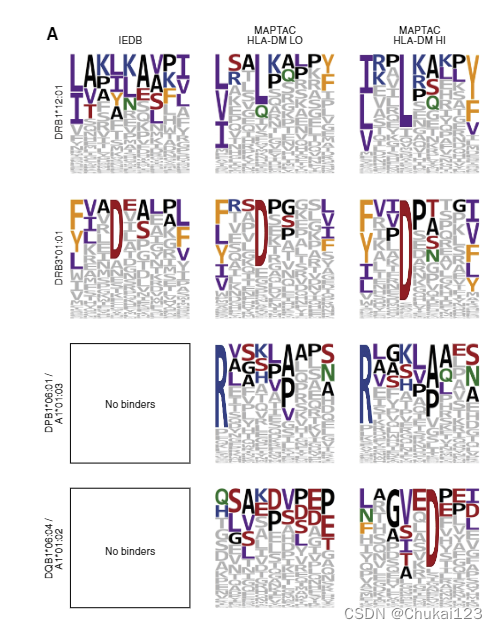
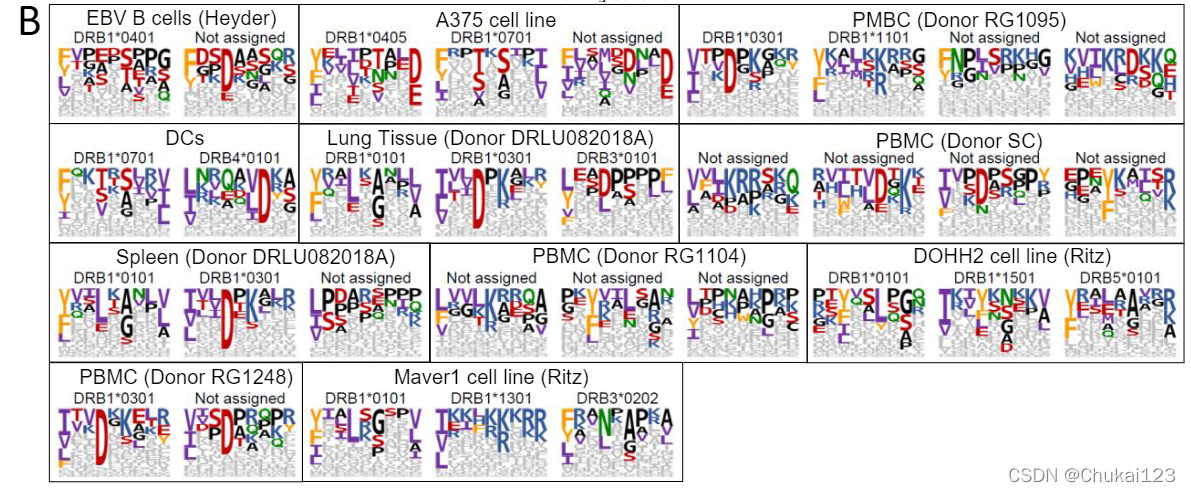
使用 MAPTAC 来解析等位基因特异性 HLA-II 结合基序,其中 15 个以前未被表征(在 IEDB 中 <30 个具有 <100 nM 亲和力的肽),包括非欧洲人群中常见的等位基因(DRB1*12:02,DRB1* 15:03 和 DRB1*04:07)。 对于先前表征的 HLA-II 等位基因,基序(由 GibbsCluster 解决;Andreatta 等人,2017 年)与 IEDB 密切匹配(下图 S2)。
?对于 HLA-DP 和 -DQ 等位基因,其特征通常不太好,我们注意到非规范模式,包括 P1 中非疏水残基的存在。 MAPTAC 观察到的肽段并不总是显示常见等位基因的 NetMHCIIpan 得分很高(图 S3A); 图S3A中绿色表示MAPTAC,蓝色表示从蛋白质组中随机取样的。
?然而,NetMHCIIpan 预测不佳的观察到的结合剂显示出具有非常强的测量亲和力(图 S3B),表明这些观察结果不太可能是假阳性。
?值得注意的是,MAPTAC 基序在多个细胞系中始终是稳定的(图 S3C)

?通常,MAPTAC 和 IEDB 就锚定位置的最高频率氨基酸(4 个最高度保守的位置)达成一致,但 MAPTAC 基序通常显示较低的熵(表现为序列徽标中较高的字母高度)。当我们用 MAPTAC 构建体和 HLADM 共转染细胞时,大多数等位基因的锚定位置的熵进一步降低(图 2A 和 S4A)。这在 12 个 HLA-DR 等位基因中始终如一地观察到,显示 HLA-DM 作为曲目“编辑器”的普遍作用(van Lith 等人,2010),并表明基于缺乏 HLA-DM 和其他负载的亲和力测定的模型伴侣可能会学习在体内不适用的绑定规则。 HLA-DQ 似乎也对 HLA-DM 高度敏感,以至于我们在没有 HLA-DM 共转染的情况下难以辨别基序。相比之下,我们分析的六个 HLA-DP 等位基因中没有一个对 HLA-DM 敏感,无论 P1 锚是疏水性的还是碱性的(van Lith 等人,2010)。
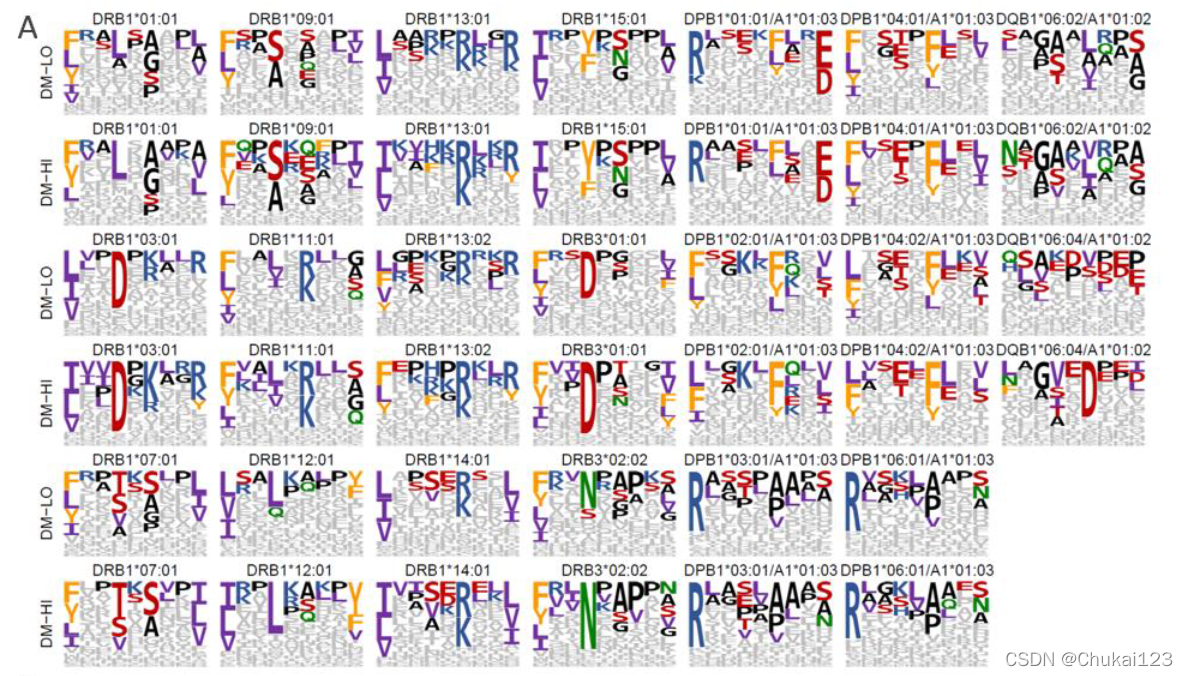
?HLA-DM 的作用对于 II 类相关不变链肽 (CLIP) 的存在也很明显。在没有 HLA-DM 共转染的情况下,观察到 9 个 HLA-DR 等位基因和匹配的已知 CLIP 变体的 CD74 衍生肽(图 S4B;Bergseng 等人,2015;Costantino 等人,2012;Schr?der,2016);同时,在我们的任何 HLA-DM 共转染实验中均未观察到 CLIP 肽。

?鉴于已发表的多等位基因 HLA-II 数据集的可用性,我们研究了是否可以使用计算机解卷积方法有效地鉴定我们的等位基因特异性肽。几个小组已经成功地从多等位基因 HLA-I 数据中解卷积 HLA-I 等位基因基序(Alvarez 等人,2018;BassaniSternberg 和 Gfeller,2016;Chong 等人,2018;Nielsen 和 Lund,2009);然而,由于需要同时解决每个肽的结合核心和等位基因分配,HLA-II 基序的去卷积变得复杂。为了评估 HLA-II 去卷积的准确性,我们分析了 11 个由 pan-DR 抗体分析的样本中的 HLA-DR 配体组(data?S1A 和 S1B)。
https://www.cell.com/immunity/fulltext/S1074-7613(19)30363-2?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS1074761319303632%3Fshowall%3Dtrue#supplementaryMaterial![]() https://www.cell.com/immunity/fulltext/S1074-7613(19)30363-2?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS1074761319303632%3Fshowall%3Dtrue#supplementaryMaterial对于每个数据集,我们计算加标了单等位基因数
https://www.cell.com/immunity/fulltext/S1074-7613(19)30363-2?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS1074761319303632%3Fshowall%3Dtrue#supplementaryMaterial对于每个数据集,我们计算加标了单等位基因数
据的 20 个肽段,匹配样本基因型中的每个等位基因(1 或 2 个 DR1 等位基因加上 0-2 个 DR3/4/5 等位基因,取决于单倍型和接合性;data?S2).我们使用 GibbsCluster(也可用于反卷积;Andreatta 等人,2017 年)将肽分成组,并观察尖峰肽是否根据其已知的起源等位基因适当地共聚。在所有情况下,肽分布在不同的簇中,仅显示与正确源等位基因的适度关联(图 2B),并表明基于反卷积的 HLA-II 训练数据可能带有不可忽略的错误。
GibbsCluster:检测蛋白质短线性基序
生信在线工具
https://www.jianshu.com/p/765ebc6f85f1?clicktime=1578141219GibbsCluster-2.0在线版本
https://services.healthtech.dtu.dk/service.php?GibbsCluster-2.0

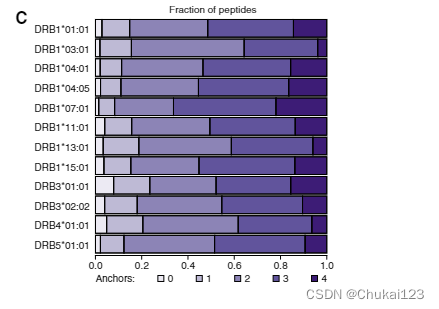
?要了解反卷积的糟糕性能,我们审查了我们的单位等位基因MAPTAC数据,以确定“显而易见”锚的频率,可作为GIBBSCluster的指南。因此,我们将每个HLA-II等位基因的每个锚定位置(具有最低熵的四个位置)定义明显的氨基酸(具有频率> 10%的那些)。只有 10%–20% 的肽在所有四个锚点位置都表现出理想的残基,而多达 50% 的肽表现出两个或更少的明显锚点(图 2C)。 鉴于显示大多数预期锚的肽的频率较低,因此大部分肽很难在纯粹的计算基础上进行分类也就不足为奇了。 因此,MAPTAC 解决了一个主要的歧义来源,这是用计算机方法解决的重要问题。
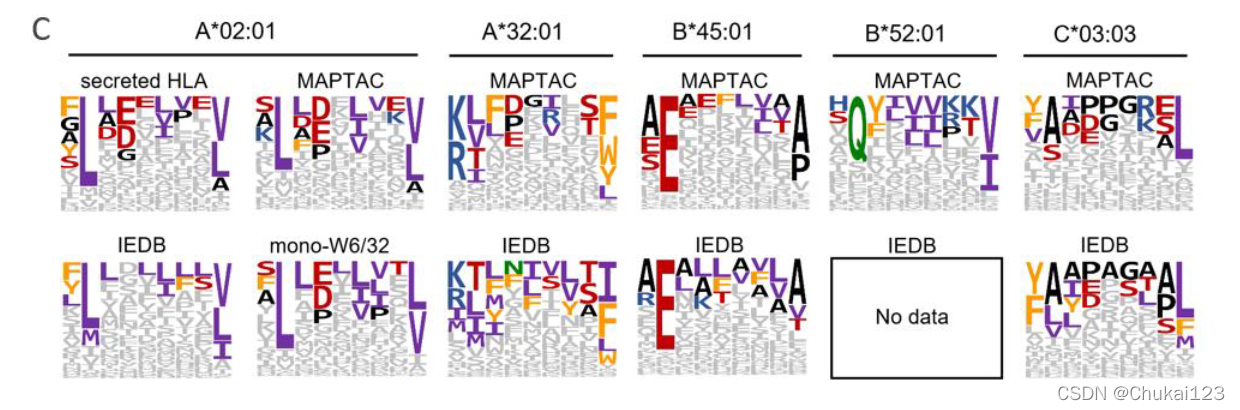
?虽然不是本文的重点,我们还能够使用 MAPTAC 定义 HLA-I 等位基因的基序。 这包括以前未定义结合谱的等位基因(例如,B*52:01,在日本很常见)。对于以前表征的等位基因,我们看到来自基于亲和力的方法和以前的单等位基因 MS 研究的基序有很好的对应关系。 尽管如此,我们注意到在多等位基因、基于 MS 的研究方面存在一些差异,这些研究采用反卷积方法来定义基序。(Figure S4C; Bulik-Sullivan et al., 2018; Chong et al., 2018).
?3.3、在MAPTAC数据上训练的算法预测免疫原性
我们考虑了 MAPTAC 数据是否可以生成具有更高准确性的 HLA-II 结合预测因子。 由于 HLA-II 肽的结合核心相对于 N 或 C 末端不在固定位置,因此算法必须动态考虑每个肽的不同潜在结合核心。 为了解决这个限制,我们采用了卷积神经网络 (CNN),它在计算机视觉领域取得了成功,因为它们精通平移不变模式识别 (LeCun et al., 1989)。 对于每个等位基因,我们训练了一组 CNN(图 3A;STAR 方法),将整体预测器称为“neonmhc2”。
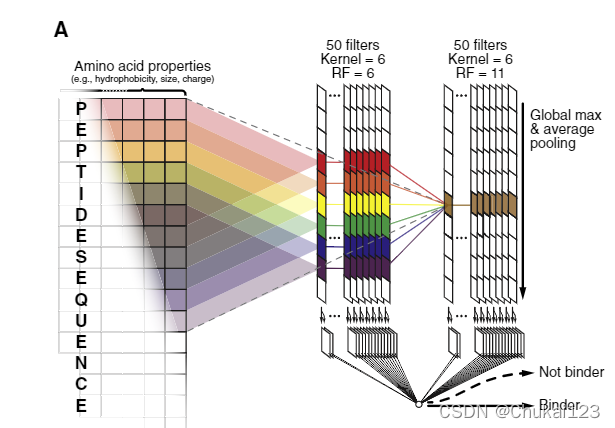
这里是针对每一个分型采用CNN训练了一个模型,将所有统称为“neonmhc2”
考虑到 MS 表现出某种程度的氨基酸残基偏差这一事实,特别是针对 C(图 S1E),我们通过随机排列观察到的结合物(“hits”)的序列来生成负训练示例(“decoys”) )。由于这种方法存在学习天然蛋白质序列特性的风险,因此在评估模型性能时,阴性样本是从 HLA-II 配体源基因的未观察到的子序列中随机抽样的。
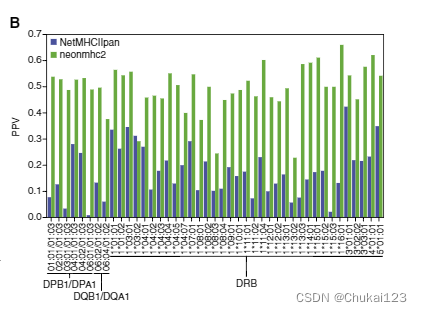
为了计算每个等位基因的阳性预测值 (PPV),n 个 MS 观察到的肽与从同一组源基因中采样的 19n 个长度匹配的阴性样本以及每个预测因子的 n 个排名靠前的肽(即前 5 %) 被称为阳性。在这种情况下,PPV 与召回相同,因为误报的数量和漏报的数量相等。以 1:19 的阳性:阴性比例计算 PPV 表明,nenmhc2 在预测 MAPTAC 观察到的肽方面相对于 NetMHCIIpan 改进了 PPV(图 3B)。
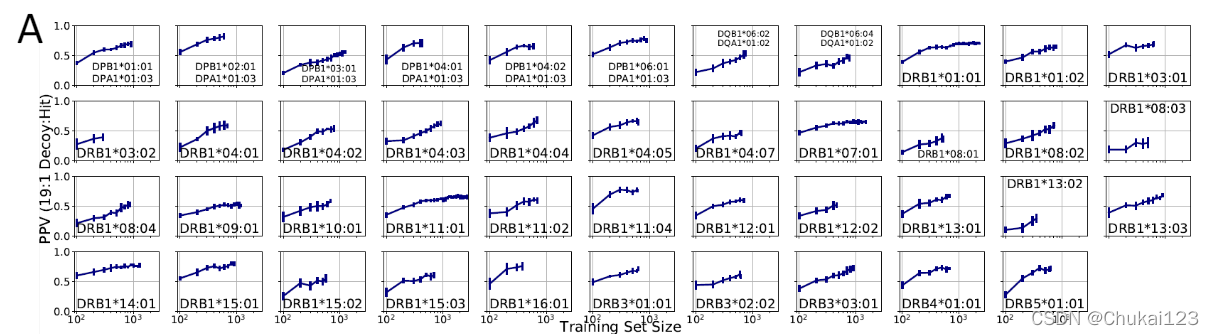
?饱和实验,其中训练数据集大小在不同程度上被下采样,表明 neonmhc2 的性能可能会随着数据的增加而提高(图 S5A)。
?我们的分析显示 HLA-II 反卷积的低保真度(图 2B)表明,如果没有单等位基因数据,就无法实现可比的预测性能。为了测试这一点,我们遵循了最近发布的计算工作流程,该工作流程使用反卷积在多等位基因 MS 数据上训练等位基因特异性结合预测因子 (Barra et al., 2018)。
检查 11 个泛 DR 样本的 GibbsCluster 徽标(与图 2B 相同),我们观察到许多簇(13/32)与样本基因型中的等位基因没有任何相似之处(图 S5B)。使用预先存在的关于主题应该是什么样子的知识,我们只选择了合法的集群(在图 S5B 中标记)并使用我们相同的 CNN 架构构建了预测器。然后,这些模型与 neonmhc2 一起在真正的单等位基因数据(未用于训练的 MAPTAC 数据的保留分区)上进行评估。
在反卷积多等位基因数据上训练的模型通常超过 NetMHCIIpan,但通常不如 MAPTAC 训练的 neonmhc2(图 3C)。即使我们对 MAPTAC 训练数据集进行下采样以匹配较小尺寸的反卷积训练数据集,单等位基因数据的优势也得以保持。
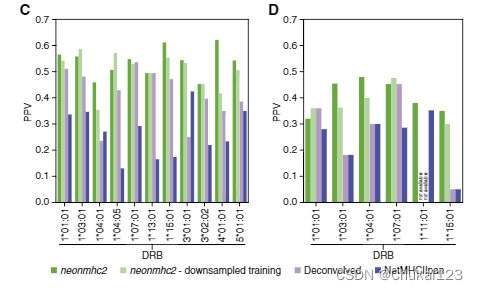
为了确保我们的预测改进在对非 MS 数据进行评估时能够保持,我们策划了一个大型数据集,其中包含通过四聚体引导表位映射 (TGEM) 检测到的等位基因特异性 CD4+ 记忆 T 细胞反应(Archila 和 Kwok,2017;Vita 等人,2019;data?S3)。值得注意的是,四聚体数据依赖于无伴侣的肽交换,因此它们可能会受到与传统亲和力测定相同的偏差(Archila 和 Kwok,2017)。尽管如此,对于所有等位基因,neonmhc2 的表现都优于 NetMHCIIpan,具有足够的评估数据(至少 20 个阳性例子;图 3D)。 NetMHCIIpan 的性能参差不齐,DRB1*15:01 的 PPV 低至 5%(neonmhc2 的性能从未低于 30% PPV)??,并且仅在两个等位基因上接近 neonmhc2,包括经过充分研究的 DRB1*01: 01.另一方面,neonmhc2 在所有其他评估的等位基因上显示出改善,包括两个最常见的欧洲 HLA-DR 等位基因(DRB1*07:01 和 DRB1*15:01)。在基于反卷积的预测器的结果中分层,nenmhc2 在大多数情况下具有明显更好的性能,即使我们将 MAPTAC 训练集下采样到相同大小。这些结果表明neonmhc2 的预测改进可以在基于非 MS 的基准中得到验证,并且可能扩展到大多数等位基因。
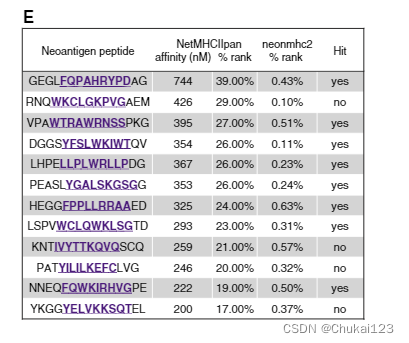
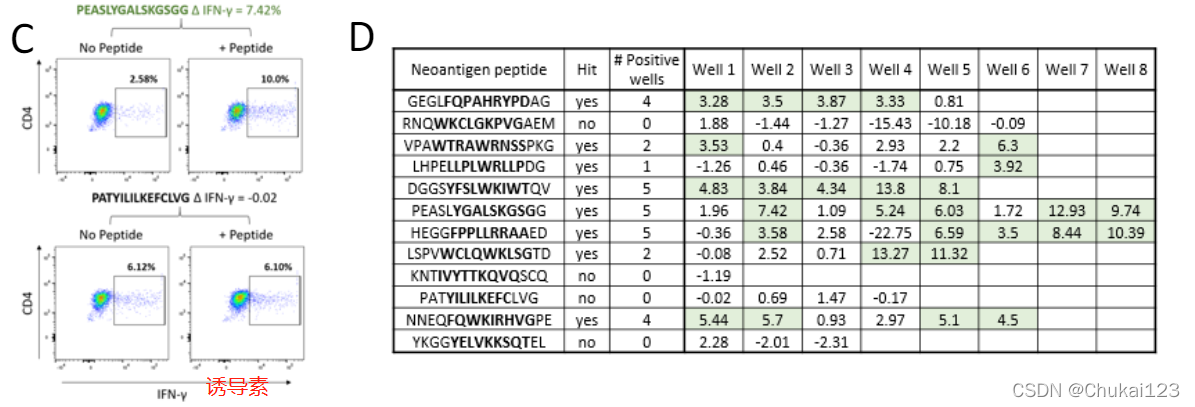
为了评估 neonmhc2 的治疗相关性,我们试图确定 neonmhc2 是否可以在离体诱导试验中鉴定能够引发 CD4+ T 细胞反应的新抗原。专注于 DRB1*11:01,这是一个常见的等位基因,在 IEDB 中具有许多亲和分析证实的结合物(仅被 DRB1*01:01 和 DRB1*07:01 超越;图 S1A),我们对一组癌症基因组图谱进行了评分(TCGA) 观察到新抗原序列并选择了 neonmhc2 首选的子集(预测的前 1%)但 NetMHCIIpan 未选择的子集(预测的下 90%)。通过去除可能与诱导材料中存在的其他 HLA-DR 等位基因结合的肽,进一步完善了该组。大多数 neonmhc2 选择的肽 (8/12) 产生 CD4+ T 细胞反应,通过干扰素 (IFN)-g 表达测量,以响应肽的回忆(图 3E、S5C 和 S5D)。这些结果表明,我们经过 MAPTAC 训练的 neonmhc2 可以识别 NetMHCIIpan 未识别的免疫原性 HLA-II 新抗原序列。
?3.4、专业 APC 是肿瘤微环境中的主要 HLA-II 呈现者
为了研究 TME (肿瘤微环境)环境下的抗原加工,我们分析了非 MAPTAC 数据集,包括单细胞 RNA 测序 (scRNA-seq)。 我们最初考虑了 TME 中的哪些细胞类型最有可能呈递可治疗靶向的抗原。 目前,对于癌症抗原是由具有内吞肿瘤蛋白的专业 APC 还是由肿瘤细胞本身呈递,尚无共识(Deniger 等人,2018;Johnson 等人,2016;Tran 等人,2014)。 为此,我们分析了 scRNA-seq 数据集中的 HLA-DRB1 表达,这些数据集描述了肺癌、头颈癌、结直肠癌和卵巢癌(Lambrechts 等人,2018;Puram 等人,2017;Cancer Genome Atlas Network,2012) 并发现典型的 APC(巨噬细胞、树突细胞和 B 细胞)比 TME 中的肿瘤细胞和其他基质细胞类型表达更多的 HLA-II。 这一观察结果在多个患者和肿瘤类型中是一致的(图 4A)。
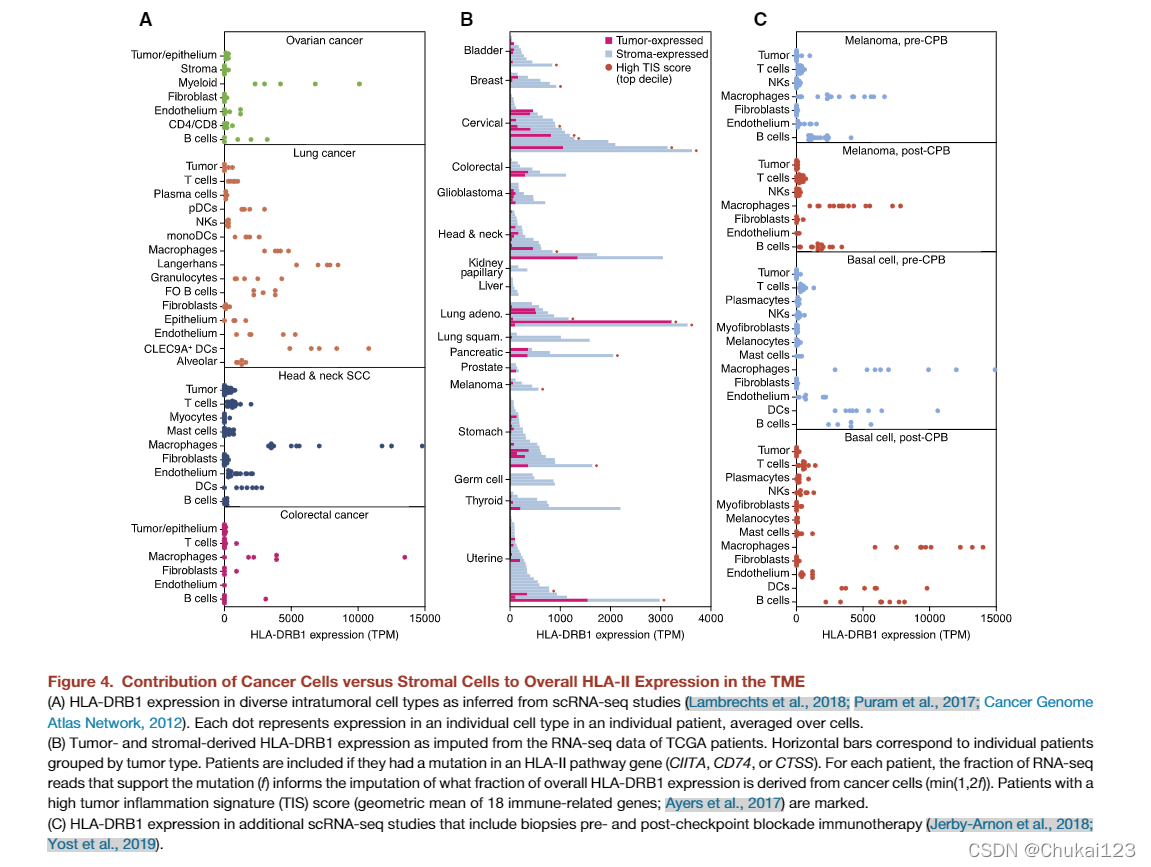
?由于肿瘤细胞在 TME 中的数量可能超过 APC,因此它们较低的 HLA-II 表达可能与免疫学相关。为了评估总体 HLA-II 表达中有多少来自肿瘤细胞和基质,我们确定了具有 HLA-II 特异性基因(CIITA、CD74 和 CTSS)突变的 TCGA 患者,并确定了 RNA-seq 读数的哪一部分表现出 体细胞变异来估算肿瘤衍生的 HLA-DRB1 表达的分数(图 4B)。 基于在代表 17 种不同肿瘤类型的 153 名患者中发现的突变,大多数 HLA-II 表达似乎来自非肿瘤细胞。 事实上,45% 的患者表现出零肿瘤来源的 HLA-II 表达。 专注于 T 细胞浸润最高的患者(前 10%,使用已发表的 18 基因特征确定;Ayers 等人,2017),低肿瘤 HLA-DR 表达似乎仍然是常态,16 人中只有 3 人 患者每百万表达> 1000个转录本(TPM)。
为了探究免疫疗法是否破坏了这一趋势,我们分析了来自检查点阻断反应性肿瘤类型的额外 scRNA-seq,并评估了治疗前后 HLA-DRB1 的表达。 一个确认有反应者的黑色素瘤队列(Jerby-Arnon 等人,2018 年)在治疗前和治疗后活检中均显示肿瘤细胞的 HLA-DRB1 表达均较低(图 4C)。 同样,对抗 PD-1 治疗有 55% 临床反应率的基底细胞癌队列(Yost 等人,2019 年)表现出低肿瘤细胞衍生的 HLADRB1 表达,无论时间点如何(图 4C)。 这些结果表明,大多数肿瘤内 HLA-II 呈递是由专业 APC 驱动的,即使在免疫学“hot”的 TME 中也是如此。
3.5、特定基因有权访问 HLA-II 呈递途径
我们接下来试图通过分析三个肿瘤 HLA-II 配体组研究来确定优先由肿瘤驻留 APC 呈递的配体的源基因,以及它们是否来自自噬或内吞作用(Bassani-Sternberg 等人,2016;Lo ffler 等人) .,2018 年;舒斯特等人,2017 年)。
首先,我们量化了每个基因在肿瘤 HLA-II 配体组中的表现程度,假设观察次数应与其长度和表达的乘积成正比(图 5A)。
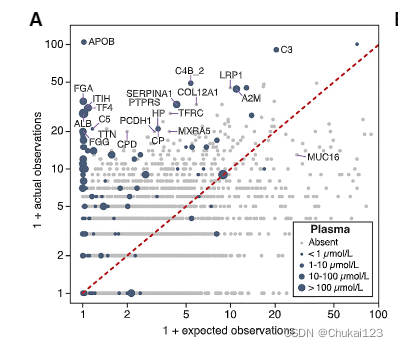
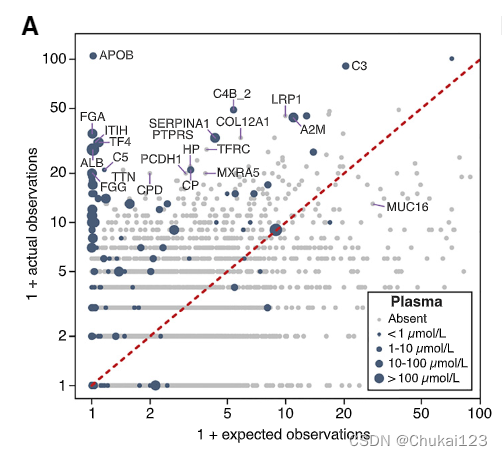
我们观察到人血浆中表达的蛋白质明显富集,尤其是白蛋白、纤维蛋白原、补体因子、载脂蛋白和转铁蛋白,尽管它们并未在天然组织中表达。考虑到这些鉴定可能代表非特异性结合,我们评估了四个外周血单核细胞 (PBMC) HLA-DR 配体组中血浆衍生肽的 neonmhc2 结合评分(图 S6A);这些肽显示出很强的结合分数,表明它们是 HLA 结合的。血浆衍生蛋白在肿瘤 HLA-I 配体组数据中未富集(图 S6B)。血浆基因的富集与 APC 从组织血清中“吸取”细胞外蛋白一致(Bozzacco et al., 2011; Mommen et al., 2016)。我们还观察到参与白细胞细胞粘附的基因富集,例如 ITGAM(11 倍富集)、LCP1(8 倍)、ITGAV(6 倍)和 ICAM1(6 倍),表明 APC 正在积极回收他们自己的膜(Rock et al., 2016)。据报道,MUC16 富含卵巢癌 HLA-I 配体组(Schuster 等人,2017 年),但并没有被过多代表。
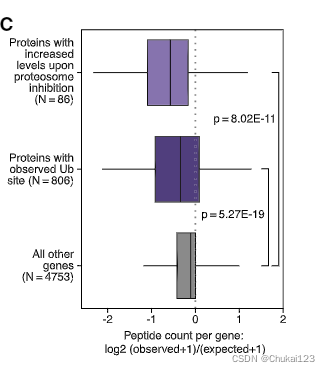
?细胞定位也被认为是进一步询问优先处理的基因类别。当基因按定位分组时,分泌基因和膜基因的表达频率是基于基因表达的预期的两倍,这强调了巨胞饮作用在塑造 HLA-II 配体组中的重要作用。尽管如此,超过一半的 HLA-II 肽对应于与巨胞饮作用不一致的隔室,例如细胞核和细胞质。我们推断,如果这些基因中的许多是通过自噬呈现的,那么应该存在相应的已知被蛋白酶体清除的基因缺陷。事实上,已知含有泛素位点的蛋白质(Kro ? nke 等人,2014 年,2015 年;Udeshi 等人,2012 年)根据其长度和表达方式生成肽的频率低于预期(图 5C)。还观察到在蛋白酶体抑制后增加丰度的蛋白质的消耗(Rose 等人,2016)。这些是自噬的预期模式,但不一定是吞噬作用,这表明 APC 配体组部分代表它们自己的细胞内蛋白质组。
?为了解决 TME 中 APC 呈递的 HLA-II 抗原的起源,我们考虑是否有可能通过确定核和细胞溶质肽鉴定是否与 APC 特异性或大量肽更一致来直接解卷积源基因的起源肿瘤基因表达谱(图 S6C)。 尽管我们的估计(STAR 方法)存在不确定性,但 HLA-II 配体组通常最好用肿瘤和 APC 基因表达谱的混合来解释。 结合观察到的蛋白酶体清除蛋白的消耗,该结果表明肿瘤内 APC 呈现外源性和内源性蛋白的混合物。
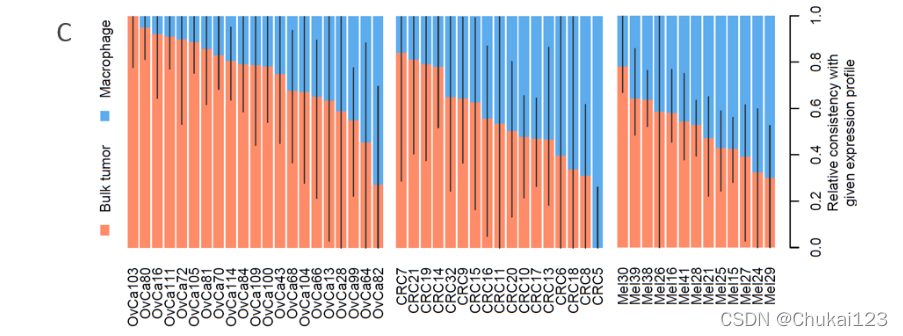
?3.6、一些基因区域被优先加工但缺乏明显的切割基序
因为加工预测改进了 HLA-I 表位的识别 (Abelin et al., 2017),我们试图为 HLA-II 构建一个类似的加工预测器。 事实上,HLA-II 表位在其 N 和 C 末端附近显示出强烈的氨基酸富集,这在细胞类型和组织中是保守的(图 S7A)。 因为对于 HLA-II 肽的生成方式存在竞争模型(Sercarz 和 Maverakis,2003),我们研究了三种不同的预测框架(图 5D)。
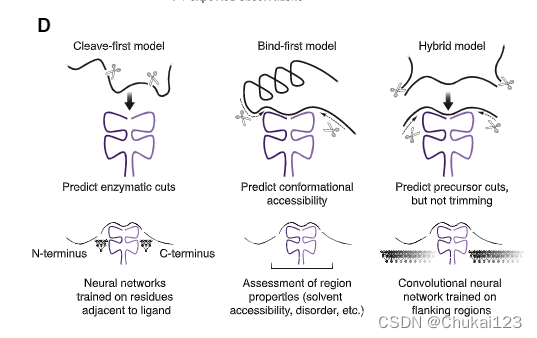
第一个假设内肽酶占主导地位(“先切割”); 第二个模型假设 HLA-II 与随后被外肽酶向内修剪的全长蛋白质结合(“首先结合”); 第三种模型表明酶消化发生在 HLA 结合之前和之后(“混合”)。 每个模型都需要不同的算法方法。 具体来说,由切割优先观点推动的算法应该关注 MS 观察到的配体边缘的氨基酸基序(Paul 等人,2018 年); 然而,由绑定优先观点驱动的算法会更好地忽略这些基序并专注于决定 HLA 结合可及性的局部蛋白质结构特性。 受混合模型启发的算法应该在观察到的 HLA-II 肽的上游和下游寻找候选前体切割位点。
在考虑的三种方法中,只有 cleave-first 算法对基线模型产生了可测量的改进(图 5E 和 S7B)。 然而,这种方法似乎通过学习阳性示例肽中存在的外肽酶修剪标志(例如倒数第二个脯氨酸特征;Barra 等人,2018 年)来“欺骗”,因为如果精确切割它无法增加价值 查询肽的位点被掩盖(STAR 方法)。
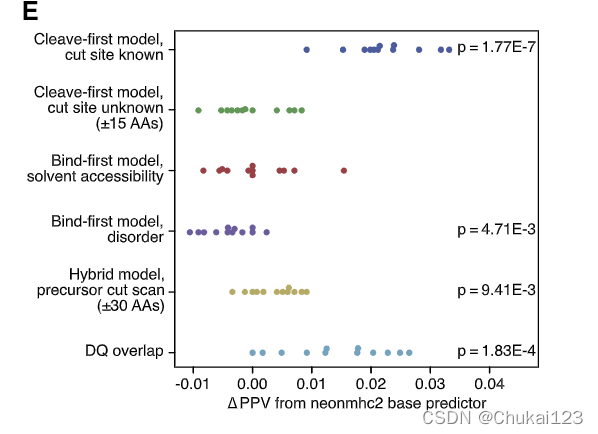
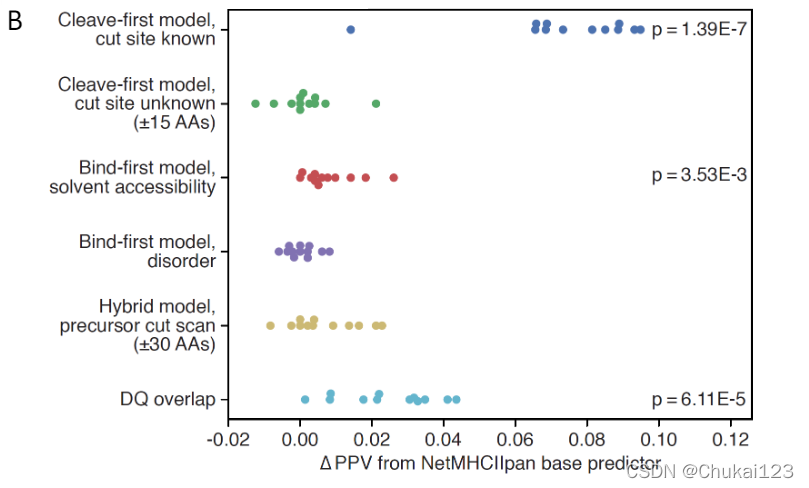
?转向纯粹的经验方法,我们对已发表的 HLA-DQ 配体组中观察到的蛋白质区域进行分类(Bergseng 等人,2015),并使用重叠来预测 HLA-DR 配体。 重叠变量对预测性能产生了适度的改进(PPV 平均比单独的 neonmhc2 增加 3.1%;图 5E)。 假设 HLA-DQ 和 HLA-DR 等位基因共享相同的 HLA-II 加工环境但不共享结合基序,该结果表明某些基因区域确实有利于加工,但与切割基序或构象特性没有明显的联系。
3.7、集成表示规则极大地增强了 HLADR 配体组预测
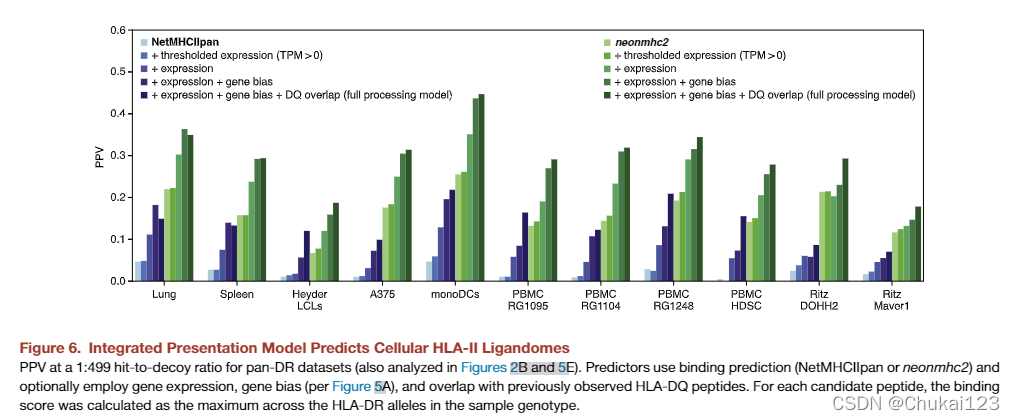
为了量化结合规则如何与加工相关特征协同作用,我们创建了多变量模型 (Hoof et al., 2012),用于预测 HLA-II 呈递细胞系、原代组织、树突状细胞 (DC) 和健康的 HLA-DR 配体组。供体 PBMC。尽管所呈现的肽没有发生突变,但预测方案模拟了新抗原预测,其中必须评估随机采样的基因组位点产生 HLA-II 肽的能力。使用 1:499 的hit-to-decoy比和从蛋白质编码外显子组中随机采样decoy,我们评估了基于 neonmhc2 和 NetMHCIIpan 的模型的性能,以及包含额外处理特征的模型,包括 RNA-seq 衍生模型表达、基因偏差(根据图 5A)以及与先前观察到的 HLA-DQ 肽重叠。为了使模型与我们可能优先考虑免疫治疗的新表位靶点的方式一致,我们修改了基因偏倚特征以中和对与新抗原无关来源的血浆基因的偏好。
这些综合算法证实了绑定和处理预测的实质性改进(图 6)。 具体来说,我们的完整模型显示出比仅使用 NetMHCIIpan 结合预测的模型提高了 7.4-61 倍的变化,具体取决于正在评估的数据集。 表达和基因偏倚都为预测准确性提供了重要的独立贡献。 DQ 重叠功能进行了较小但始终如一的积极改进。重要的是,基于亲和力的模型的准确度只有基于 MAPTAC 的模型的一半,即使提供了与处理相关的预测变量的全部好处。
?3.8、模型可以预测由专业 APC 提供的肿瘤衍生 HLA-II 肽
在评估了我们预测 HLA-II 配体组的准确性之后,我们转向测试我们是否可以预测由专业 APC 提供的肿瘤衍生配体。 我们观察到 TME 中的大多数 HLA-II 表达来自专业 APC,这表明这可能是肿瘤抗原呈递最相关的途径。 不幸的是,传统的基于 MS 的肿瘤配体组不能区分源自内吞肿瘤材料的肽和源自自噬的肽。 因此,我们设计了一个实验,在该实验中我们分析了“fed”重同位素标记的肿瘤细胞的 DCs 的 HLA-DR 配体组(图 7A)。
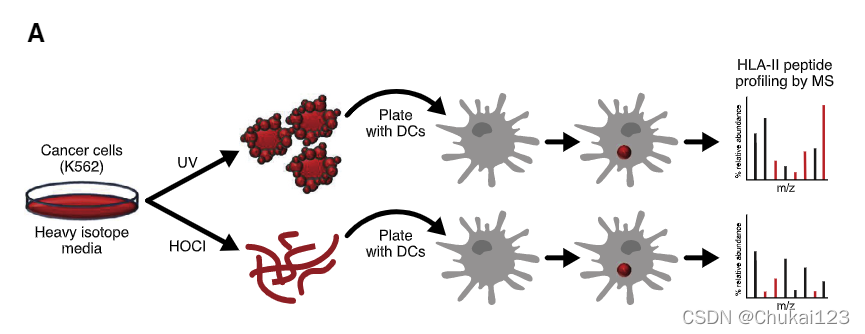
?为了标记肿瘤衍生蛋白,我们在含有同位素标记的 L 和 K 的培养基中培养了 HLA-II 缺陷型癌细胞系 (K562),标记效率 > 95%。 DCs 被喂食裂解的肿瘤细胞(以模拟肿瘤碎片的巨胞饮作用)或经紫外线处理的整个肿瘤细胞(以模拟整个细胞的吞噬作用)。 用 MS 分析 HLA-DR 结合肽并区分为重标记或轻标记。 实验分别产生了 29 和 56 个重标记肽用于裂解物和 UV 实验(data?S1B)。 除了两种情况外,含有超过一个 L 或 K 的肽在所有情况下都显示完全标记,这表明重标记的肽来源于肿瘤细胞而不是来自新翻译的 DC 蛋白,这将显示不一致的标记。 未经处理的 DCs 和用裂解物孵育 10 分钟后收获的 DCs 均未产生重标记肽。
使用我们的集成预测算法,我们评估了我们预测肿瘤衍生肽的能力。 和以前一样,基于 neonmhc2 的模型比基于 NetMHCIIpan 的模型实现了更高的预测精度(图 7B)。
与基因表达不同,基因偏差和 DQ 重叠特征并没有改善对内吞抗原的预测,这表明我们从大块组织配体组中学到的模式与这类表位无关。 分析重标记肽的源基因,我们观察到 RNA 结合 (RBP)、DNA 结合 (DBP)、热休克 (HSP) 和线粒体蛋白 (图 7C) 而不是富含大量配体组的分泌蛋白和膜蛋白 (图 5A)。 目前尚不清楚这是否代表了不同的处理偏好。 事实上,源蛋白通常在 K562 中高度表达(中位数为 430 TPM,而光标记肽源蛋白为 130 TPM),这表明检测限可能会驱动观察到的基因偏好。
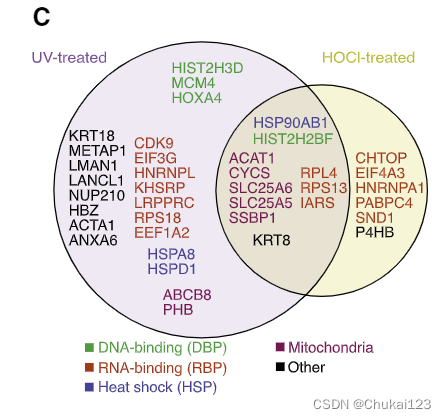
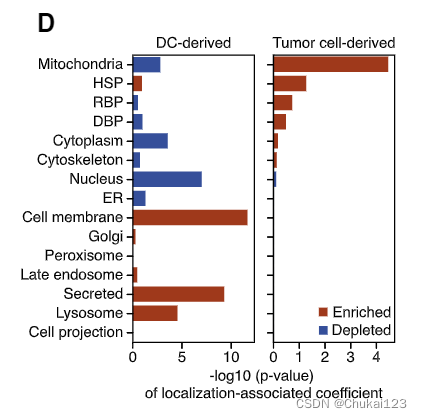
?为了清楚起见,我们建立了逻辑回归模型来测试基因定位和功能类别是否可以改进肽预测,而不是已经考虑了结合和基因表达的模型。 当我们考虑结合和表达时,RBP、DBP 和 HSP 不再显着,但线粒体蛋白仍然显着(p = 2.6e-4;图 7D)。 值得注意的是,富集模式与我们观察到的光标记肽完全不同。
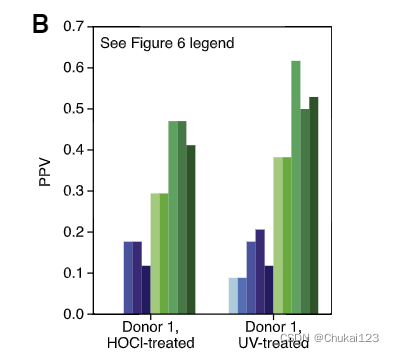
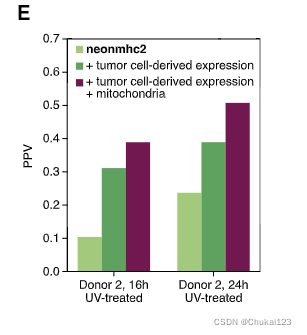
?为了确定线粒体偏差是否可以改善预测,我们收集了来自不同供体的数据,并通过增加细胞输入来实现更深的覆盖,仅关注紫外线治疗方案,并增加 24 小时孵育时间点。 该实验分别在 16 h 和 24 h 时间点产生了 77 和 59 个重标记肽,并共同鉴定了 78 个独特的源基因。 使用说明线粒体偏好的逻辑回归模型(根据第一个供体的数据进行训练),我们能够将 PPV 比仅包括结合和表达的模型净增加 8%–12%(图 7E)。 这些改进是显着的(16 小时 p = 1.1e-9 和 24 小时 p = 1.5e-8)。 因此,呈递来自吞噬细胞的抗原的 DCs 不会从所有细胞定位中同等地取样。 这些偏好无法从大块组织配体组中学习,可用于实现更准确的表位预测。
4、Conclusion
研究背景:TME(肿瘤微环境) 中的 HLA-II 呈现与接受癌症免疫治疗的患者的阳性结果相关。不幸的是,HLA-II 配体预测的不准确性以及肿瘤抗原如何在 TME 中呈现的模糊性减缓了靶向 HLA-II 表位的疗法的发展。
为此,本文全面分析了肿瘤配体组以定义 HLA-II 配体处理规则,并开发了一种称为 MAPTAC 的单等位基因分析技术,该技术能够快速分析超过 40 个 HLA-II 等位基因,包括 35 个 HLA-DR 等位基因,覆盖率达 95% 的美国患者。
针对HLA-II型的,并且针对美国患者!,能快速分析超过40个等位基因,包括35个HLA-DR等位基因;
此外,在 MAPTAC 数据上训练的结合预测算法 neonmhc2 在预测 HLA-II 配体组实验中呈现的肽方面的表现优于 NetMHCIIpan。
尽管 neonmhc2 并非旨在学习 T 细胞识别规则,但预计改进的 HLA-II 呈递预测因子将增强治疗相关 CD4+ 表位的识别。
同时观察到 neonmhc2 在识别 TGEM 验证数据集中的记忆 CD4+ T 细胞反应方面的性能优于 NetMHCIIpan。 此外,本文的算法还擅长预测体外诱导的 CD4+ T 细胞对新抗原的反应,成功识别出 NetMHCIIpan 不会优先考虑的免疫原性新表位。
与传统的肽结合测定相比,使用 MAPTAC 直接分析内源性加工和呈递的 HLA-II 配体的一个优点是存在肽加载伴侣,例如 HLA-DM。已知 HLA-DM 在编辑 APC 的 HLA-II 肽库中发挥作用(Rock 等人,2016;Yin 等人,2015),这促使我们研究其差异表达对 HLA-II 的影响配体。当 HLA-DM 在 HLA-DR 和 HLA-DQ MAPTAC 实验中过表达时,与没有 HLA-DM 过表达的实验相比,结合基序更清晰。相反,HLA-DM 似乎对 HLA-DP 等位基因的结合基序没有影响。这些数据表明,为了构建准确的 HLA-II 表位预测因子,可能需要学习等位基因特异性的肽负载要求。除了 HLA-DM,MAPTAC 平台提供了一种快速了解参与 HLA-II 通路的其他伴侣和蛋白质(如 CD74 或 HLA-DO)如何影响不同 HLA-II 等位基因的肽结合库的方法。
HLA-DM:
HLA-DM 存在于内吞途径的细胞内 MHC II 类区室 (MIIC) 中,它在 II 类分子与肽的负载中起重要作用。 新生 II 类分子的肽结合位点被称为 CLIP 的不变链的一部分占据,其功能是在到达 MIIC 囊泡之前抑制肽负载。 HLA-DM 与 II 类分子的结合催化 CLIP 的释放并促进用适当的肽替代。
关于肿瘤生物学,本文的观察结果是 APC 是多种肿瘤类型中大部分瘤内 HLA-II 表达的原因。这表明治疗相关肿瘤抗原的呈递可能取决于凋亡肿瘤细胞的吞噬作用或分泌的肿瘤蛋白的巨胞饮作用。
尽管有直接杀死 CD4+ T 细胞的报道(Tran 等人,2014),但本文的数据表明,CD4+ T 细胞通常在 TME 中发挥支持作用,主要识别浸润性白细胞上呈递的肿瘤抗原(Friedman 等人, 2012;Kenter 等人,2009;Ossendorp 等人,1998;Quezada 等人,2010)。因此,CD4+ T 细胞的抗肿瘤作用可能是通过分泌趋化因子和细胞因子来调节其他免疫细胞的运输和激活,包括具有直接溶细胞功能的细胞(Kreiter 等,2015;Tran 等。 , 2014)。尽管这在机制上更复杂,但一个好处是肿瘤对 HLA-II 抗原是否呈递的控制较少,这表明免疫逃逸是通过功能丧失突变,这是肿瘤避免 HLA-I 呈递的一种常见机制(McGranahan 等人)。 ., 2017; Sade-Feldman et al., 2017),可能不会像 HLA-II 那样频繁。
未来的研究将仔细定义哪些 APC 群体负责呈递内吞的肿瘤抗原,以及是否有办法增强这些吞噬细胞向 TME 的募集,这将对该领域有益(Saxena 和 Bhardwaj,2018 年)。此外,了解肿瘤细胞死亡的不同模式(如缺氧、化疗和放疗)如何影响 APC 捕获肿瘤抗原的效率将是有用的,这可能导致与 HLA-II 靶向治疗的最佳治疗组合(Emens 和 Middleton , 2015)。
细胞死亡的不同模式:
最后,对 HLA-II 配体组的全面分析导致观察到某些基因似乎比它们的表达预测的更频繁地出现。从肿瘤细胞中学习基因特异性偏差有助于改进 APC HLA-II 配体组的预测;然而,这些信号似乎与新抗原预测不太相关。例如,本文检测到似乎与 APC 中的自噬和膜再循环有关的富集,而不是与外源抗原的摄取有关。相反,当我们在体外将肿瘤细胞“fed”到 DC 时,源基因鉴定反而显示线粒体基因的富集。这种偏好的机制尚不清楚,但可能与线粒体的双膜结构有关,并且可能通过追踪吞噬细胞内单个细胞器降解的成像研究来阐明(Yadav 等,2014)。在任何情况下,要注意的是,基于同位素标记的 HLA 配体组学工作流程的效用不仅限于肿瘤抗原,因为它还可以应用于研究涉及传染病和自身免疫的抗原。
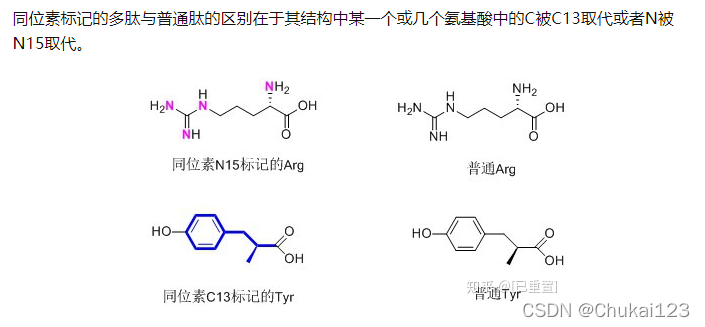
随着多肽在生物医药领域越来越广泛和深入的应用,标记和修饰性的多肽种类的需求越来越多,质量需求也越来越高。同位素标记就是其中典型的一种。同位素标记示踪,可以实现肽类代谢途径研究,能够随时追踪含有同位素标记的多肽在体内或体外位置及数量的变化情况。同位素标记具有高灵敏度、定位简单、定量准确等优点,使得同位素修饰在医学及生物化学领域得到越来越广泛的关注。
?参考知乎:同位素标记法
总之,HLA-II 处理和呈现的规则比 HLA-I 更复杂。 基于这个原因,驱动 CD4+ T 细胞反应的抗原通常仍未确定。 本文在定义 HLA-II 结合和处理规则方面取得的进展将能够识别可靶向的癌症抗原和其他与疾病相关的表位,这些表位可以转化为更有效的治疗方法。