本篇博客参考:iris-经典案例解析-机器学习

我们要解决的问题如下:
已知鸢尾花iris分为三个不同的类型:山鸢尾花Setosa、变色鸢尾花Versicolor、韦尔吉尼娅鸢尾花Virginica,这个分类主要是依据鸢尾花的花萼长度、宽度和花瓣的长度、宽度四个指标(也可能还有其他参考)。我们并不知道具体的分类标准,但是植物学家已经为150朵不同的鸢尾花进行了分类鉴定,我们也可以对每一朵鸢尾花进行准确测量得到花萼花瓣的数据。
那么问题来了,你女朋友家的一株鸢尾花开花了,她测量了一下,花萼长宽花瓣长宽分别是3.1、2.3、1.2、0.5,然后她就问你:“我家这朵鸢尾花到底属于哪个分类?”
一、检查数据
数据格式有无问题?
数据数值有无问题?
数据是否需要修复和删除?
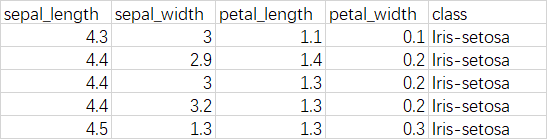
表格说明:横行属于一朵花的数据
Sepal length/width:花萼的长度/宽度数据
Petal length/width:花瓣的长度/宽度数据
class:植物学家鉴定的花的类型
import seaborn as sns
import numpy as np
import pandas as pd
import matplotlib as mpl
import matplotlib.pyplot as plt
%matplotlib inline
plt.rcParams['font.sans-serif']=['Microsoft YaHei'] # 用来正常显示中文标签
plt.rcParams['axes.unicode_minus']=False # 用来正常显示负号
from datetime import datetime
plt.figure(figsize=(16,10))
import pyecharts.options as opts
from pyecharts.charts import Line
from pyecharts.faker import Faker
from pyecharts.charts import Bar
import os
from pyecharts.options.global_options import ThemeType
iris_data=pd.read_csv("iris.csv",na_values='NA') # na_values:在读取的时候直接将空值赋值为NA
iris_data.head()
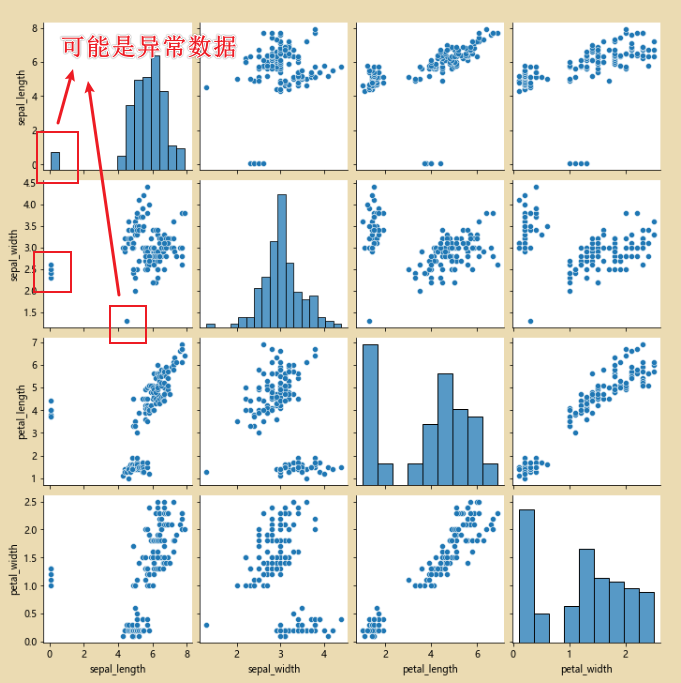
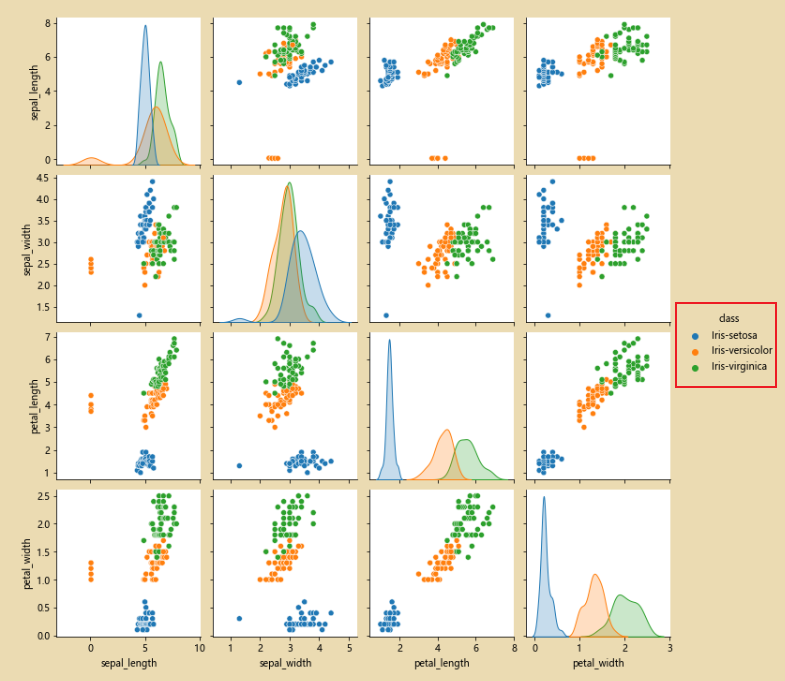
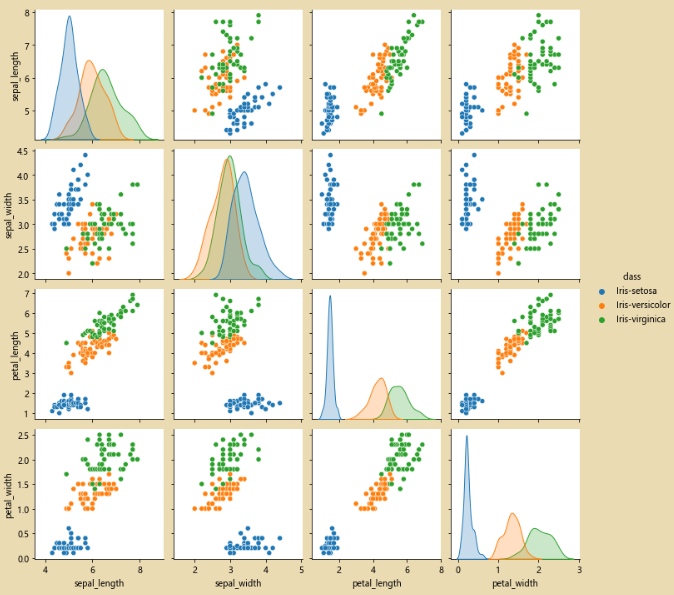
# 使用pairpoint进行检查,看看原始的数据是否有问题
sns.pairplot(iris_data.dropna())
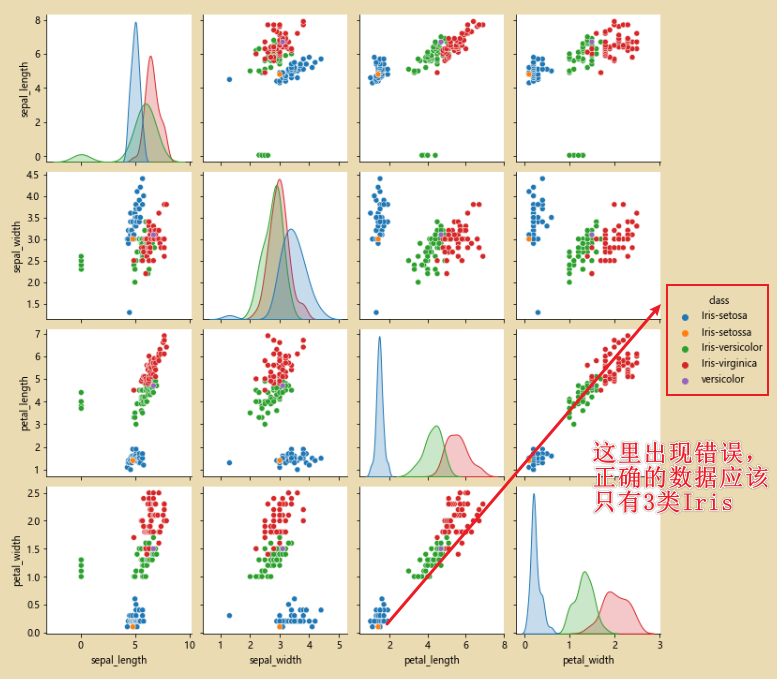
sns.pairplot(iris_data.dropna(),hue='class')# hue='class' 按照class进行分类
二、清理数据
# 3 对数据进行修正
cond=(iris_data['class']=='Iris-setosa') & (iris_data['sepal_width']<2.5)
iris_data.loc[cond]
iris_data.loc[iris_data['class']=='versicolor','class']='Iris-versicolor'
iris_data.loc[iris_data['class']=='Iris-setossa','class']='Iris-setosa'
sns.pairplot(iris_data.dropna(),hue='class')
此时经过修正完之后,就只有三类数据了
显示图片:
from PIL import Image
img = Image.open('test.jpg')
plt.imshow(img)
plt.show()

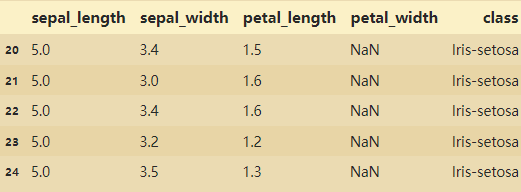
1、对缺失值NAN进行修正
# 对缺失值NAN进行修正(找出所有字段的缺失值)
iris_data.loc[
(iris_data['sepal_width'].isnull()) |
(iris_data['sepal_length'].isnull()) |
(iris_data['petal_width'].isnull()) |
(iris_data['petal_length'].isnull())
]
找到的异常值:
# 使用mean均值对nan进行替换
irissetosa=iris_data['class']=='Iris-setosa'
irissetosa
avgpetalwd=iris_data.loc[irissetosa,'petal_width'].mean()
avgpetalwd
iris_data.loc[irissetosa & (iris_data['petal_width'].isnull()),'petal_width']=avgpetalwd

2、对sepal_width异常数据进行纠正
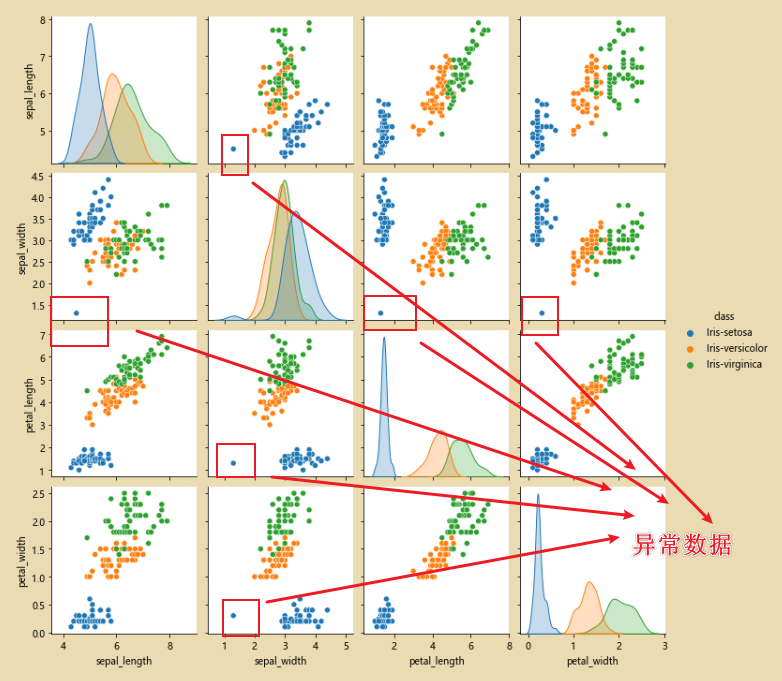
分析:有图中该异常点的颜色可知,该数据是属于Iris-setosa,找到异常数据之后,填充为均值即可。
通过切片方式找到这个异常点:
cond=(iris_data['class']=='Iris-setosa') & (iris_data['sepal_width']<2.0)
iris_data.loc[cond]
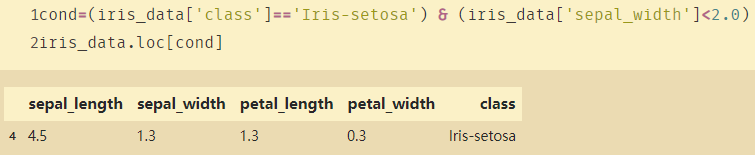
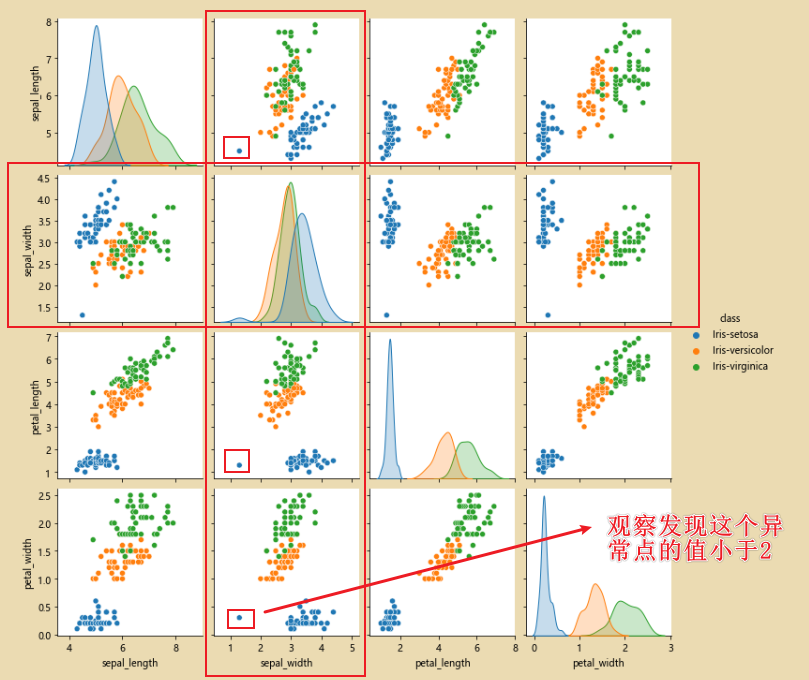
接下来寻找sepal_width的均值:
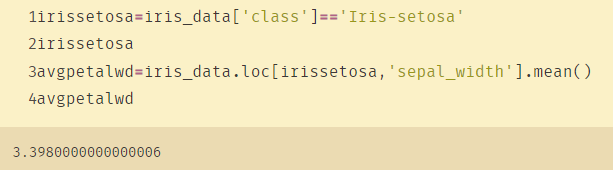
对异常值赋值为均值:
iris_data.loc[irissetosa & (cond),'sepal_width']=avgpetalwd
此时就没有异常数据了:
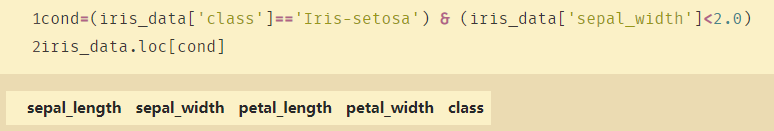
在来看看图片,如下图,异常点就消失了:
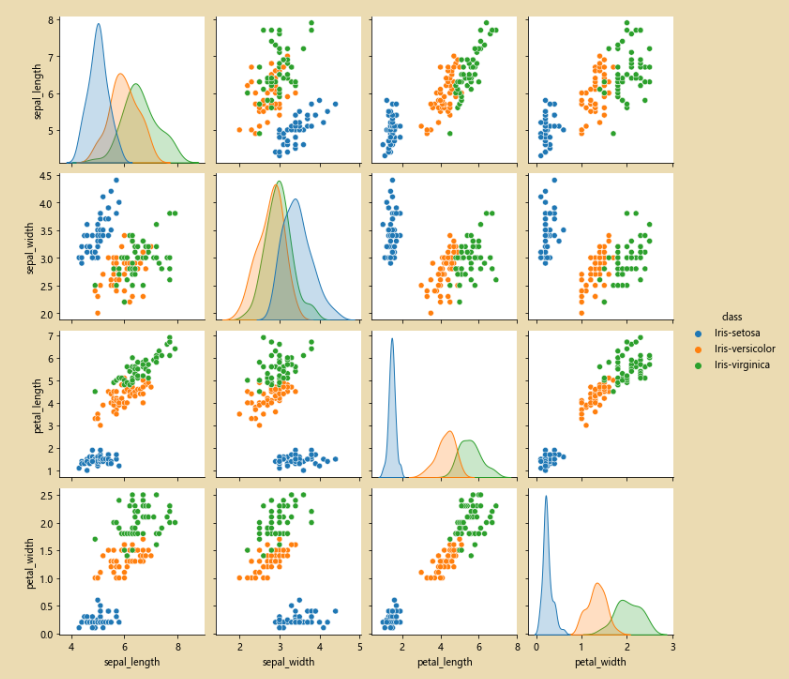
三、决策树和随机森林
1、绘制图形
%matplotlib inline
import matplotlib.pyplot as plt
import seaborn as sb
sb.pairplot(iris_data.dropna(),hue='class')
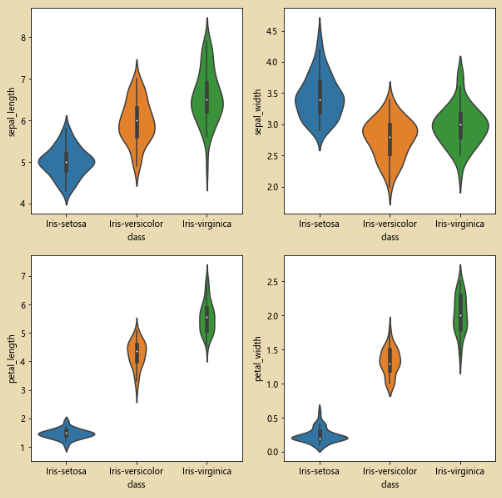
绘制小提琴图:
plt.figure(figsize=(10,10))
for column_index,column in enumerate(iris_data.columns):
if column == 'class':
continue
plt.subplot(2,2,column_index + 1)
sb.violinplot(x='class' ,y=column,data=iris_data)
2、比较参数
from sklearn.model_selection import train_test_split
all_inputs=iris_data[['sepal_length','sepal_width','petal_length','petal_width']].values
all_inputs
all_classes=iris_data['class'].values
(training_inputs,
testing_inputs,
training_classes,
testing_classes)=train_test_split(all_inputs,all_classes,train_size=0.75,random_state=1)
all_classes
training_inputs
# train_size=0.75 :75%作为训练集
from sklearn.tree import DecisionTreeClassifier
decision_tree_classifier=DecisionTreeClassifier()
decision_tree_classifier.fit(training_inputs,training_classes)
-
criterion :gini or entropy
评判标准:基尼系数或者熵
例:criterion='gini'(表示使用基尼系数实现决策树) -
splitter:best or random
在所有特征中找最好的切分点 (best)或者随机选取( random) -
max_features:表示是否需要把所有特征加到决策树当中。
有参数:None(all), log2, sqrt, N。 特征小于50时一般取所有,大于50则可以考虑使用log2作为参数。 -
min_depth
数据少或者特征少时可以不管这个值,如果模型样本多,可以适当限制这个值 -
max_depth:树的最大深度
-
min_samples_split:
如果某节点的样本数少于min_samples_split,则不会继续再尝试选择最优特征来进行划分。如果样本不大,不需要管这个值。如果样本数量级非常大,建议增大这个值。 -
min_samples_leaf:叶子节点的最小样本数。
这个值限制了特兹节点最少的样本数,如果某叶子节点数目小于样本数,则会和兄弟节点一起剪枝。如果样本量不大,不需要管这个值,大些如10万可以尝试下5。 -
min_weight_fraction_leaf
这个值限制了叶子节点所有样本权重和最小值,如果小于这个值,则会和兄弟节点一起被剪枝默认为0,就是不考虑权重问题
一般来说,如果我们有较多样本有缺失值,或者分类树样本的分布类别偏差很大,就会引入样本权重,这是要注意这个值 -
max_leaf_nodes:
通过限制最大叶子节点数,可以防止过拟合,默认是’None’,即不限制最大的叶子节点数。如果加了限制,算法会简历在最大叶子节点数内最优的决策树。如果特征不多,可以不考虑这个值,但是如果特征成分多的话,可以加限制,具体的值可以通过交叉验证得到。 -
class_weight
指定样本各类别的权重,主要是为了防止训练集某些类别的样本过多,导致尊练的决策树过于偏向这些类别
这里可以自己指定各个样本的权重,如果使用’balanced’,则算法会自己计算权重,样本最少的类别所对应的样本权重会高 -
min_inpurity_split
这个值限制了决策树的增长,如果某杰底单的不纯度(基尼系数,信息增益,均方差,绝对值)小于这个阈值
则节点不再生成子节点,即为叶子节点 -
train_size=0.75 :75%作为训练集
-
min_impurity_decrease:节点的纯度。节点可能出现分割的不够干净的问题。导致后期在叶子节点生成部分无法生成叶子节点或者太细的问题。
-
random_state:其数值发生变化,则得到的数据也会发生变化。
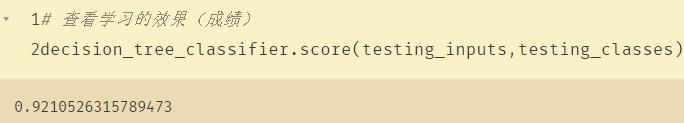
查看学习的效果(成绩):
decision_tree_classifier.score(testing_inputs,testing_classes)
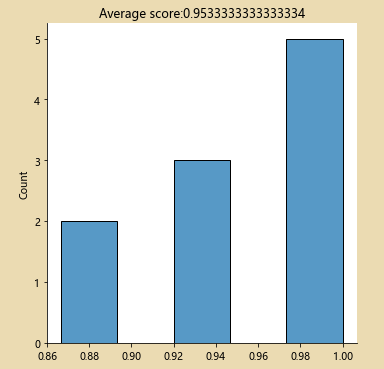
查看交叉比较之后的成绩 :
from sklearn.model_selection import cross_val_score
import numpy as np
decision_tree_classifier=DecisionTreeClassifier()
cv_scores=cross_val_score(decision_tree_classifier,all_inputs,all_classes,cv=10)
print(cv_scores)
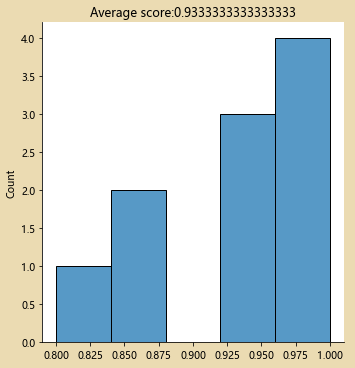
sb.displot(cv_scores)
plt.title('Average score:{}'.format(np.mean(cv_scores)))
decision_tree_classifier=DecisionTreeClassifier(max_depth=3) # max_depth:表示分类是否有区别,分几类
cv_scores=cross_val_score(decision_tree_classifier,all_inputs,all_classes,cv=10)
print(cv_scores)
sb.displot(cv_scores,kde=False)
plt.title('Average score:{}'.format(np.mean(cv_scores)))
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import StratifiedKFold
decision_tree_classifier=DecisionTreeClassifier()
parameter_grid={'max_depth':[1,2,3,4,5],
'max_features':[1,2,3,4]}
# 将每个max_depth与每个max_features都组合一遍,找出其中最好的
skf=StratifiedKFold(n_splits=10)
cross_validation=skf.get_n_splits(all_inputs,all_classes)
grid_search=GridSearchCV(decision_tree_classifier,
param_grid=parameter_grid,
cv=cross_validation)
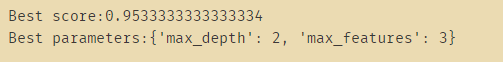
grid_search.fit(all_inputs,all_classes)
print('Best score:{}'.format(grid_search.best_score_)) # 得分最高
print('Best parameters:{}'.format(grid_search.best_params_)) # 得分最好
绘制热力图
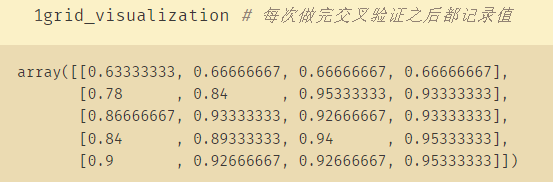
grid_visualization=[]
for grid_pair in grid_search.cv_results_['mean_test_score']:
grid_visualization.append(grid_pair)
grid_visualization=np.array(grid_visualization)
grid_visualization.shape=(5,4)
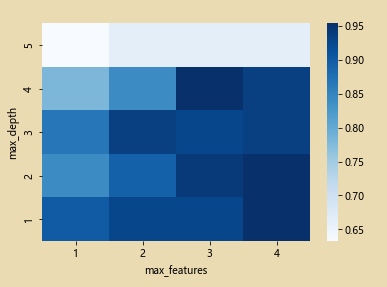
sb.heatmap(grid_visualization,cmap='Blues') # 蓝色
plt.xticks(np.arange(4)+0.5,grid_search.param_grid['max_features'])
plt.yticks(np.arange(5)+0.5,grid_search.param_grid['max_depth'][::-1])
plt.xlabel('max_features')
plt.ylabel('max_depth')
3、调用Graphviz绘制决策树
decision_tree_classifier=grid_search.best_estimator_
decision_tree_classifier # 拿到得分最高的数据
import sklearn.tree as tree
from six import StringIO
with open('iris_dtc.dot','w') as out_file:
out_file=tree.export_graphviz(decision_tree_classifier,out_file=out_file)
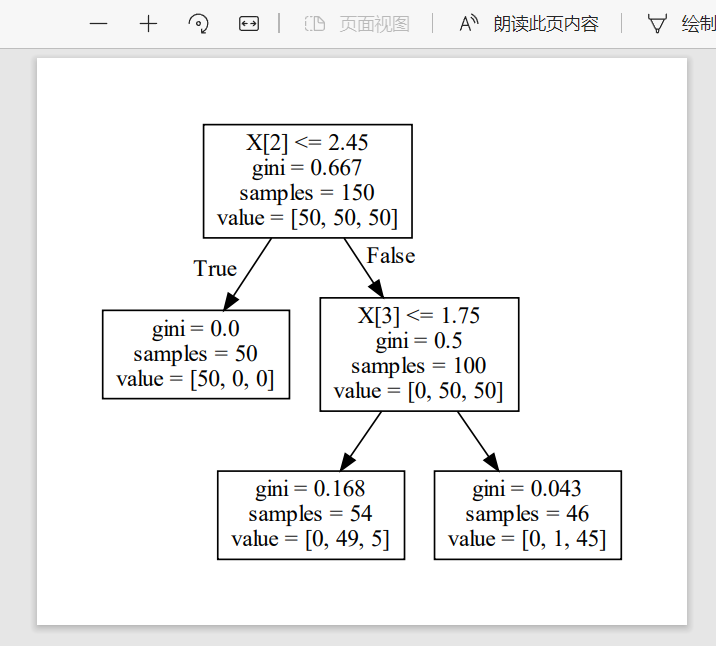
此时就生成一个文档:(第几个节点选择哪几个,把结果写在dot文档)

文档内容:
digraph Tree {
node [shape=box] ;
0 [label="X[2] <= 2.45\ngini = 0.667\nsamples = 150\nvalue = [50, 50, 50]"] ;
1 [label="gini = 0.0\nsamples = 50\nvalue = [50, 0, 0]"] ;
0 -> 1 [labeldistance=2.5, labelangle=45, headlabel="True"] ;
2 [label="X[3] <= 1.75\ngini = 0.5\nsamples = 100\nvalue = [0, 50, 50]"] ;
0 -> 2 [labeldistance=2.5, labelangle=-45, headlabel="False"] ;
3 [label="gini = 0.168\nsamples = 54\nvalue = [0, 49, 5]"] ;
2 -> 3 ;
4 [label="gini = 0.043\nsamples = 46\nvalue = [0, 1, 45]"] ;
2 -> 4 ;
}
使用Graphviz绘制决策树图片,注意,这里要先安装Graphviz,配置环境变量,并且打开dot.exe。
在命令行中先转入iris_dtc.dot文件所在的目录,然后调用如下命令

dot -Tpdf iris_dtc.dot -o irisdt.pdf
此时就生成我们需要的pdf文件: