数据集的详细说明如下:

从最后一列的输出值可以看出为0或1,属于二分类,因此可以使用逻辑回归实现。
用pandas读取csv文件,前面几个都用的是np.genfromtxt文件读取。个人觉得没什么大的区别,pandas用的更多吧,常用于数据分析。
数据分析基本过程:①提出问题,②理解数据需求 ,③数据清洗, ④构建模型 。⑤数据可视化;
数据清洗的基本过程:①选择子集,②列名重命名,③缺失数据处理,④数据类型转换,⑤数据排序,⑥异常值处理。
1. 读取数据
# 读取前5行
diabetes_data = pd.read_csv('diabetes.csv')
print(diabetes_data.head())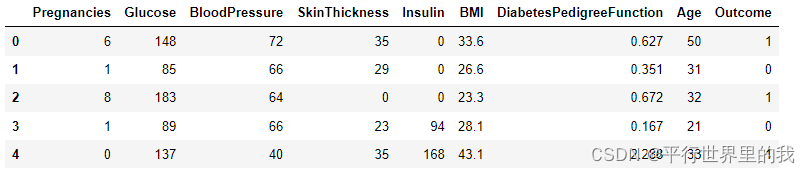
?2.查看数据形状
print(diabetes_data.shape)![]()
共有768行9列
3.查看数据信息
print(diabetes_data.info(verbose=True))
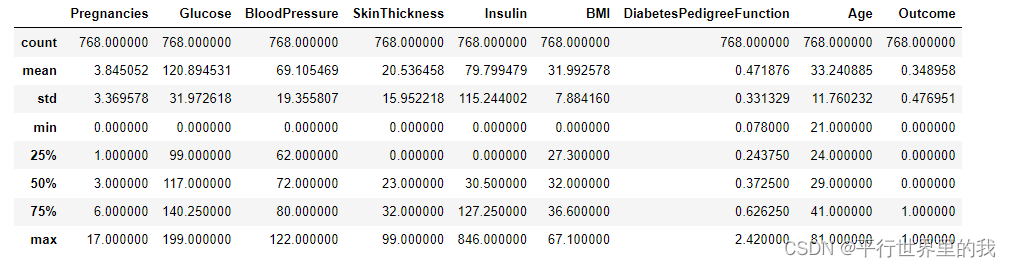
可以看出每一个数据都是768行,没有缺失值。后面是数据类型。
4.查看数据描述
print(diabetes_data.describe())5.重命名(rename)以及缺失值处理
本实验中用不到,所以不做此变换。感兴趣的可查看
numpy genfromtxt 读取字符_Numpy和Pandas常用数据处理方法
6.查看标签分布
print(diabetes_data.Outcome.value_counts())
# 使用柱状图的方式画出标签个数统计
p=diabetes_data.Outcome.value_counts().plot(kind="bar")
plt.show()
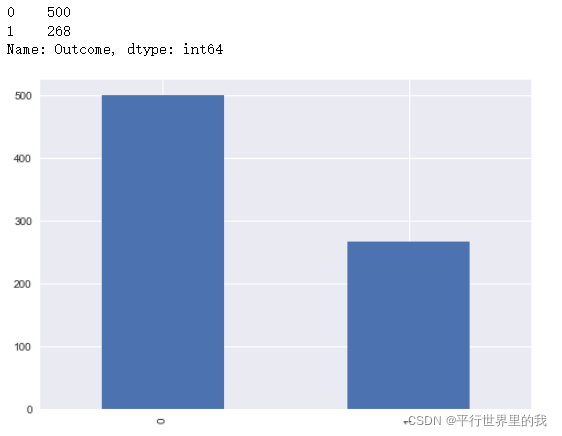
?属于0这一类的有数据500个,属于1这一类的有268个数据
7.可视化数据分布
p=sns.pairplot(diabetes_data, hue = 'Outcome')
plt.show()?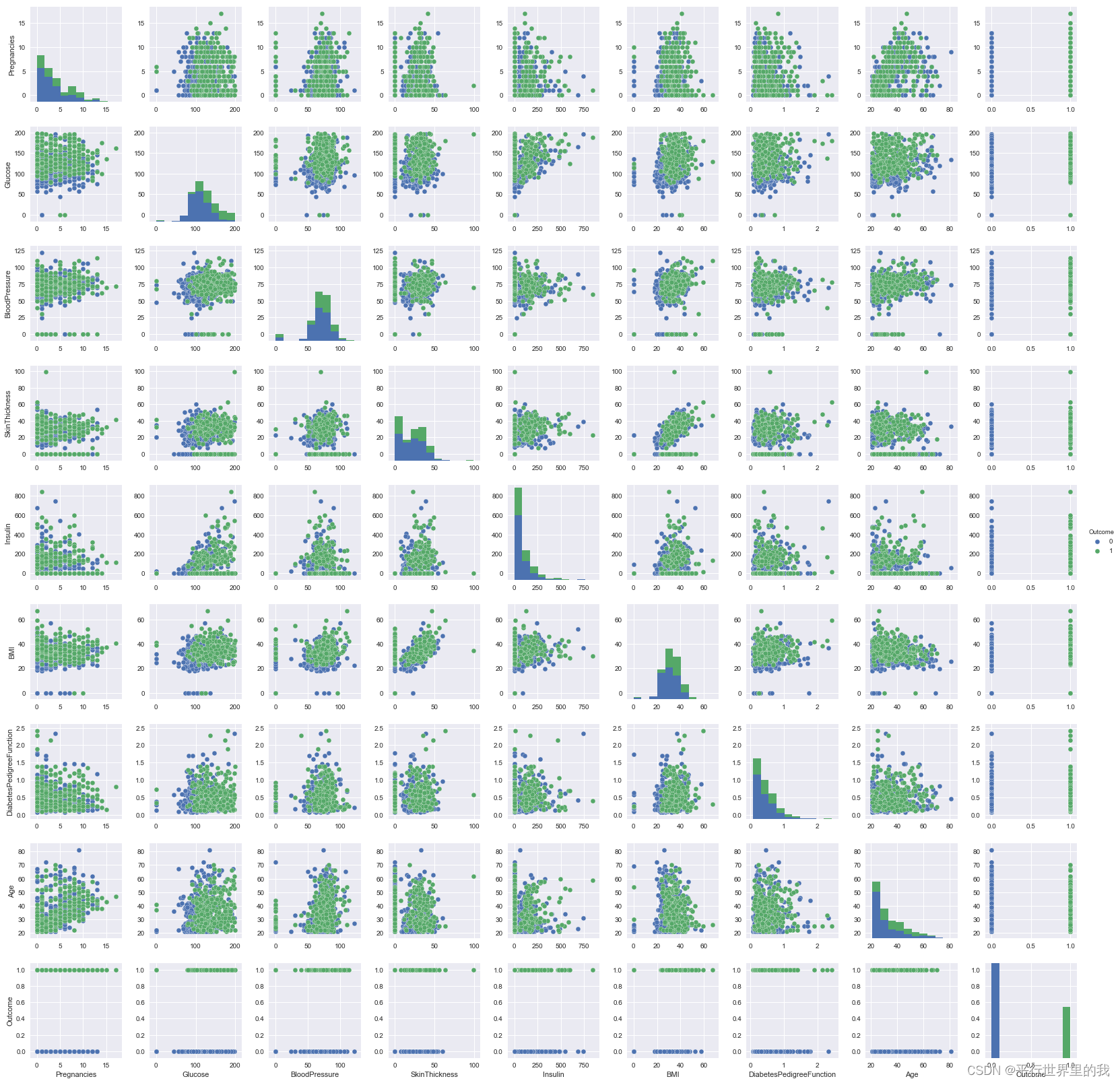
结论:这里画的图主要是两种类型,直方图和散点图。
单一特征对比的时候用的是直方图,不同特征对比的时候用的是散点图,显示两个特征的之间的关系。
观察数据分布我们可以发现一些异常值,比如Glucose葡萄糖,BloodPressure血压,SkinThickness皮肤厚度,Insulin胰岛素,BMI身体质量指数这些特征应该是不可能出现0值的。
8.异常值处理
colume = ['Glucose', 'BloodPressure', 'SkinThickness', 'Insulin', 'BMI']
diabetes_data[colume] = diabetes_data[colume].replace(0,np.nan)
用空值替换。
missingno可视化空值
# pip install missingno
import missingno as msno
p=msno.bar(diabetes_data)
plt.show()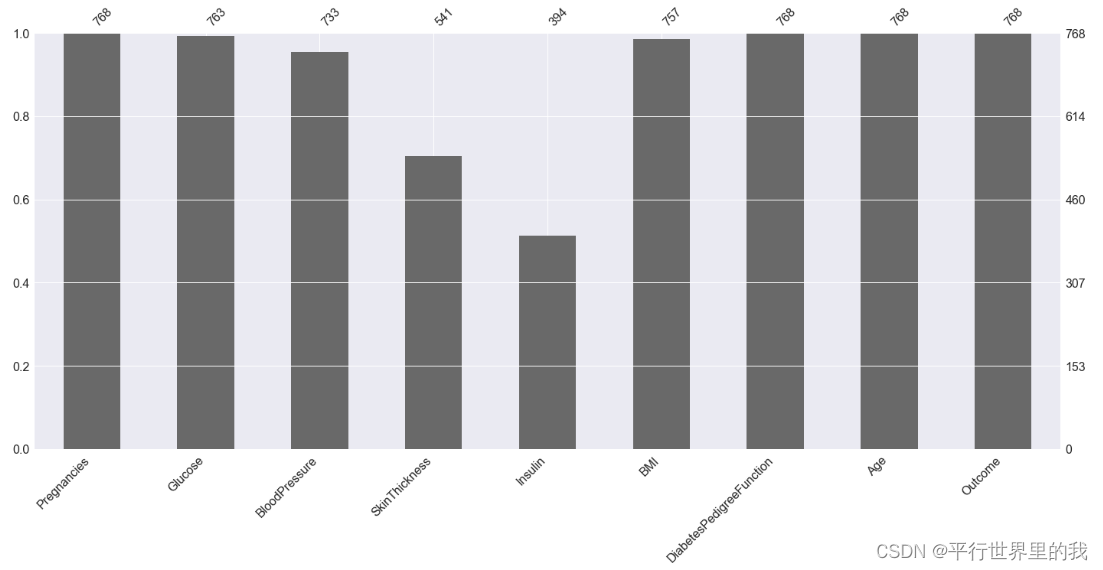
?可以看出有两列很多空值,一般对于这种空值很多的数据直接删除。
对于有一些缺失值的数据,可以进行填充,中位数或平均值
# 设定阀值
thresh_count = diabetes_data.shape[0]*0.8
# 若某一列数据缺失的数量超过20%就会被删除
diabetes_data = diabetes_data.dropna(thresh=thresh_count, axis=1)# 导入插补库
from sklearn.preprocessing import Imputer
# 对数值型变量的缺失值,我们采用均值插补的方法来填充缺失值
imr = Imputer(missing_values='NaN', strategy='mean', axis=0)
colume = ['Glucose', 'BloodPressure', 'BMI']
# 进行插补
diabetes_data[colume] = imr.fit_transform(diabetes_data[colume])9.查看数据的相关性
plt.figure(figsize=(12,10))
# 画热力图,数值为两个变量之间的相关系数
p=sns.heatmap(diabetes_data.corr(), annot=True)
plt.show()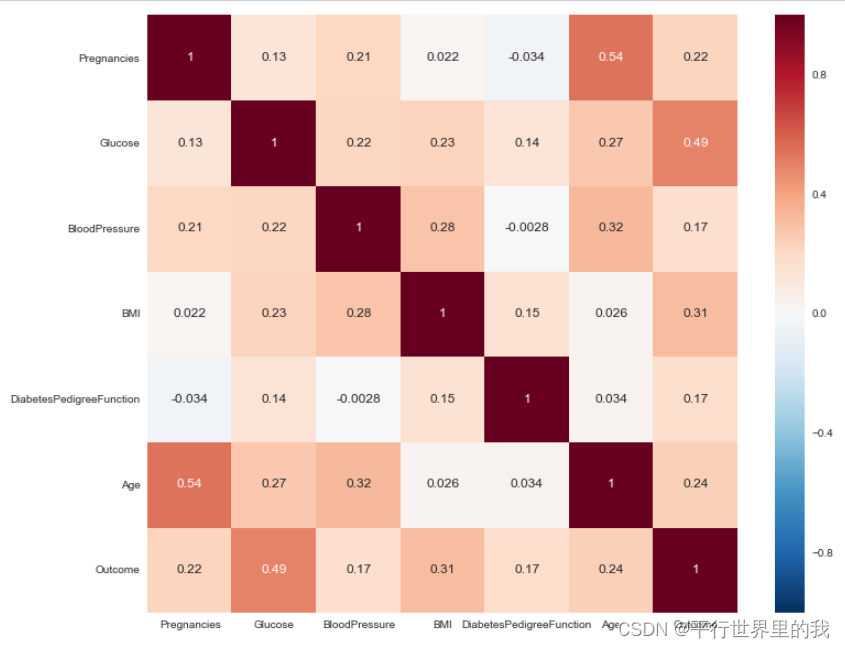
?对称。只看一半,一般只看较大的值,相关性较大
整体代码
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import missingno as msno
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import classification_report
# 载入数据
diabetes_data = pd.read_csv('diabetes.csv')
print(diabetes_data.head())
# 数据信息
print(diabetes_data.info(verbose=True))
# 数据描述
print(diabetes_data.describe())
# 数据形状
print(diabetes_data.shape)
# 查看标签分布
print(diabetes_data.Outcome.value_counts())
# 使用柱状图的方式画出标签个数统计
p=diabetes_data.Outcome.value_counts().plot(kind="bar")
plt.show()
# 可视化数据分布
p=sns.pairplot(diabetes_data, hue = 'Outcome')
plt.show()
'''
这里画的图主要是两种类型,直方图和散点图。
单一特征对比的时候用的是直方图,不同特征对比的时候用的是散点图,显示两个特征的之间的关系。
观察数据分布我们可以发现一些异常值,比如Glucose葡萄糖,BloodPressure血压,SkinThickness皮肤厚度,Insulin胰岛素,BMI身体质量指数这些特征应该是不可能出现0值的。
'''
# 把葡萄糖,血压,皮肤厚度,胰岛素,身体质量指数中的0替换为nan
colume = ['Glucose', 'BloodPressure', 'SkinThickness', 'Insulin', 'BMI']
diabetes_data[colume] = diabetes_data[colume].replace(0,np.nan)
p=msno.bar(diabetes_data)
plt.show()
# 设定阀值
thresh_count = diabetes_data.shape[0]*0.8
# 若某一列数据缺失的数量超过20%就会被删除
diabetes_data = diabetes_data.dropna(thresh=thresh_count, axis=1)
p=msno.bar(diabetes_data)
plt.show()
# 导入插补库
from sklearn.preprocessing import Imputer
# 对数值型变量的缺失值,我们采用均值插补的方法来填充缺失值
imr = Imputer(missing_values='NaN', strategy='mean', axis=0)
colume = ['Glucose', 'BloodPressure', 'BMI']
# 进行插补
diabetes_data[colume] = imr.fit_transform(diabetes_data[colume])
p=msno.bar(diabetes_data)
plt.show()
plt.figure(figsize=(12,10))
# 画热力图,数值为两个变量之间的相关系数
p=sns.heatmap(diabetes_data.corr(), annot=True)
plt.show()
# 把数据切分为特征x和标签y
x = diabetes_data.drop("Outcome",axis = 1)
y = diabetes_data.Outcome
# 切分数据集,stratify=y表示切分后训练集和测试集中的数据类型的比例跟切分前y中的比例一致
# 比如切分前y中0和1的比例为1:2,切分后y_train和y_test中0和1的比例也都是1:2
x_train,x_test,y_train,y_test = train_test_split(x,y,test_size=0.3, stratify=y)
LR = LogisticRegression()
LR.fit(x_train,y_train)
predictions = LR.predict(x_test)
print(classification_report(y_test, predictions))
整体结构大概在80%的正确率