??在做微生物生态学分析时,我们会时常使用一些探索性的数据分析方法,例如:
- 利用基于距离的PCoA、NMDS、t-SNE降维方法,探索群落结构的样点效应;
- 用RDA、CCA等约束性排序,探索群落结构的主要影响因素;
- 将环境因子与alpha多样性做相关分析;
- 利用随机森林确定主要影响因子;
- …
??如果这些方法都得不到想要的结果呢?譬如:我想探究某一生态过程(有机质的分解、反硝化作用等)与微生物群落结构的关系。这些生态过程可能和alpha多样性,群落结构的NMDS1等等都没什么关系,此时如何快速地确定与该生态过程显著相关的OTUs,基于模型的方法可以实现这一点。下面以gllvm包探索与某一生态过程显著相关的OTUs。
GLLVM_t <- gllvm(Y,X=X,formula=~t,family="negative.binomial", num.lv = 2,seed = 123)
coefplot.gllvm(GLLVM_t)
- Y: OTU table
- X: 生态过程度的度量,例如有机质分解速率、土壤呼吸等
运行上述代码后可得下图:
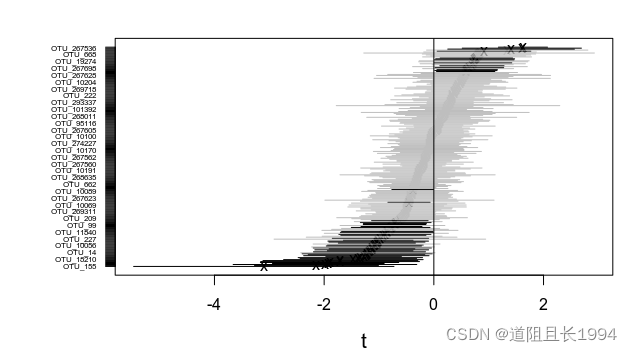
左边的黑线是有显著负贡献的OTUs,右边黑线是有显著正贡献的OTUs,这里的黑线是标准偏差的两倍。这样我们就找到了与该生态过程显著相关的标志性OTUs集合了,将显著正效应的OTUs累加起来与这一生态过程做散点图可得:
z <- GLLVM_t$params$Xcoef / GLLVM_t$sd$Xcoef
ny <- Y[,colnames(Y) %in% rownames(z)[z>2]]
nper <- rowSums(ny)/rowSums(Y)
plot(nper,X$t)
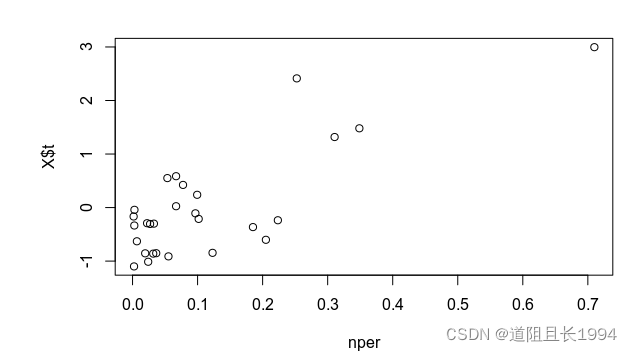
??不过这只是统计性质的数据探索,其中不一定有因果关系,怎么解释就要根据自己的实验设计和专业知识来解读啦。基于模型的群落生态学分析也是近十年才开始慢慢兴起,与诞生于上个世纪的数量生态学统计方法相比,计算速度偏慢(特别是用于OTU table这类动则上千上万列的高维数据),但基于模型的方法往往可以挖掘出更多的生态学信息,可参考如下文献进行深入学习:
- gllvm: Fast analysis of multivariate abundance data with generalized linear latent variable models in r
- Fast model-based ordination with copulas
- A new joint species distribution model for faster and more >-accurate inference of species associations from big community data
- How to make more out of community data? A conceptual framework and its implementation as models and software
