荷兰生态研究所Carrión等研究人员利用植物内生菌的宏基因组学(metagenomics)技术,表明某些“常驻”细菌可以保护植物根系免受真菌感染。揭示了生活在植物根内部的微生物协同工作下,可以促进植物的生长和对胁迫的耐受性。该研究成果以“Pathogen-induced activation of disease-suppressive functions in the endophytic root microbiome”为题发表在《科学》(Science)杂志上。

一些土壤表现出显著的植物病原菌的抑病功能,这种能力主要归因于与植物相关的微生物组。本研究通过研究枯萎真菌茄枯萎病菌感染甜菜的内生菌(在根中发现的亲密微生物群落)在真菌疾病抑制中的作用。转录组分析表明,几种细菌内生菌激活生物合成基因簇(BGCs),从而抑制疾病。这些生物产生抗真菌效应,包括可消化真菌细胞壁的酶和次生代谢产物(包括吩嗪、聚酮类化合物和铁载体),可能有助于抗真菌表型。
摘要:生活在植物内部的微生物可以促进植物的生长和健康,但它们的基因组和功能多样性在很大程度上尚不清楚。本研究中,植物内生菌的宏基因组学研究和网络推论表明,植物根部的真菌感染几丁质菌(Chitinophagaceae)和黄杆菌(Flavobacteriaceae)在根内富集;同时,几丁质酶基因、编码非核糖体肽合成酶(nonribosomal peptide synthetases,NRPSs)和聚酮合成酶(polyketide synthases,PKSs)产生的各种未知生物合成基因簇(BGCs)。在菌株基因组重建后,由几丁质菌和黄杆菌合成的菌群持续抑制真菌根部疾病。定点诱变表明,黄杆菌中此前未鉴定的NRPS-PKS基因簇对内生菌群抑病至关重要。研究结果表明,内生根微生物组(植物内生菌)具有许多尚不为人所知的功能性状,可以共同保护植物内外。
背景:植物菌群研究已经积累了大量的测序数据和丰富的信息,表明了许多植物根际、叶际、种子和胚层中不同微生物群落的多样性和丰富度。然而迄今为止,很少有研究论证微生物组对特定植物表型(即植物生长、发育和健康)的功能影响。因此,在分子和化学层面上,许多植物表型与微生物组结构和功能之间在的因果关系仍然未知。本研究旨在探讨植物内生微生物菌群(植物内生菌)的基因多样性及对真菌感染植株的保护作用。
方法:为了解生活在植物根组织中的植物内生菌的抑病功能,作者对生长在抑病土壤中的甜菜幼苗根内进行宏基因组测序分析;并鉴定与抑病相关的微生物群落和功能组成,以区分哪些生物合成基因簇(BGCs)在感染过程中上调,然后重组内生菌群;最后进行位点定向诱变分析,检测特异性BGCs是否在抑病过程中起着重要作用。
作者设置甜菜种植在感病(Conducive, C)和抑病 (Suppressive, S) 土壤和是否接种病原菌R.solani(R)共计四个处理(C、C+R、S、S+R)。在抑病土壤中接种病原菌(S+R)的甜菜发病率为15-30%,在感病土壤中接种病原菌(C+R)的甜菜发病率为80%。
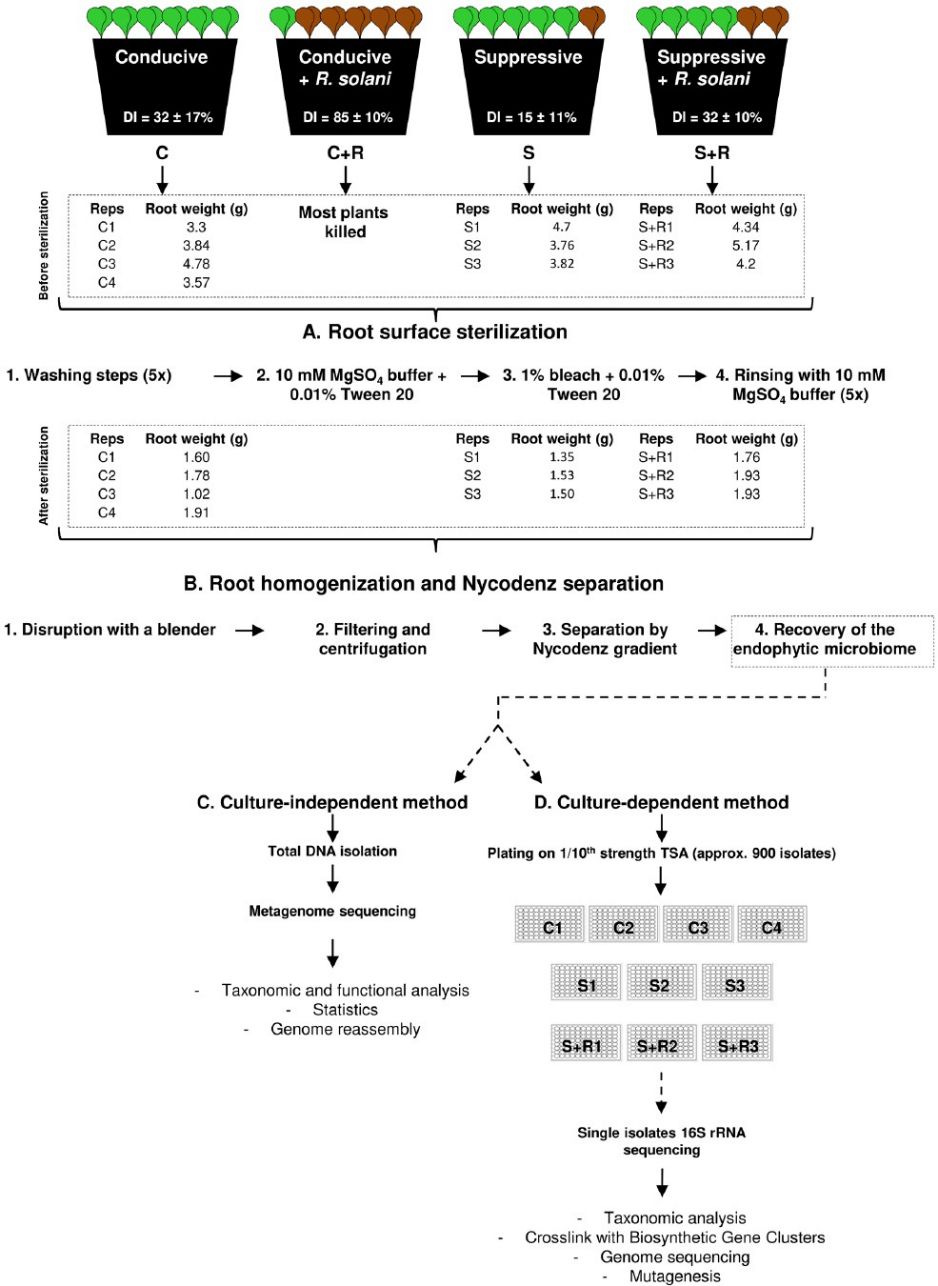
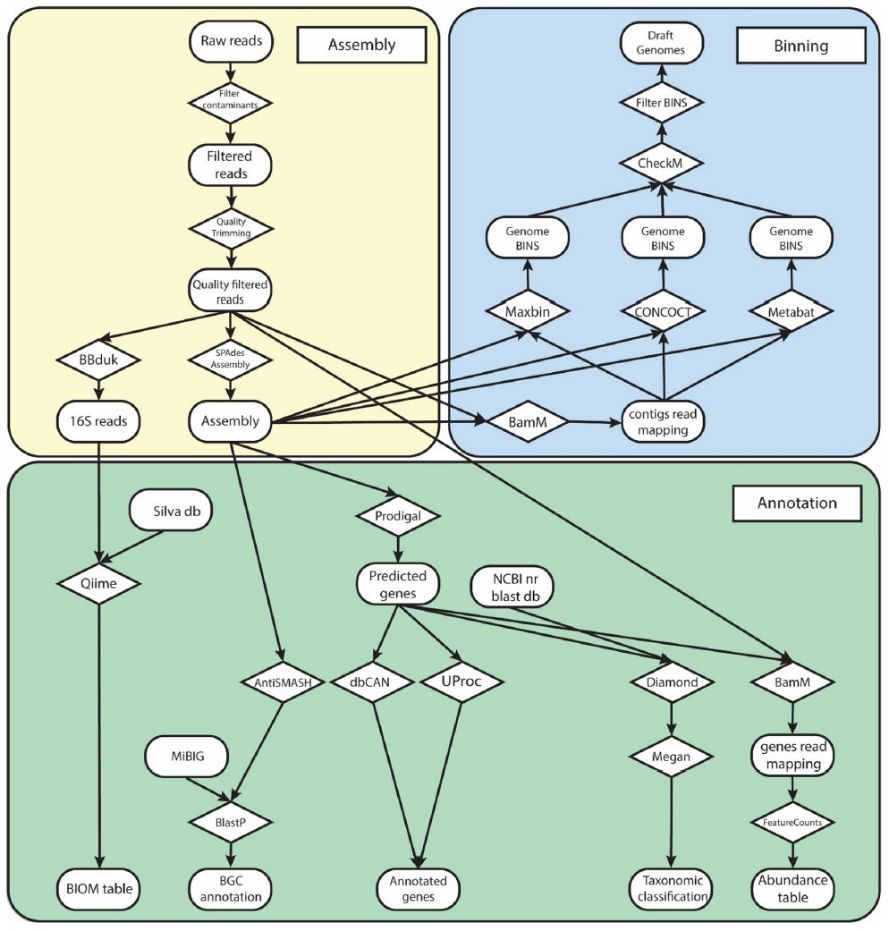
植物内生菌宏基因组测序分析流程

质控数据基因注释比例
主要结果
(1)内生菌群落多样性和网络分析

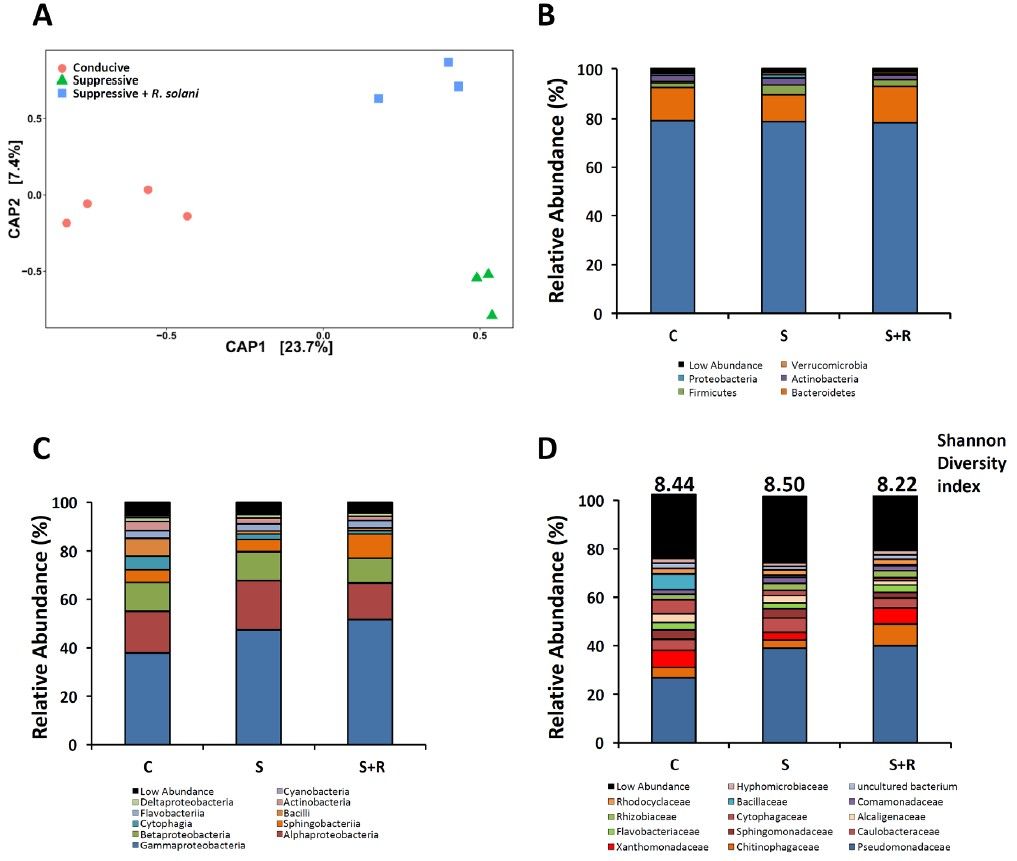
图1:病原菌改变植物内生菌群落结构和功能,S和S+R两个土壤中的不同植物内生菌群落丰度差异。
-
分类差异:通过宏基因组提取16S rRNA基因序列注释细菌群落并统计不同门类细菌差异。最大圈代表门水平,内圈从大到小分别代表纲、科和属。
-
功能差异:基于宏基因组序列注释功能和细菌相关功能差异。最小的圈代表COG功能组。圆圈大小代表不同细菌或功能的平均丰度。绿色表示S组,蓝色表示S+R组富集,黄色表示不显著。(FDR<0.1)
-
基因丰度:左散点图描绘全部S+R组(Sr)的全部基因与S组对应基因的丰度比;右散点图描绘S组的全部基因与C组对应基因的丰度比。从上到下按照S+R/S比值。每种COG类别缩写(C能量代谢;D细胞周期、细胞分裂和染色体分区;E氨基酸转运和代谢;F核苷酸转运和代谢;G碳水化合物转运和代谢;H 辅酶转运和代谢;I脂质转运和代谢;J翻译、核糖体结构和生物发生;K转录;L复制、重组和修复;M细胞壁、细胞膜和细胞被膜的生物发生;O翻译后修饰、蛋白质转换和分子伴侣;P无机离子的转运和代谢;Q次生代谢产物的生物合成、转运和分解代谢;T信号转导机制;U细胞内转运、分泌和囊泡转运;V防御机制)
(2)内生菌群落功能多样性
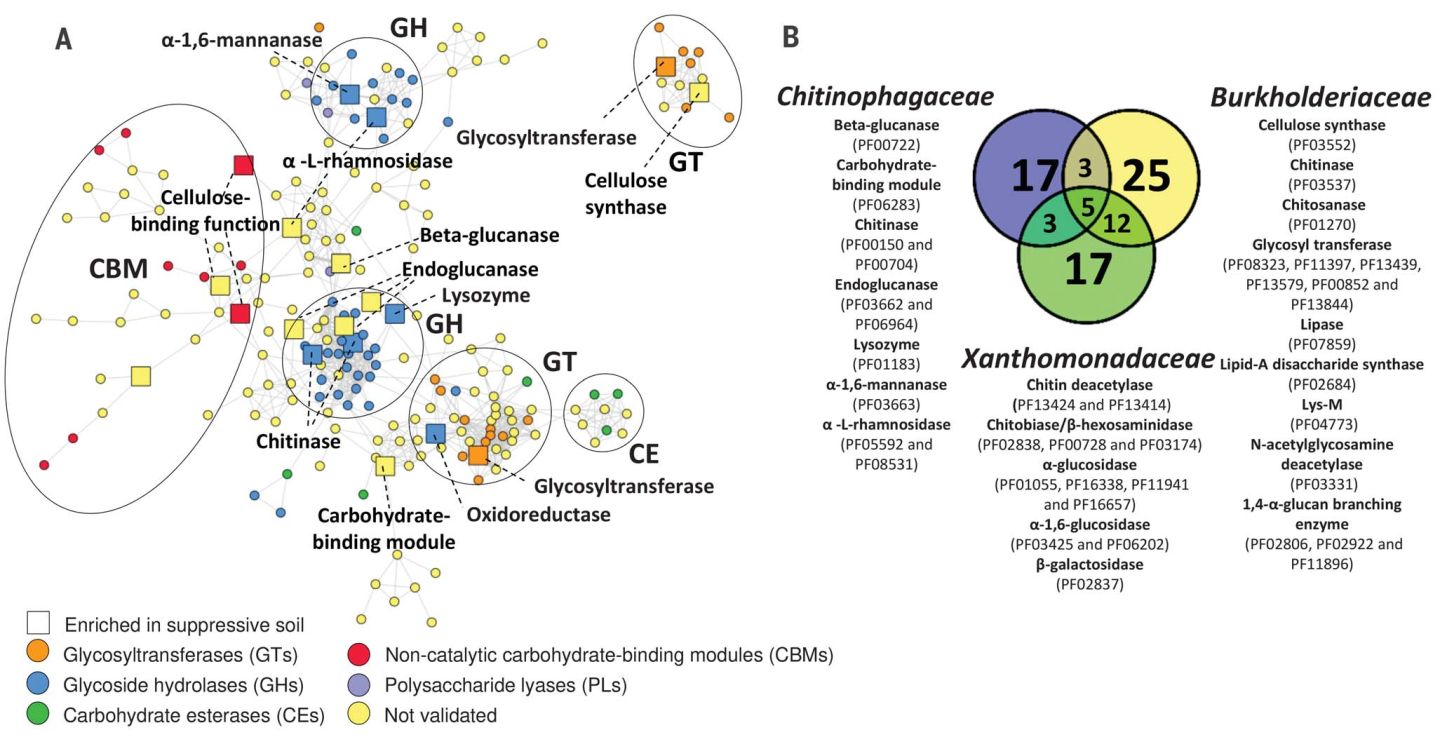
图2:内生菌群碳水化合物活性酶的多样性和分布
-
参与糖代谢(CAZymes)相关的已知蛋白和预测蛋白结构域的相似性网络。CAZymes对生长在抑病土壤S组或接种了真菌根病原菌R.solani的抑病土壤S+R组的植物内生菌进行注释,共得到1822个基因被注释为CAZymes。节点被分为五个功能类:糖苷水解酶(GH,蓝色)、糖基转移酶(GT,橙色)、多糖裂解酶(PL,紫色)、糖酯酶(CE,绿色)和非催化性碳水化合物结合组件(CBM,红色),未知结构域或功能尚未验证的蛋白结构标记为黄色。
-
三种富集在S+R组细菌的CAZymes注释数量韦恩图。Burkholderiaceae(黄色),Chitinophagaceae(蓝色)和Xanthomonadaceae(绿色)。
(3)内生菌群落生物合成基因簇的多样性和分布
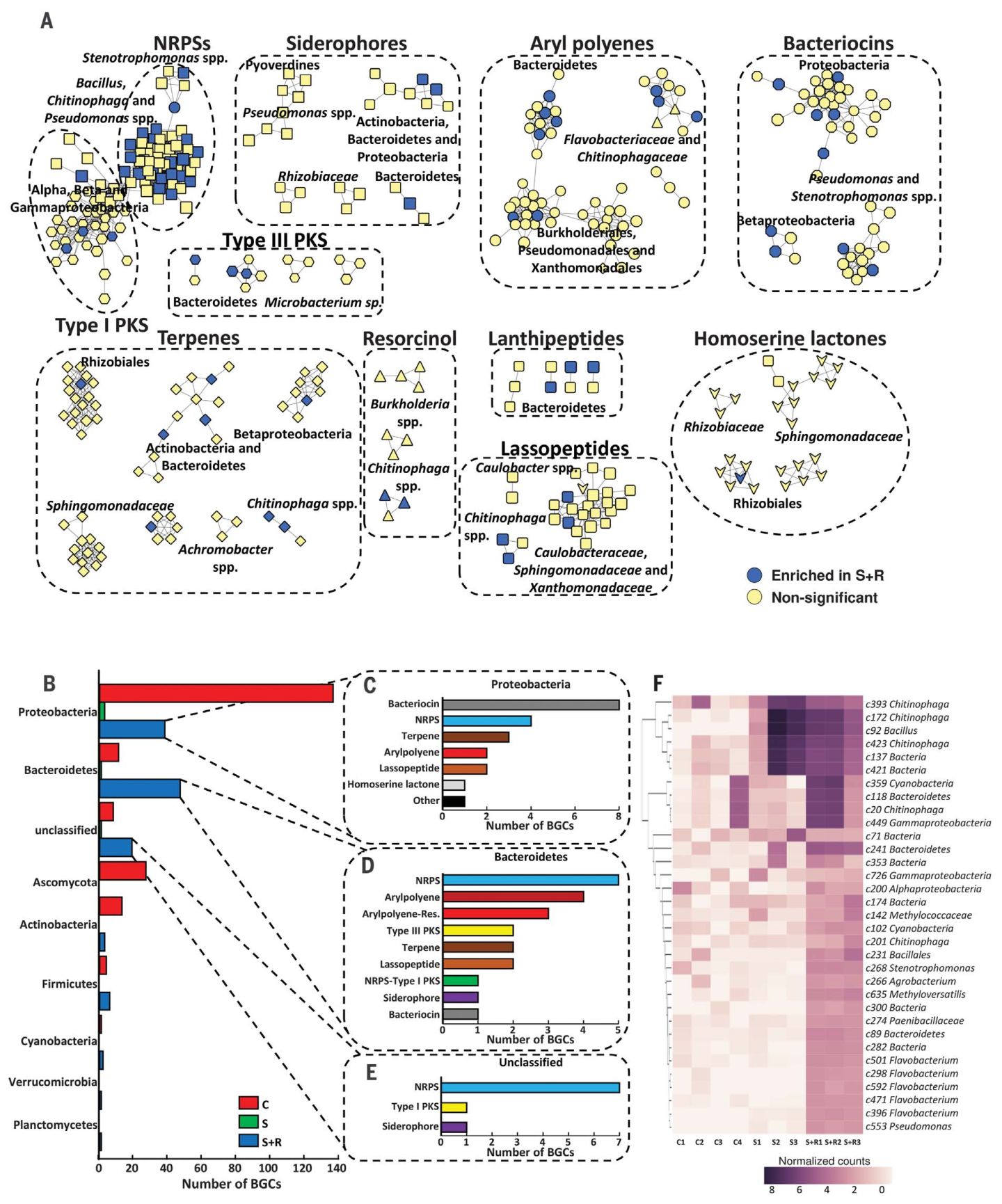
图3:内生菌群落生物合成基因簇的多样性和分布
-
在内生菌群中检测到的不同BGCs序列相似性网络。节点颜色代表基于Welch’s t test检验的显著性(FDR<0.1):黄色不显著;蓝色代表在S+R中显著上调的基因。
-
通过antiSMASH数据库使用Clusterfinder 算法检测到不同处理植物内生菌群过表达的BGCs。
(C-E)分别展示Proteobacteria(C)、Bacteroidetes (D) 和未分类unclassified(E)中显著在S+R中富集各类型BGCs的数量(不包含无法分类的基因)。
(F) 33个NRPS基因簇的相对丰度聚类热图。
(4)抑病微生物组的转录和功能分析
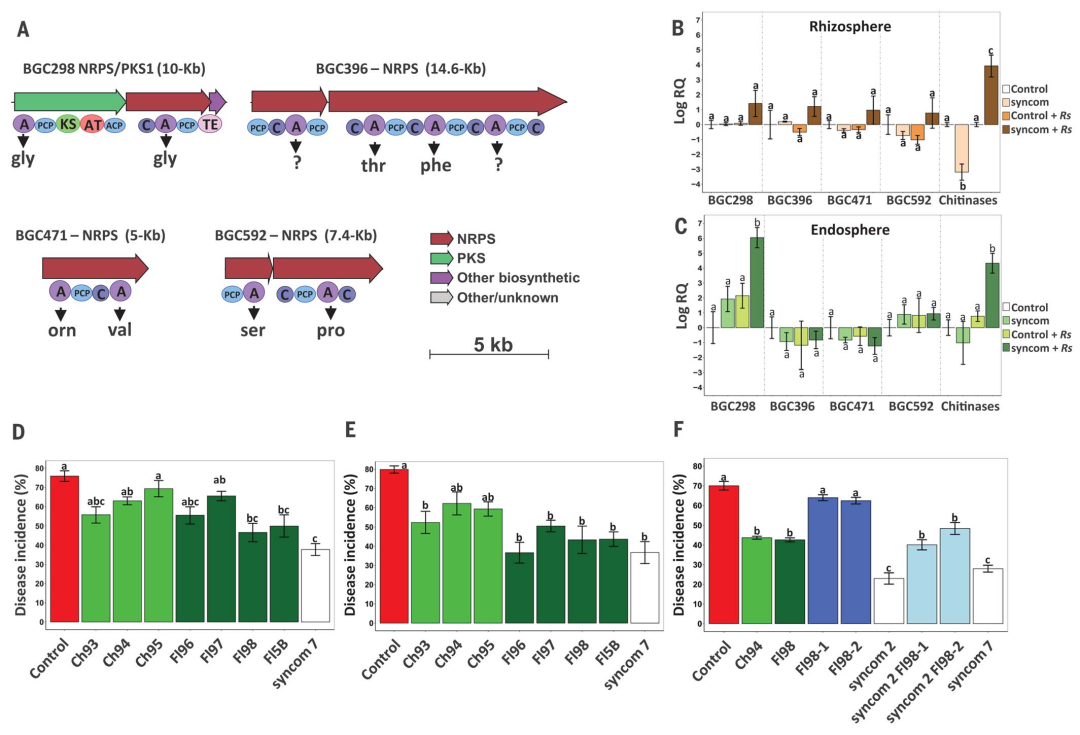
图4:抑病菌群的转录和功能分析
(A) 在黄杆菌MAG nbed44b64和四株分离的内生黄杆菌基因组序列中均鉴定出4个基因簇:BGC298、BGC396、BGC471和BGC592。图中展示的是NRPS和PKS基因家族编码蛋白组件和结构域。
(B-C)对几丁质菌和黄杆菌株分离物合成的内生syncom处理甜菜幼苗,使用qPCR对根际(rhizosphere)和根内(endosphere)的BGC298、BGC396、BGC471、BGC592和几丁质酶(GH18)基因表达进行定量分析。
(D-F)使用三株几丁质菌(Ch93, Ch94和 Ch95)和四株黄杆菌(Fl96, Fl97, Fl98和 Fl5B)单独或者组合验证甜菜发病率,实验表明这些处理菌降低了甜菜枯萎病发病率。
宏基因组测序分析关键图形
(1)宏基因组中提取的真菌reads整体分析
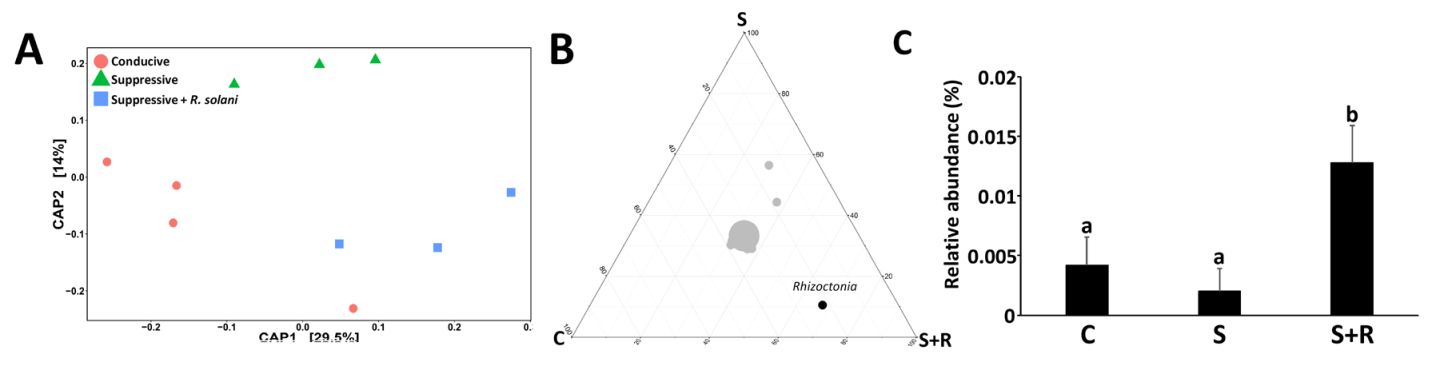
(2)甜菜内生菌群的多样性分析
(3)内生菌群宏基因组数据分箱
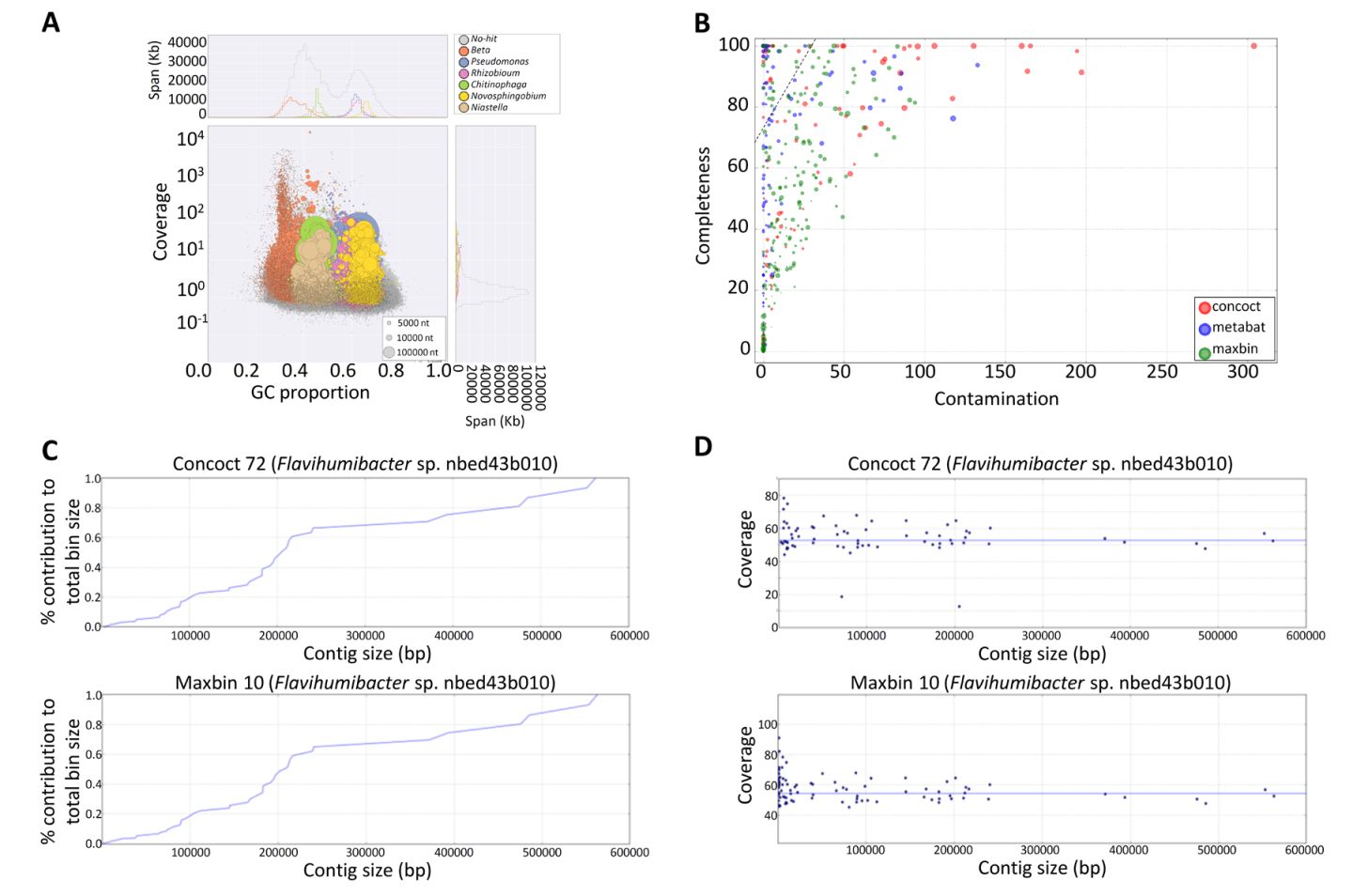
(4)内生菌宏基因组(MAG)与参考基因组比较
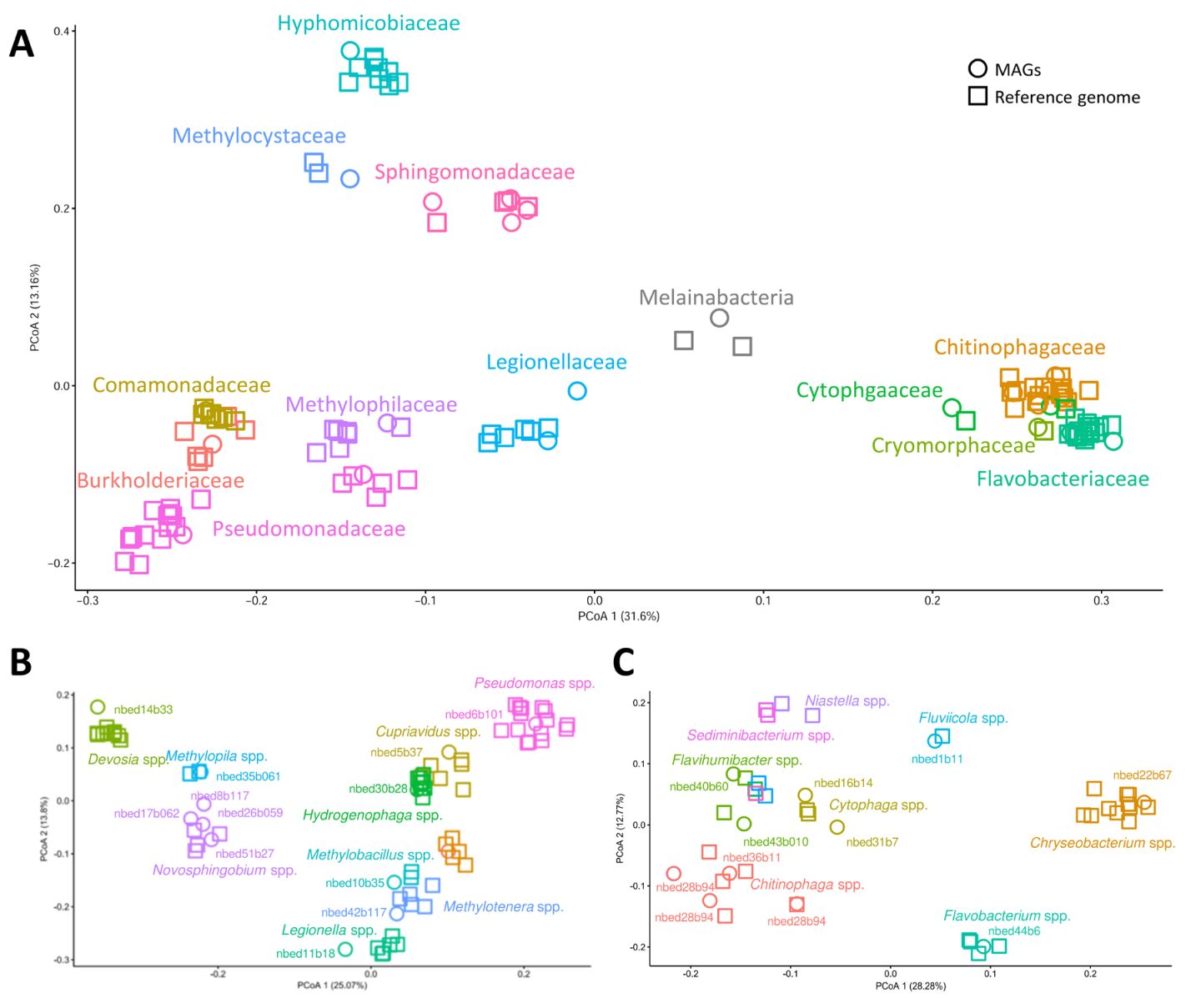
(5)分离内生菌株的细菌多样性
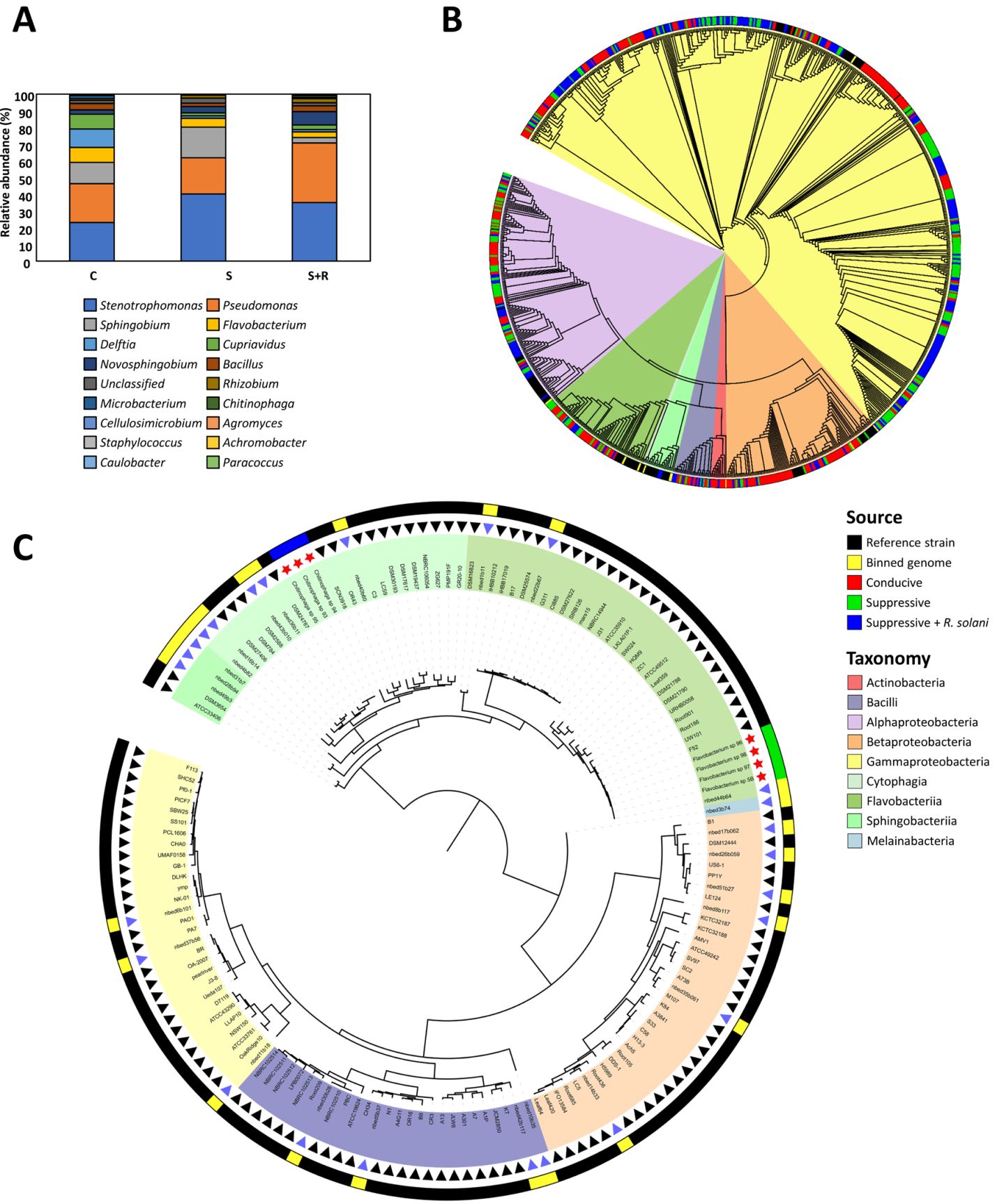
(6)测序的黄杆菌基因组和宏基因组组装基因组(MAG)的比较基因组学分析

结论:
本研究表明,在病原菌入侵植物根系的第二阶段,植物内生菌可以提供额外的保护层。在病原菌入侵时,几丁质菌(Chitinophagaceae)和黄杆菌(Flavobacteriaceae)家族成员在植物内富集,并表现出与真菌细胞壁降解相关的酶活性增强,以及NRPSs和PKSs编码的次生代谢产物生物合成。宏基因组序列重新组装(de novo)25个细菌基因组后,可以重建一个黄杆菌和几丁质菌的合成群落(syncom),从而提供疾病保护。定点诱变进一步验证黄杆菌BGC298对该表型的作用。这两个细菌在根组织内定位的位置以及它们在内胚层分子水平上的相互作用尚不清楚。几丁质酶产生的壳寡糖可能会诱导黄杆菌BGC298表达。本研究结果突出了内生根微生物组中丰富的未知微生物群落和功能性状。采用宏基因组分析和网络推断成功地确定了微生物群落的类别和功能,从而获得与特定微生物组相关的植物表型。
关于植物内生菌的宏基因组测序
植物内生菌中可培养的微生物种类十分有限,宏基因组学(metagenomics)是研究植物内生菌的有效手段之一。植物内生菌宏基因组研究通过不依赖于纯培养的宏基因组学(metagenomics)研究方法,构建植物内生菌的宏基因组文库,并从该文库中直接筛选获得生物合成基因簇,进一步鉴定其功能, 对植物内生菌进行定性及定量分析,为植物的微生物起源提供直接的证据。
易基因技术优势:
-
超深度检测:提取的全宏基因组DNA建立随机小片段文库,避免了目的片段的PCR过程,降低bias,随机测序,获取更丰富的序列信息。
-
植物组织表面菌处理:通过实验方法去除植物外表面菌株污染,准确测序植物内生菌种类、丰富及互作关系,真实还原构建微生态系统的功能网络。
-
16S全长检测:植物内生菌提取DNA后,通过16S全长扩增,质控内生菌提取效果。
研究方向:
植物内生菌宏基因组学研究为植物微生物起源、植物的营养和抗病育种提供内生菌角度的新策略。
有植物内生菌研究或组学研究的老师,可咨询了解。
参考文献:
Carrión VJ,et al.Pathogen-induced activation of disease-suppressive functions in the endophytic root microbiome. Science. 2019 Nov 1;366(6465):606-612.
