聚类是一种机器学习算法,它试图把数据集的观测值分为不同的簇。即相似观测值聚为簇,反之不相似的在不同簇中。聚类属于无监督,它尝试从数据集中发现结构,而不是预测响应变量的值。
聚类通常用于市场分析,例如某公司有下列信息:
- 家庭收入
- 家庭人数
- 户主职业
- 距市区距离
如果这些是有效信息,聚类可以识别类似家庭可能会购买一定产品或对某类广告响应较好。聚类最常用算法是KMeans,但需要预先设定聚类数量。
对应的层次聚类算法不需要预先设定聚类数量,它能够生成树形结构——称为系统树图。
介绍层次聚类
与KMeans类似,分层聚类的目标是产生相互非常相似的观测结果簇,而不同簇中的观测结果则截然不同。一般采用以下步骤进行分层聚类:
- 分别计算数据集中每两个变量的相异度
- 首先必须确定距离度量指标——如欧式距离,用于计算数据集中每个观测值之间的相异度
- 如果数据集包括n个观测值,那么共需要计算
n(n-1)/2次对相异度
- 将观测数据融合成簇
- 把上一步中两个最相似的观测值融合到一个簇中。
- 重复这个过程,直到所有观测到的都是一个大簇的成员。最终结果是一个树,可以绘制成系统树图。
为了确定两个簇之间的距离,我们可以使用下列几种方法,包括:
- 完全连接聚类: 求属于两个不同聚类的点之间的最大距离。
- 单连接聚类: 求属于两个不同聚类的点之间的最小距离。
- 均值连接聚类: 找出所有属于两个不同聚类的点之间的成对距离,然后计算平均值。
- 质心连接聚类: 求每个簇的质心,计算两个不同簇的质心之间的距离。
- ward最小方差法:最小化集群内的总方差,每一步都将簇间距离最小的一对簇进行合并。
- …
对于不同数据集,这些方法之间会有差异。下面通过示例逐步实现层次聚类算法。
加载包及数据
这两个包中有几个实现聚类算法的有用函数:
library(factoextra)
library(cluster)
#load data
df <- USArrests
#remove rows with missing values
df <- na.omit(df)
#scale each variable to have a mean of 0 and sd of 1
df <- scale(df)
#view first six rows of dataset
head(df)
# Murder Assault UrbanPop Rape
# Alabama 1.24256408 0.7828393 -0.5209066 -0.003416473
# Alaska 0.50786248 1.1068225 -1.2117642 2.484202941
# Arizona 0.07163341 1.4788032 0.9989801 1.042878388
# Arkansas 0.23234938 0.2308680 -1.0735927 -0.184916602
# California 0.27826823 1.2628144 1.7589234 2.067820292
# Colorado 0.02571456 0.3988593 0.8608085 1.864967207
该示例使用R 内置 USArrests 数据集,包括美国1973年每10万人的犯罪信息,包括谋杀、人身袭击、强奸、以及城镇人口率(Murder, Assault, Rape, UrbanPop)。
上面代码首先加载数据,接着删除缺失值行,最后对变量进行归一化,即统一为均值为0,标准差为1 .
确定连接方法
为了执行层次聚类方法,可以使用cluster 包中agnes()函数,语法如下:
** agnes(data, method)
- data : 数据集
- method:计算簇之间相异度的方法
因为事先不了解那个方法效果最好,我们定义函数使用不同方法执行层次聚类。该函数能够计算每种方法的凝聚系数,用于度量族的聚集程度,值越接近1,族凝聚度越高:
#define linkage methods
m <- c( "average", "single", "complete", "ward")
names(m) <- c( "average", "single", "complete", "ward")
#function to compute agglomerative coefficient
ac <- function(x) {
agnes(df, method = x)$ac
}
#calculate agglomerative coefficient for each clustering linkage method
sapply(m, ac)
# average single complete ward
# 0.7379371 0.6276128 0.8531583 0.9346210
从输出可以看到ward 方法效果最好,因此我们将使用该方法实现层次聚类:
#perform hierarchical clustering using Ward's minimum variance
clust <- agnes(df, method = "ward")
#produce dendrogram
pltree(clust, cex = 0.6, hang = -1, main = "Dendrogram")
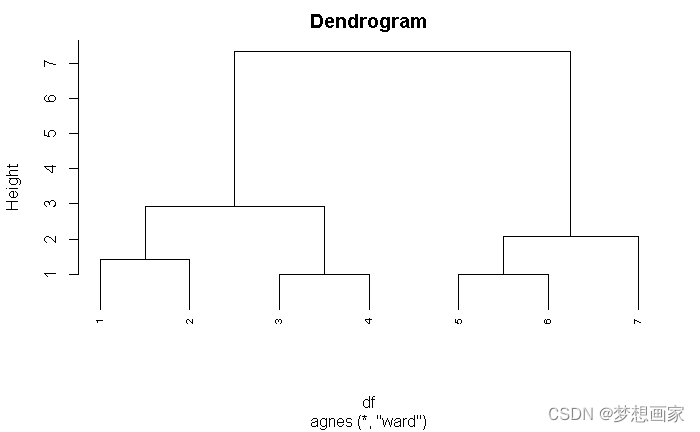
系统树图的每个叶子表示原始数据集中的观测值,从下往上,类似观测值融为枝,最后融合为一棵树。
确定最佳簇数
为了决定数据集分为几个簇,需要使用间隙统计指标,它比较在没有聚类的分布中,k的不同值的总簇内变异与它们的期望值。使用cluster包中的clusGap函数计算间隙统计指标,fviz_gap_stat()函数能够以图形方式展示簇数与指标之间的关系:
#calculate gap statistic for each number of clusters (up to 10 clusters)
gap_stat <- clusGap(df, FUN = hcut, nstart = 25, K.max = 10, B = 50)
#produce plot of clusters vs. gap statistic
fviz_gap_stat(gap_stat)
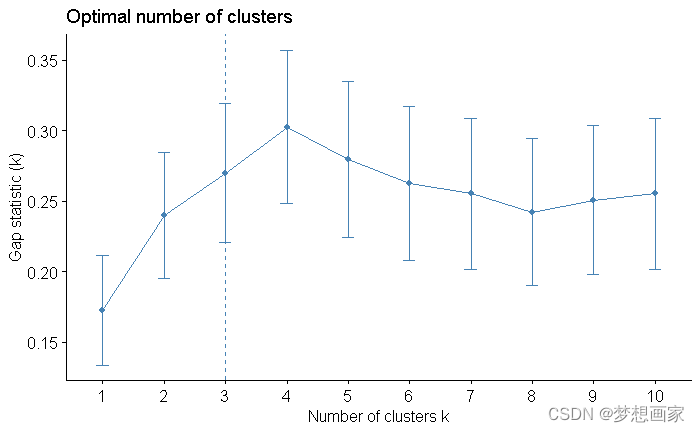
从图中我们可以看到,间隙统计指标在k = 4集群是最高的。因此,我们将选择将数据集分成4个不同簇。
给原始数据集增加分类
下面我们把数据分类结果增加至原始数据集中,使用cutree()方法把生成树切分为四个簇:
# 计算矩阵距离
d <- dist(df, method = "euclidean")
# 使用Ward方法执行层次聚类
final_clust <- hclust(d, method = "ward.D2" )
# 切分为四个簇
groups <- cutree(final_clust, k=4)
# 统计每个簇的数量
table(groups)
#
# 1 2 3 4
# 7 12 19 12
下面给原始数据每个州追加簇标签:
# 给原始数据追加簇标签
final_data <- cbind(USArrests, cluster = groups)
# 显示前6条数据
head(final_data)
# Murder Assault UrbanPop Rape cluster
# Alabama 13.2 236 58 21.2 1
# Alaska 10.0 263 48 44.5 2
# Arizona 8.1 294 80 31.0 2
# Arkansas 8.8 190 50 19.5 3
# California 9.0 276 91 40.6 2
# Colorado 7.9 204 78 38.7 2
最后使用aggregate函数计算每个簇的变量均值:
# 最后计算每个族的均值
aggregate(final_data, by=list(cluster=final_data$cluster), mean)
# cluster Murder Assault UrbanPop Rape cluster
# 1 1 14.671429 251.2857 54.28571 21.68571 1
# 2 2 10.966667 264.0000 76.50000 33.60833 2
# 3 3 6.210526 142.0526 71.26316 19.18421 3
# 4 4 3.091667 76.0000 52.08333 11.83333 4
从输出可以看到:
- 第1组的各州中,每10万公民中平均有14.67人被谋杀。
- 第1组中的各州,每10万公民中平均发生251.28起人身袭击事件。
- 第1组的各州中,居住在城市地区的居民的平均百分比为54.28%。
- 第1组的各州中,每10万公民中平均发生21.68起强奸案。
