学习笔记,仅供参考,有错必纠
Authors:Chen Shengquan,Zhang Boheng,Jiang Rui
文章目录
stPlus: a reference-based method for the accurate enhancement of spatial transcriptomics
Abstract
Motivation: 单细胞RNA测序(scRNA-seq)技术已经彻底改变了对单个细胞内转录组情况的调查. 最近空间转录组技术的进步使基因表达谱和细胞的空间组织图同时得以实现。在这些技术中,imaging- based可以提供更高的spatial resolutions,但它们受到检测的基因数量少或基因检测灵敏度低的限制。尽管已经提出了一些方法来提高spatially resolved transcriptomics,但基因表达预测的准确性不足和细胞群识别的能力不足仍然阻碍了这些方法的应用。
Results: 我们提出了stPlus,该方法利用scRNA-seq数据中的信息来增强空间转录组学. 基于一个具有自定义的损失函数的自编码器,stPlus执行联合嵌入,并通过weighted KNN预测空间基因表达。stPlus以更高的gene-wise 和 cell-wise Spearman correlation coefficients超越了baseline methods。我们还引入了一种基于聚类的方法来系统地评估增强性能。使用stPlus增强的数据,可以比使用测量数据更好地识别细胞群。scRNA-seq数据特有的基因预测表达也能很好地描述空间细胞的异质性。此外,stPlus对不同基因检测灵敏度水平、样本量和空间测量基因数量的数据集是稳健和可扩展的。
Availability and implementation: stPlus with detailed documents is freely accessible at http://health.tsinghua.edu. cn/software/stPlus/ and the source code is openly available on https://github.com/xy-chen16/stPlus.
Introduction
最近,单细胞RNA测序(scRNA-seq)技术的进展刺激了人们对细胞类型异质性(heterogeneity)的解读,彻底改变了对各种复杂组织的理解,并推动了现代细胞和分子生物学的发展。然而,scRNA-seq需要一个解离(dissociation step)步骤来获得细胞悬液( cell suspension),导致空间背景的丧失。空间的保持对于理解细胞特征和重建正常生理(normal physiology)或扰动(perturbation)下的组织结构至关重要.
为了阐明单细胞的异质性和定义细胞类型,同时也保留空间信息,最近开发了一些方法来描述空间分辨率的转录组学。这些方法可以分为两个主要类别:
(i) sequencing-based technologies that provide unbiased capture of the transcriptomic landscape via capturing and quantifying the mRNA population of molecules in situ, such as ST (Stahl et al., 2016), HDST (Vickovic et al., 2019) and Slide-seq (Rodriques et al., 2019)
(ii) imaging-based technologies,通过荧光原位杂交(FISH)或原位测序提供更高的空间分辨率,如osmFISH (Codeluppi et al., 2018)、seqFISHt (Eng et al., 2019)、MERFISH (Moffitt et al., 2018)和STARmap(Wang et al., 2018)。
这些方法通常是互补的,在目标吞吐量、覆盖率和空间分辨率方面有所不同. 例如,seq-based的方法提供相对较高的吞吐量,但存在空间分辨率的限制,而imaging-based的方法可以提供subcellular分辨率,但在序列覆盖率和整体吞吐量方面受到限制(Larsson et al., 2021; Zhuang, 2021). 在本研究中,我们侧重于imaging-based 的方法,认为提高空间分辨率有助于更准确地定义组织内的transcriptomic gradients,并允许检测转录物的亚细胞定位(Chen et al., 2021a).
在 imaging-based 的方法中,早期基于FISH的方法,如osmFISH,可以在组织成像中提供优秀的信号,甚至比scRNA-seq具有更高的基因检测灵敏度(Codeluppi et al., 2018). 尽管如此,分子拥挤问题对扩大成像的基因数量提出了挑战. 尽管最近的先进技术可以帮助缓解分子拥挤问题,对单个细胞中超过10000个基因进行成像(Eng et al., 2019; Xia et al., 2019),但一般来说,增加成像的基因数量将导致整体测量时间的增加和/或多路FISH和原位测序的测量精度降低(Zhuang, 2021). 因此,imaging-based的空间分辨率转录组学分析的一个更普遍的挑战是如何平衡成像的基因数量和基因检测灵敏度. 正如Wang et al. (2018)所建议的,一个基于分而治之思想的概念上的简单方法是将目标基因分为多组,并按顺序一次一组进行成像. 此外,计算方法可以帮助填补这一空白,将scRNA-seq数据作为参考,predicting genome-wide expression of cells profiled with a targeted gene set (cell atlas consortiums have generated massive amounts of scRNA-seq data).
加强空间分辨率转录组学的计算方法有望在the data with small number of genes imaged上predict expression of unmeasured genes,并effectively impute expression of imaged genes,以便在基因检测灵敏度低的数据上更好地识别cell populations. 现有的计算方法可以分为两大类.
-
概率方法,如gimVI(Lopez等人,2019年),通过领域适应性对空间转录组数据进行生成性建模,同时整合参考scRNA-seq
-
基于联合嵌入的方法,如Seurat(Stuart等人,2019年)和Liger(Welch等人,2019年),对空间数据和参考数据进行联合降维,然后根据这两个数据集中细胞之间的联系,估算空间转录组数据中未测量的基因
最近,SpaGE利用空间数据和scRNA-seq数据集之间共享的gene进行linearly joint embedding,然后通过k-NN方法预测空间基因表达(Abdelaal et al., 2020). 对于基于联合嵌入的方法,在第一步增强空间转录组数据方面起着最重要的作用,而第二步通常是基于一个总体思路,参考scRNA-seq数据中相邻细胞的信息(Abdelaal et al., 2020; Welch et al., 2019). Abdelaal等人还首次对现有的方法进行了评估,并证明SpaGE达到了整体上最先进的性能. 然而,只使用某一部分特征(即两个数据集中都存在的基因)不能充分利用参考scRNA-seq数据,从而限制了联合嵌入和数据增强的性能. 此外,虽然基于Spearman相关系数的评价可以在一定程度上评估预测的准确性,但它可能不是最佳的评价指标,因为the Spearman correlation coefficients are relatively low in most instances even the visual inspection shows good enhancement for genes with known spatial pattern (Abdelaal et al., 2020; Lopez et al., 2019). 此外,Spearman相关系数不能反映使用增强空间转录组数据识别细胞群的性能.
在上述认识的激励下,我们提出了stPlus,该方法用于增强空间转录组学数据. stPlus建立在一个自编码器的基础上,它有一个自定义的损失函数,用于利用scRNA-seq中的整体信息,而不仅仅是与空间转录组学数据共享的基因. 利用学到的 cell embeddings,stPlus通过weighted k-NN预测空间转录组学中的基因表达. 我们还引入了一种基于聚类的方法,通过不同指标来评估the cell heterogeneity maintained in the predicted spatial profiles. 我们通过gene和cell的Spearman相关系数以及细胞聚类的4个指标进行了综合评估. 在各种空间和scRNA-seq数据集对中与最先进的方法进行比较,stPlus提供了卓越的增强性能,并能扩展到大型数据集. 我们还证明,the predicted spatial gene expression can offer comparable or even better performance to identify cell populations than the measured spatially resolved transcriptomics. 我们预计stPlus将有助于缓解技术上的限制,更好地描述复杂组织的转录组模式.
Materials and methods
The model of stPlus
stPlus旨在通过predicting expression of unmeasured genes和imputing expression of measured genes来增强空间转录组学数据. stPlus的输入是the target spatial data和the reference scRNA-seq data,这些数据来自与空间数据相匹配或相似的组织. 这两个数据可以分别用两个gene-by-cell matrix 来表示. 注意,这两个数据之间的cell是不匹配的,reference data中的gene通常包括spatial data中的大部分基因. 用户可以从参考数据中指定任何要预测的基因. stPlus的输出是一个以cell为单位的gene矩阵,包含spatial data中每个细胞的每个指定基因的预测表达.
stPlus的增强过程可分为3个主要步骤(Fig. 1):
- data processing,为joint embedding做准备
- 对空间转录组数据和scRNA-seq数据中的单细胞进行joint embedding
- 根据cell embedding和scRNA-seq数据预测空间上无法测量的基因的表达
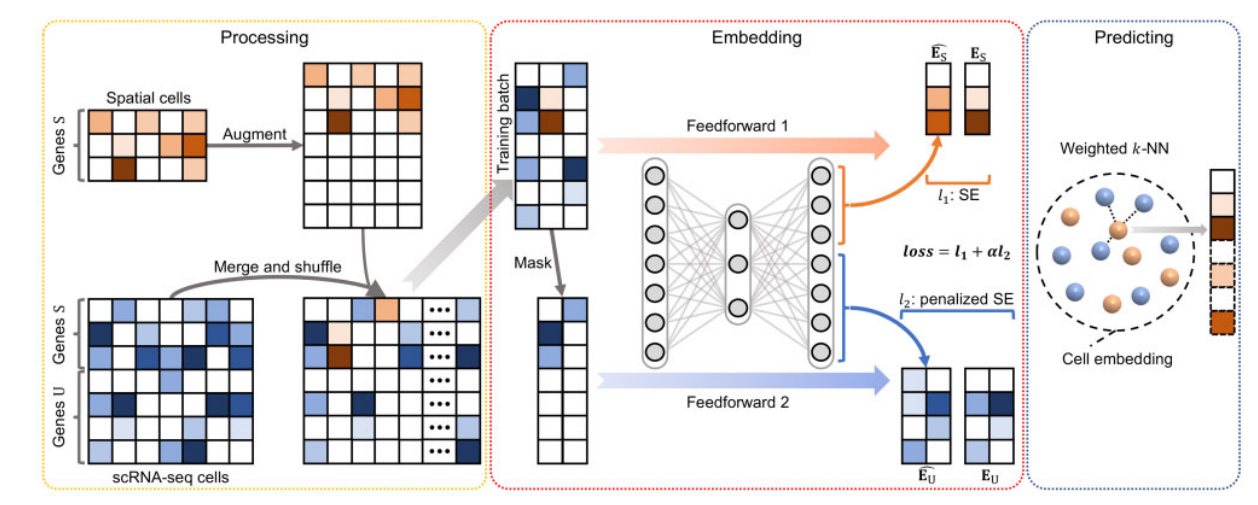
Fig. 1. stPlus模型的图形说明. stPlus首先增强空间转录组数据,将其与参考scRNA-seq数据相结合. 然后使用自编码器joint embedding这些数据. 最后,stPlus根据weighted k-NN预测空间上无法测量的基因的表达.
In the data processing step,为了避免引入高噪声基因,stPlus从只存在于scRNA-seq数据中的基因中选择前2000个高变量基因,这是广泛使用的scRNA-seq分析工具包的建议(Stuart et al., 2019; Wolf et al., 2018). 选定的基因集表示为 U U U,而空间转录组数据和参考scRNA-seq数据之间共享的基因集表示为 S S S. 对于空间转录组数据,stPlus用0来增加基因 U U U的表达测量值,并将基因顺序与经过处理的scRNA-seq数据的顺序统一起来. 然后,空间转录组和scRNA-seq数据集被merged together,在各细胞间进行shuffled,并进入下一步骤.
The joint embedding step旨在将每个空间转录组的cell与最相似的scRNA-seq细胞相匹配,但不进行single- cell data integration和batch-effect correction (Tran et al., 2020), or dimensionality reduction for downstream analyses such as data visualization and cell clustering. The joint embedding step在空间转录组数据的增强中起着最关键的作用(Abdelaal et al., 2020),而最先进的方法(线性嵌入)没有纳入scRNA-seq数据特有的基因信息,这对增强性能构成了主要障碍. 在这一步骤中,stPlus使用了一个自编码器和一个自定义的损失函数,以纳入scRNA-seq独有基因的生物学变化.
更具体地说,自动编码器用一个全连接层将维度为 S + U S+U S+U 的基因表达数据投射到一个1000维的潜空间. 然后用另一个全连接层将latent embedding decode到原始空间. 这两个层都使用ReLU激活函数,对负值输出为0,以实现非线性转换并克服梯度消失的问题. 作为stPlus的本质,损失函数由两部分组成:
(i) loss of the reconstruction for shared genes
S
S
S in the spatial transcriptomic data
(ii) data sparsity-penalized loss of the prediction for genes
U
U
U in the reference scRNA-seq data
To access the two parts of loss function, stPlus feedforwards each training batch twice, respectively. 首先,stPlus将batch传入自编码器,并提取对应空间转录组细胞的基因
S
S
S的解码结果(表示为
E
^
S
\hat{E}_S
E^S?). 利用空间转录组细胞中基因
S
S
S的原始表达数据(记为
E
S
E_S
ES?),损失函数的第一部分计算如下
l
1
=
∑
S
E
(
E
S
,
E
^
S
)
,
l_1 = \sum SE(E_S, \hat{E}_S),
l1?=∑SE(ES?,E^S?),
其中,$SE \left ( \cdot , \cdot \right ) $denotes squared error (squared L2 norm) between each element in the two inputs.
其次,为了模仿增强的空间转录组数据,stPlus提取batch中的scRNA-seq cell,并通过将gene
U
U
U (表示为
E
U
E_U
EU?,注意,这里的
U
U
U是没有设置为0的expression) 的expression设为0来masks the entries of genes
U
U
U. The masked scRNA-seq data被作为自动编码器的输入,解码器输出(表示为
E
^
U
\hat{E}_U
E^U?)被视为gene
U
U
U 在scRNA-seq cell中的预测结果. 因此,element-wise squared error可以通过
S
E
(
E
U
,
E
^
U
)
SE(E_U, \hat{E}_U)
SE(EU?,E^U?)得到. scRNA-seq数据通常包含技术artifacts (e.g. dropouts) ,不能反映实际表达水平,stPlus进一步惩罚了
S
E
(
E
U
,
E
^
U
)
SE(E_U, \hat{E}_U)
SE(EU?,E^U?),即通过参考scRNA-seq数据中非零元素的百分比(denoted as Q)来降低
E
U
E_U
EU?中零元素的误差,i.e. one minus data sparsity. 这一策略是基于这样的想法:scRNA-seq数据越稀疏,零元素越可能是假阴性,预测错误应该用较低的权重进行惩罚. 损失函数的第二部分被计算为以下公式
l
2
=
∑
[
S
E
(
E
U
,
E
^
U
)
⊙
Γ
]
,
l_2 = \sum \left [ SE(E_U, \hat{E}_U ) \odot \Gamma \right],
l2?=∑[SE(EU?,E^U?)⊙Γ],
where
⊙
\odot
⊙ denotes element-wise multiplication,
Γ
\Gamma
Γ denotes a matrix with the same dimension as
E
U
E_U
EU?, in which an entry is set to
Q
Q
Q if the corresponding entry in
E
U
E_U
EU? is zero, and is set to 1 otherwise. The two parts of loss function are then scaled by the number of gene sets
S
S
S (denoted as
E
S
E_S
ES?) versus that of
U
U
U (denoted as
E
U
E_U
EU?), and the number of spatial transcriptomic cells (denoted as
M
T
M_T
MT?) versus that of scRNA- seq cells (denoted as
M
R
M_R
MR?). The final loss is the sum of these two scaled losses:
l
o
s
s
=
l
1
+
α
l
2
=
l
1
+
N
S
M
T
N
U
M
R
l
2
.
loss = l_1 + \alpha l_2 = l_1 + \frac{N_S M_T}{N_U M_R} l_2 .
loss=l1?+αl2?=l1?+NU?MR?NS?MT??l2?.
After feedforwarding each training batch twice,stPlus performs backward propagation with the final loss. stPlus在自动编码器中优化参数直到收敛,最后得到空间转录组数据和参考scRNA-seq数据的cell embeddings.
from torch import nn
reconstruction_function = nn.MSELoss(reduction='sum')
def loss_recon_sparsity_func(recon_x, origi_x, data_quality):
"""
recon_x: reconstructed data
origi_x: original data
data_quality: user-specified or 1 minus the sparsity of scRNA-seq data (default)
"""
zero_ind = origi_x==0
non_zero_ind = ~zero_ind
recon_x_0 = recon_x[zero_ind]
recon_x_1 = recon_x[non_zero_ind]
origi_x_0 = origi_x[zero_ind]
origi_x_1 = origi_x[non_zero_ind]
MSE_0 = reconstruction_function(recon_x_0, origi_x_0)
MSE_1 = reconstruction_function(recon_x_1, origi_x_1)
MSE = data_quality * MSE_0 + MSE_1
return MSE
我们还介绍了stPlus的两种变种,即,stPlus without the first part of loss 和 stPlus without the second part of loss,以证明这两部分损失如何影响增强结果.
In the predicting step, stPlus predicts the expression of spatially unmeasured genes using a strategy similar to SpaGE. For each spatial transcriptomic cell
T
i
T_i
Ti?, stPlus calculates its cosine distance with each scRNA-seq cell
R
j
R_j
Rj? based on the learned cell embeddings. The neighboring 50 scRNA-seq cells are then used to predict the expression of unmeasured genes in cell
T
i
T_i
Ti? via a weighted k-NN approach. Specifically, stPlus filters out the neighbors with negative cosine similarity, and calculates the weight between
T
i
T_i
Ti? and kth neighbor in the remaining
K
K
K neighbors by:
w
i
k
=
(
1
?
d
i
s
t
a
n
c
e
(
T
i
,
R
k
)
∑
k
d
i
s
t
a
n
c
e
(
T
i
,
R
k
)
)
/
(
K
?
1
)
w_{ik} = \left ( 1-\frac{distance(T_i, R_k)}{\sum_k distance(T_i, R_k)} \right )/\left ( K-1 \right )
wik?=(1?∑k?distance(Ti?,Rk?)distance(Ti?,Rk?)?)/(K?1)
Finally, stPlus predicts expression of spatially unmeasured genes in cell
T
i
T_i
Ti? by the weighted expression of these genes in the
K
K
Kneighbors from reference scRNA-seq data:
Y
i
=
∑
k
w
i
k
X
k
Y_i = \sum_k w_{ik} X_k
Yi?=k∑?wik?Xk?
where
X
k
X_k
Xk? denotes expression data of genes to predict in the kth neighbor.
利用ensemble learning strategy,StPlus在训练自编码器时,采用five epochs with minimal loss,并对预测结果进行平均,以获得更好的性能.
Data collection and preprocessing
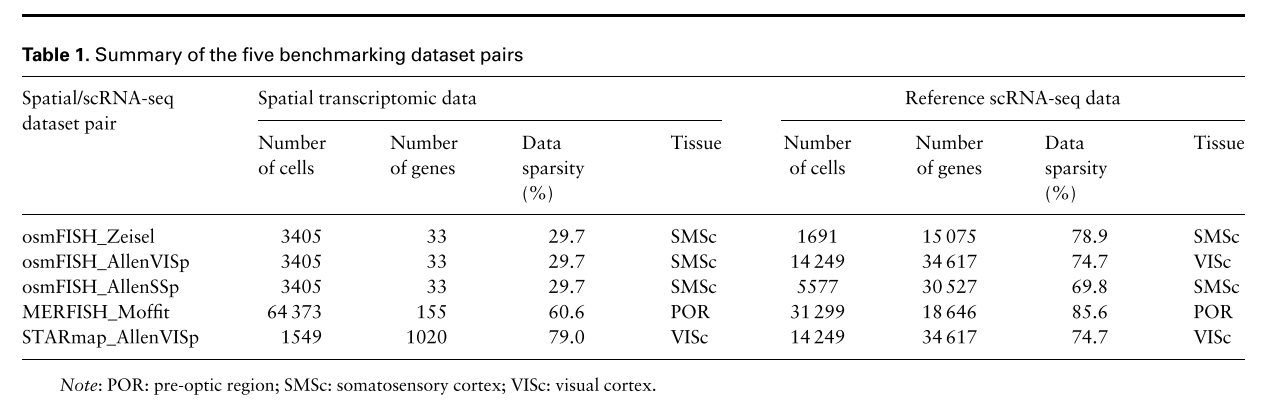
We collected the five benchmarking dataset pairs adopted in SpaGE (Abdelaal et al., 2020) to evaluate the enhancement performance of different computational methods. As shown in Table 1, the dataset pairs have diverse gene detection sensitivity levels, sample sizes and number of spatially measured genes. Specifically, the five dataset pairs are made up of three spatial transcriptomic datasets (osmFISH, MERFISH and STARmap) from different mouse brain regions, and four reference scRNA-seq datasets (Zeisel, AllenVISp, AllenSSp and Moffit). The osmFISH dataset was retrieved from http://linnarssonlab.org/osmFISH/ (Codeluppi et al., 2018). The MERFISH dataset was downloaded from https://doi.org/10.5061/dryad.8t8s248 (Moffitt et al., 2018). The STARmap dataset is available at https://www.starmapresources.com/data (Wang et al., 2018). Note that the labels of cell populations in the osmFISH and MERFISH datasets are accessible, while that of the STARmap dataset cannot be suc- cessfully aligned with cells. For the reference scRNA-seq data, the Zeisel dataset, which is provided by the same lab as that of the osmFISH dataset, was downloaded from http://linnarssonlab.org/cortex/ (Zeisel et al., 2015). The AllenVISp (Tasic et al., 2018) and AllenSSp (Chatterjee et al., 2018) datasets collected from https://portal.brain-map.org/atlases-and-data/rnaseq were more deeply sequenced than the Zeisel dataset. The AllenVISp was measured from a different brain region than the osmFISH dataset, while the AllenSSp dataset was measured from the somatosensory cortex, similar to the osmFISH dataset. The Moffit dataset, which was pub- lished in the same study of MERFISH, was retrieved from NCBI Gene Expression Omnibus with the accession number GSE113576 (Moffitt et al., 2018).
对于stPlus,除了最后的z-score步骤,我们均沿用了SpaGE的数据预处理步骤(Abdelaal et al., 2020). 更具体地说,我们过滤掉了空白基因和 F o s Fos Fos基因(非数值),并过滤掉了MERFISH数据集中被标记为 "模糊 "的细胞. 我们只保留了osmFISH数据集中来自皮质区域的细胞.然后,每个空间转录组数据集通过将每个细胞内的计数除以该细胞内的转录物总数进行归一化,以每个细胞的转录物median number为scaled,用pseudo-count进行log-transformed. 对于参考scRNA- seq数据集,我们过滤掉了在<10个细胞中表达的基因. 我们还根据元数据过滤掉了AllenVISp数据集中的低质量细胞("低质量 "和 "无类 "细胞),并根据元数据过滤掉了Zeisel数据集中的海马体细胞. 过滤后的scRNA-seq数据集接下来通过将每个细胞内的计数除以该细胞内的转录物总数,按10e6的比例进行独立归一化,并用pseudo-count进行log-transformed.
Evaluation approaches
考虑到imaging-based的空间分辨率转录组学分析的技术挑战是balance 成像的基因数量和基因检测灵敏度,预计用于数据增强的计算方法将:
-
准确预判未测量基因的表达,特别是对于成像的基因数量较少的数据
-
有效归纳imaged genes的表达以更好地识别cell populations,特别是对于基因检测灵敏度低的数据. 因此,我们从两个方面对增强性能进行了综合评价
首先,我们通过measured 和 predicted的spatial profiles之间的gene-wise 和 cell-wise Spearman相关系数来评估基因表达的预测性能. 由于预测对象是基因,the Spearman correlation coefficient over cells for each gene (gene-wise) can directly reflect the correlation between predicted and measured spatial profiles. 在两个专门用于增强空间转录组数据的最先进的方法中,the gene-wise coefficient被作为主要评价指标(Abdelaal et al., 2020; Lopez et al., 2019). 鉴于cell heterogeneity对单细胞研究至关重要,基于每个细胞更高的相关性可以更好地保持细胞特征,我们还通过每个细胞的基因上的Spearman相关系数(cell-wise)来评估预测的spatial profiles中保持的细胞特征. 我们在交叉验证实验中反复获得了所有基因的空间转录组预测,然后计算细胞的Spearman相关系数.
其次,我们通过4个聚类指标评估了细胞群的识别性能. 直观地看,基于the ground-truth cell labels和he enhanced spatial transcriptomic data,细胞聚类指标的得分越高,表明细胞群的识别性能越好,从而更好地增强了空间转录组学. 因此,我们评估了基于计算预测的空间转录组学的聚类结果,将基于spatially profiled data的聚类结果作为baseline. 具体来说,我们使用Scanpy(Wolf等人,2018)的标准管道与默认参数,这是一个广泛使用的用于分析单细胞数据的Python库,以进行dimension reduction 和 cell clustering. 我们采用了最近建议的基准研究的聚类策略(Chen等人,2019). 该策略基于Louvain聚类,这是一种基于群落检测的聚类方法(Blondel等人,2008;Levine等人,2015;Wolf等人,2018),并使用二进制搜索来调整Louvain聚类中的分辨率参数,使聚类的数量和基础真实细胞标签的数量尽可能接近。
Metrics for assessment of clustering results
聚类结果是根据4个广泛使用的指标进行评估的:调整的相互信息(AMI)、调整的Rand指数(ARI)、同质性(Homo)和归一化相互信息(NMI)。
Baseline methods
我们将stPlus的性能与四种基线方法进行了比较,包括SpaGE(Abdelaal et al., 2020)、Seurat(Stuart et al., 2019)、Liger(Welch et al., 2019)和gimVI(Lopez et al., 2019),其默认参数或设置在附带的例子中提供. 实现基线方法的源代码来自SpaGE的研究,该研究首次使用上述介绍的5个数据集对现有方法进行系统的基准测试(Abdelaal等人,2020). 数据处理程序,如归一化和缩放,也是按照每个方法的源代码进行的.