不会 transformer 没关系,本教程开箱即用。
Tina姐总算对transformer下手了,之前觉得难,因为刚开始学序列模型的时候就没学会。然后就一直排斥学transformer。
这两周没什么事,加上MONAI有现成的教程,就打算先跑通后,再学理论。然后,顺利的跑通了代码,再学了一周理论,发现它也不过如此嘛,入门还是很容易的。
有同学想了解的transformer的话,可以先看完这个实战教程,如果感兴趣,后续会出一个transformer入门路线。
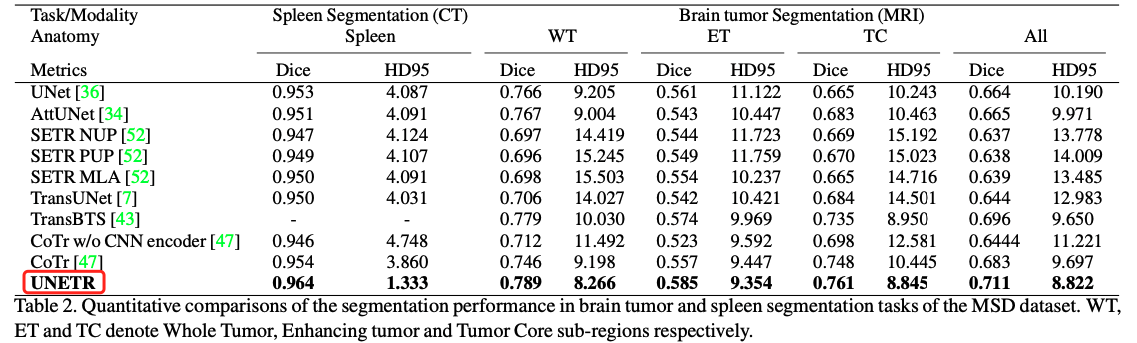
UNETR介绍
利用纯Transformers作为编码器来学习输入量的序列表示并有效地捕获全局多尺度信息。同时也遵循了编码器和解码器的成功的“U型”网络设计。Transformers编码器通过不同分辨率的跳跃连接直接连接到解码器,以得到最终的分割结果。
使用多器官分割的BTCV数据集、医学分割十项全能(MSD)数据集广泛验证了提出的模型在不同成像方式(即MR和CT)上对体积脑肿瘤和脾脏分割任务的性能,并且结果始终证明了良好的性能。
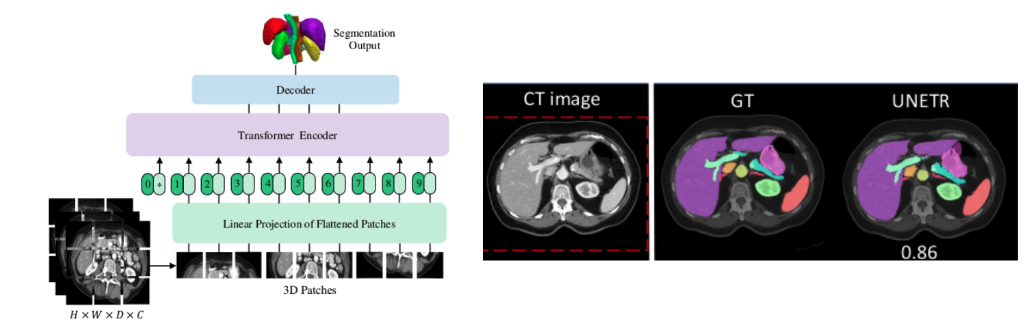
论文中,各数据集的分割结果如下
- BTCV腹部多器官分割结果
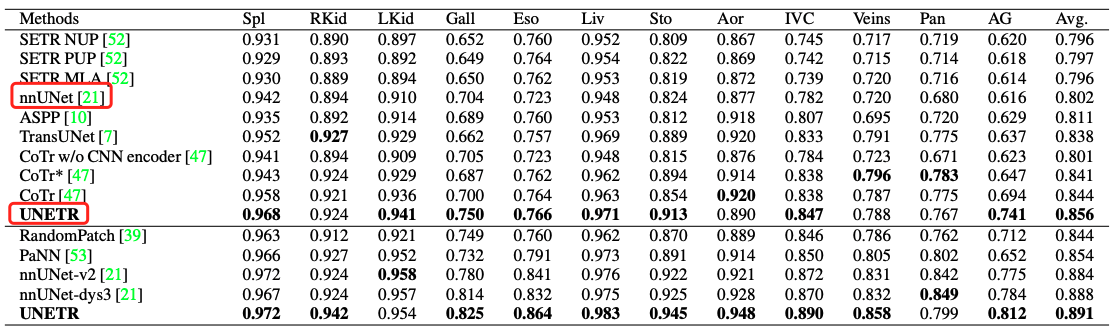
- MSD数据集上:脾脏分割和脑肿瘤分割
可以看到,UNETR在腹部多器官上打败了nnUet。
UNETR的模型结构
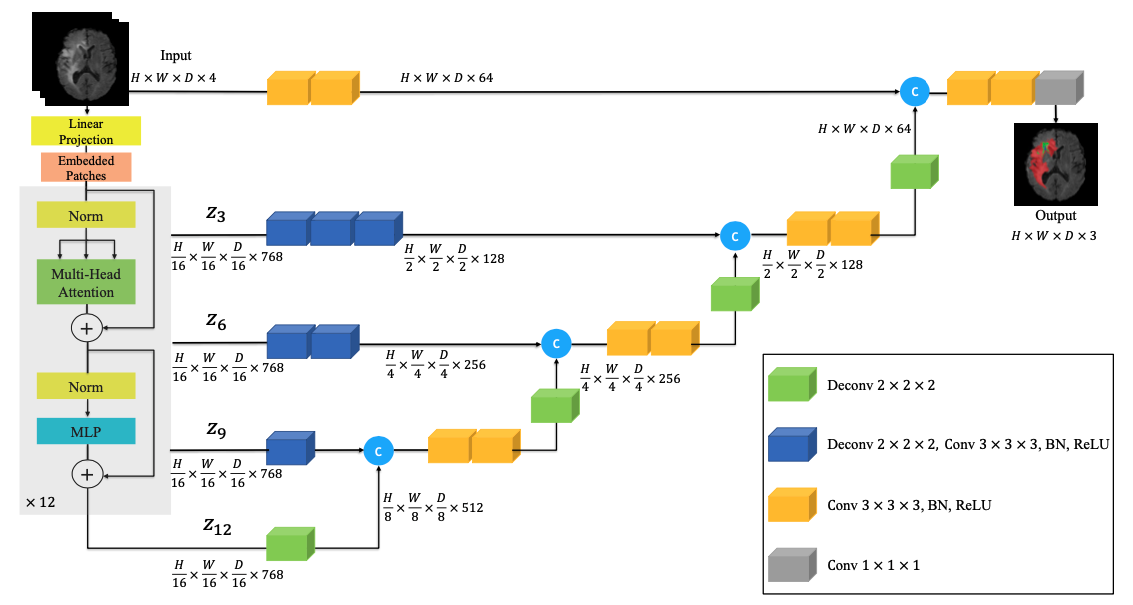
如果没学过Transformer,就把这个图当成Unet来看,左边是下采样,尺寸不断缩小,右边是上采样,尺寸不断扩大。中间是跳跃连接。
只不过这里的下采样用的是Transformer。
有了大概的了解,回归我们的实战吧
实战阶段
本教程代码连接:MONAI UNETR tutorial
把它下载下来,边跑边看我的解说,食用效果更佳🤭
主要包含以下部分:
- 字典格式数据的转换。
- 数据增强变换:根据 MONAI transform API 定义一个新的 transform。
- 从文件夹加载数据。
- 缓存 IO 和转换以加速训练和验证。
- 3D UNETR 模型、DiceCE 损失函数、多器官分割任务的平均 Dice 度量。
首先,下载数据,数据地址:BTCV挑战赛
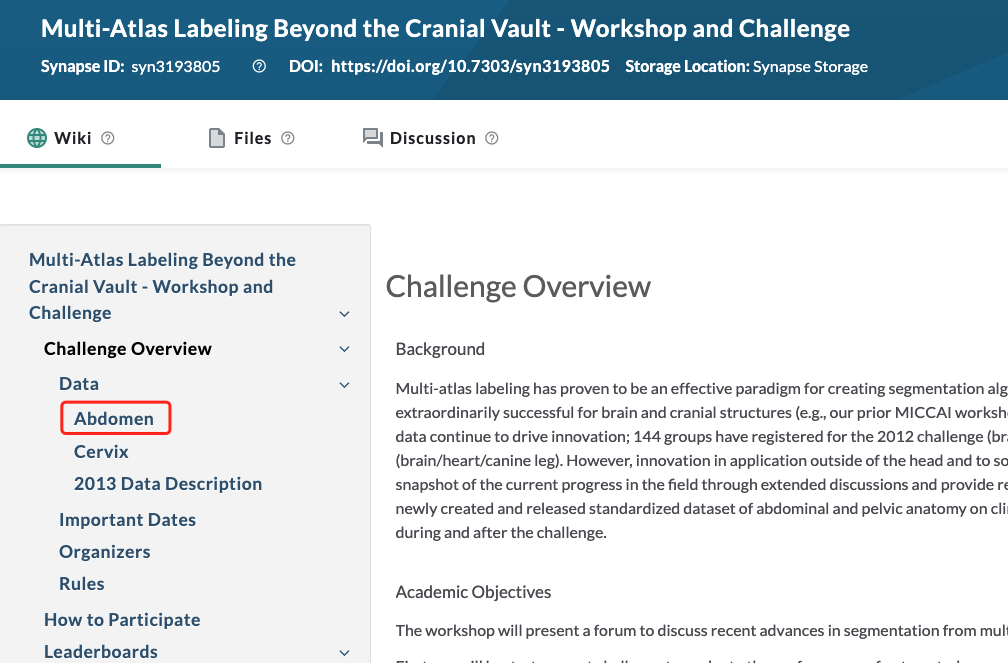
注意:从这个链接进去,很难找到数据,我也找了2天才找到,具体怎么点进去的我也忘了。好像是要加入比赛,才能到下载数据的地方。
不过,不用担心。找不到下载连接的,可以去我网盘下载:
链接: https://pan.baidu.com/s/1-0yMfZ4grBF5UYlRp1t_Rw 提取码: ejfp
了解数据
在机构审查委员会 (IRB) 的监督下,从正在进行的结直肠癌化疗试验和回顾性腹疝研究的组合中随机选择了 50 份腹部 CT 扫描。 其中,30个用于训练,20个用于测试。
- 体积:512 x 512 x 85 - 512 x 512 x 198
- 视野:280 x 280 x 280 mm3 - 500 x 500 x 650 mm3
- 平面分辨率:0.54 x 0.54 mm2 - 0.98 x 0.98 mm2
- z轴分辨率:2.5 mm 到 5.0 mm
- target: 一共是分割13种器官:1. 脾脏 2. 右肾 3. 左肾 4. 胆囊 5. 食道 6. 肝脏 7. 胃 8. 主动脉 9. 下腔静脉 10. 门静脉和脾静脉 11. 胰腺 12 右肾上腺 13 左肾上腺。
- 训练集的30个数据,再次划分为 24 Training + 6 validation
环境准备
确保MONAI版本在0.6以上,最好是跟新到最新版本。
如果你的环境ok, 这一步可以省略。不用运行
加载各种需要的包
import os
import shutil
import tempfile
import matplotlib.pyplot as plt
import numpy as np
from tqdm import tqdm
from monai.losses import DiceCELoss
from monai.inferers import sliding_window_inference
from monai.transforms import (
AsDiscrete,
AddChanneld,
Compose,
CropForegroundd,
LoadImaged,
Orientationd,
RandFlipd,
RandCropByPosNegLabeld,
RandShiftIntensityd,
ScaleIntensityRanged,
Spacingd,
RandRotate90d,
ToTensord,
)
from monai.config import print_config
from monai.metrics import DiceMetric
from monai.networks.nets import UNETR
from monai.data import (
DataLoader,
CacheDataset,
load_decathlon_datalist,
decollate_batch,
)
import torch
print_config()
这一步,如果提示缺什么包,就安装什么包
设置模型保存环境
root_dir = './checkpoints'
if not os.path.exists(root_dir):
os.makedirs(root_dir)
print(root_dir)
原教程这里使用的是临时地址,这里我们改一下,保存好训练好的模型
设置训练集和验证集的transform
train_transforms = Compose(
[
LoadImaged(keys=["image", "label"]),
AddChanneld(keys=["image", "label"]),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(
keys=["image", "label"],
pixdim=(1.5, 1.5, 2.0),
mode=("bilinear", "nearest"),
),
ScaleIntensityRanged(
keys=["image"],
a_min=-175,
a_max=250,
b_min=0.0,
b_max=1.0,
clip=True,
),
CropForegroundd(keys=["image", "label"], source_key="image"),
RandCropByPosNegLabeld(
keys=["image", "label"],
label_key="label",
spatial_size=(96, 96, 96),
pos=1,
neg=1,
num_samples=4,
image_key="image",
image_threshold=0,
),
RandFlipd(
keys=["image", "label"],
spatial_axis=[0],
prob=0.10,
),
RandFlipd(
keys=["image", "label"],
spatial_axis=[1],
prob=0.10,
),
RandFlipd(
keys=["image", "label"],
spatial_axis=[2],
prob=0.10,
),
RandRotate90d(
keys=["image", "label"],
prob=0.10,
max_k=3,
),
RandShiftIntensityd(
keys=["image"],
offsets=0.10,
prob=0.50,
),
ToTensord(keys=["image", "label"]),
]
)
val_transforms = Compose(
[
LoadImaged(keys=["image", "label"]),
AddChanneld(keys=["image", "label"]),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(
keys=["image", "label"],
pixdim=(1.5, 1.5, 2.0),
mode=("bilinear", "nearest"),
),
ScaleIntensityRanged(
keys=["image"], a_min=-175, a_max=250, b_min=0.0, b_max=1.0, clip=True
),
CropForegroundd(keys=["image", "label"], source_key="image"),
ToTensord(keys=["image", "label"]),
]
)
可以看到,transformer的这部分同CNN没有区别。该怎么处理数据还怎么处理。
按要求下载好数据
前面已经介绍了如何下载数据,在这里确保数据满足代码要求的格式。
将下载好的数据解压,放在项目的 ./data下
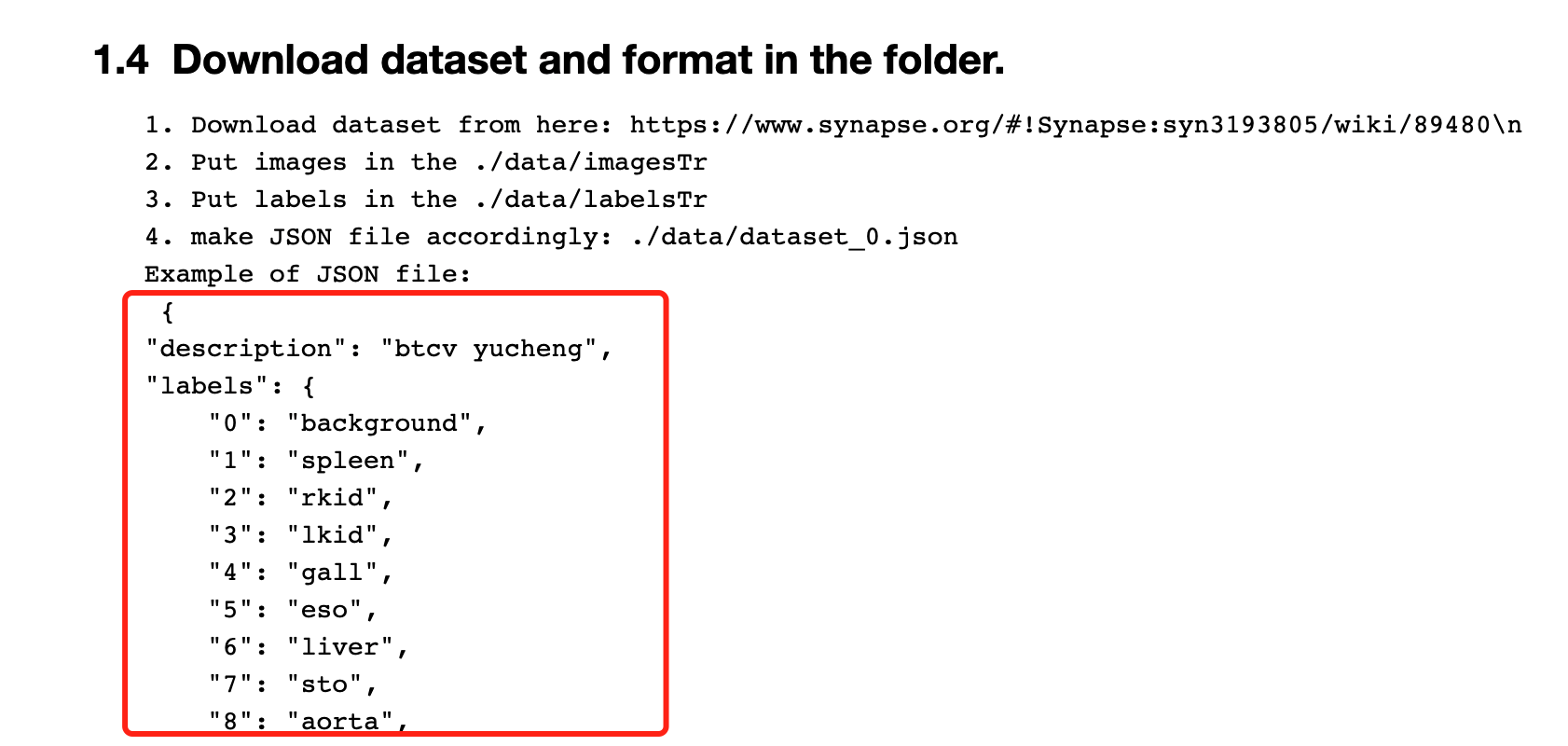
把方框及以下的所有内容复制到json文件,命名为dataset_0.json
如何创建json文件:百度一下即可,很简单,哈哈😋
构建Dataloader
data_dir = "data/"
split_JSON = "data/dataset_0.json"
# data_dir = "/home/ali/Desktop/data_local/Synapse_Orig/"
# split_JSON = "dataset_0.json"
datasets = data_dir + split_JSON
datalist = load_decathlon_datalist(datasets, True, "training")
val_files = load_decathlon_datalist(datasets, True, "validation")
train_ds = CacheDataset(
data=datalist,
transform=train_transforms,
cache_num=24,
cache_rate=1.0,
num_workers=8,
)
train_loader = DataLoader(
train_ds, batch_size=1, shuffle=True, num_workers=8, pin_memory=True
)
val_ds = CacheDataset(
data=val_files, transform=val_transforms, cache_num=6, cache_rate=1.0, num_workers=4
)
val_loader = DataLoader(
val_ds, batch_size=1, shuffle=False, num_workers=4, pin_memory=True
)
这里需要注意,你的数据地址要写对。
然后运行下一个cell,检查一下数据是否正确

构建模型,损失函数,优化器
os.environ["CUDA_DEVICE_ORDER"] = "PCI_BUS_ID"
device = torch.device("cuda" if torch.cuda.is_available() else "cpu")
model = UNETR(
in_channels=1,
out_channels=14,
img_size=(96, 96, 96),
feature_size=16,
hidden_size=768,
mlp_dim=3072,
num_heads=12,
pos_embed="perceptron",
norm_name="instance",
res_block=True,
dropout_rate=0.0,
).to(device)
loss_function = DiceCELoss(to_onehot_y=True, softmax=True)
torch.backends.cudnn.benchmark = True
optimizer = torch.optim.AdamW(model.parameters(), lr=1e-4, weight_decay=1e-5)
这里,就是把model换成了UNETR,其余也同CNN没有区别。
然后就是一个典型的pytorch训练。代码比较长,我就不粘贴了。

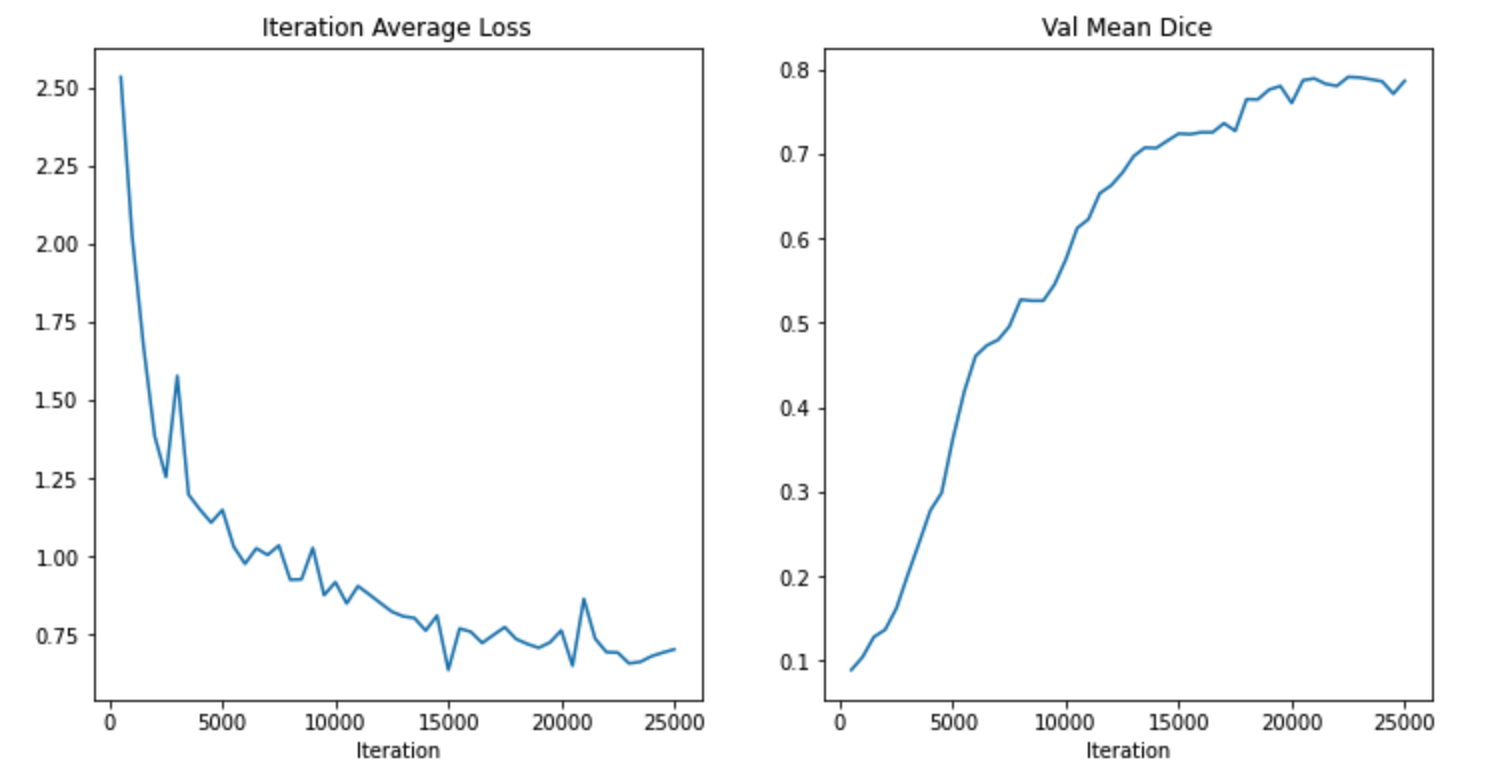
我训练的结果是0.7907,还不错。也算是初尝到了transformer的优点。
大家赶快动手试试吧~~
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连
