TorchDrug–药物属性预测
在本教程中,我们将学习如何使用 TorchDrug 训练图神经网络以进行分子特性预测。属性预测旨在根据分子的图形结构和特征预测分子的化学性质。
准备数据集
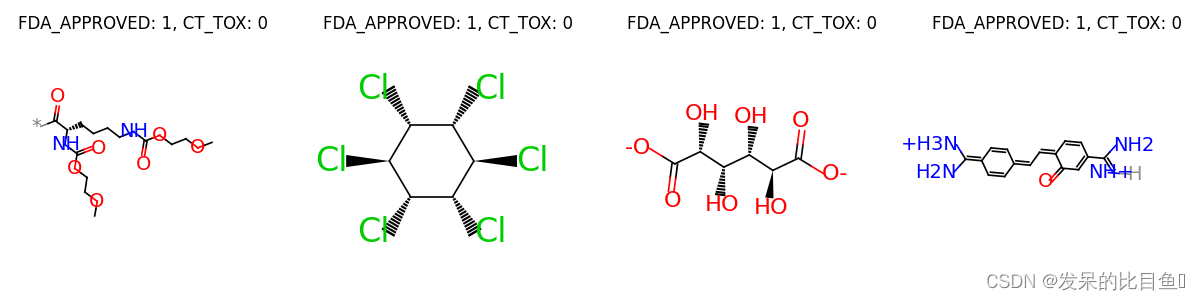
我们使用ClinTox数据集进行说明。ClinTox包含 1,484 个分子,在临床试验中标有 FDA 批准状态和毒性状态。
在这里,我们下载数据集并将其拆分为训练、验证和测试集。训练集/有效集/测试集的分割分别为 80%、10% 和 10%。
import torch
from torchdrug import data, datasets
dataset = datasets.ClinTox("~/molecule-datasets/")
lengths = [int(0.8 * len(dataset)), int(0.1 * len(dataset))]
lengths += [len(dataset) - sum(lengths)]
train_set, valid_set, test_set = torch.utils.data.random_split(dataset, lengths)
让我们可视化数据集中的一些样本。
graphs = []
labels = []
for i in range(4):
sample = dataset[i]
graphs.append(sample.pop("graph"))
label = ["%s: %d" % (k, v) for k, v in sample.items()]
label = ", ".join(label)
labels.append(label)
graph = data.Molecule.pack(graphs)
graph.visualize(labels, num_row=1)
定义我们的模型
该模型由两部分组成,一个与任务无关的图表示模型和一个特定于任务的模块。我们定义了一个具有 4 个隐藏层的图同构网络 (GIN) 作为我们的表示模型。两个预测任务将通过任务特定模块的多任务训练共同优化。
from torchdrug import core, models, tasks, utils
model = models.GIN(input_dim=dataset.node_feature_dim,
hidden_dims=[256, 256, 256, 256],
short_cut=True, batch_norm=True, concat_hidden=True)
task = tasks.PropertyPrediction(model, task=dataset.tasks,
criterion="bce", metric=("auprc", "auroc"))
训练和测试
现在我们可以训练我们的模型了。我们为我们的模型设置了一个优化器,并将所有内容放在一个 Engine 实例中。训练我们的模型可能需要几分钟。
optimizer = torch.optim.Adam(task.parameters(), lr=1e-3)
solver = core.Engine(task, train_set, valid_set, test_set, optimizer,
gpus=[0], batch_size=1024)
solver.train(num_epoch=100)
solver.evaluate("valid")
模型训练完成后,我们会在验证集上对其进行评估。结果可能类似于以下内容。
auprc [CT_TOX]: 0.455744
auprc [FDA_APPROVED]: 0.985126
auroc [CT_TOX]: 0.861976
auroc [FDA_APPROVED]: 0.816788
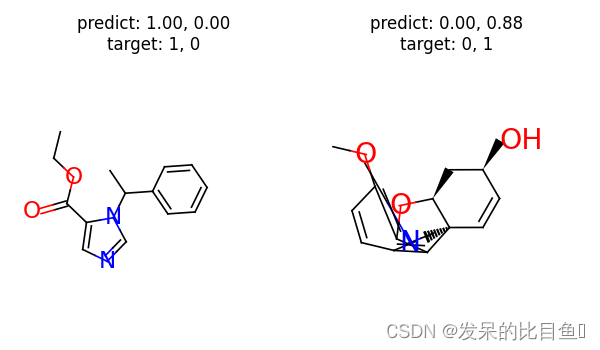
为了对模型有一些直觉,我们可以研究模型的预测。以下代码为每个类别选择一个样本,并绘制结果。