HNF-Netv2 for Brain Tumor Segmentation using multi-modal MR Imaging
使用多模态MR成像分割脑肿瘤的HNF-Netv2
Published : Jan 2022
论文:https://arxiv.org/abs/2202.05268
代码:暂无
摘要:
??在之前的工作中,作者利用HNF-Net、高分辨率的特征表示和轻量化的非局部自注意机制,利用多模态MR成像对脑肿瘤进行分割。在本文中,作者通过添加尺度间和尺度内的语义识别增强块,将HNF-Net扩展到HNF-Netv2,进一步利用所获得的高分辨率特征进行全局语义识别。作者在多模态脑肿瘤分割挑战(BraTS) 2021数据集上训练和评估作者的HNF-Netv2。测试集的结果显示,作者的HNF-Netv2的平均Dice得分为0.878514、0.872985、0.924919,增强肿瘤、肿瘤核心和整个肿瘤的Hausdorff距离(95%)分别为8.9184、16.2530和4.4895。作者的方法获得了RSNA 2021脑瘤人工智能挑战奖(分割任务),在所有1250个提交的结果中排名第八。
问题动机:
??脑胶质瘤是最常见的原发性脑恶性肿瘤,通常包含异质性的组织学亚区,如水肿/侵袭、肿瘤结构活跃、囊性/坏死成分和无增强的粗大异常。利用多模态磁共振(MR)成像对这些固有子区域进行精确和自动化的分割,对潜在的诊断和治疗这种疾病是至关重要的。为此,开展了多年的多模态脑肿瘤分割挑战(multi-modal brain tumor segmentation, BraTS),为评价目前最先进的脑肿瘤子区域分割方法提供了一个平台。
??随着深度学习在医学图像分析中的广泛应用,基于全卷积网络(fully convolutional network, FCN)的分割方法被设计用于该分割任务,并在之前的挑战中表现出了令人信服的性能。Kamnitsas等人构建了一个3D双路径CNN,即DeepMedic,它采用双路径架构,同时对输入图像进行多尺度处理,从而利用局部和全局的上下文信息。DeepMedic还使用3D全连接条件随机场来消除误报。Isensee等人使用带有实例归一化和leaky ReLU激活的3D U-Net,结合组合损失函数和基于区域的训练策略,实现了出色的分割性能。Myronenko等人将一种基于变分自编码器(V AE)的重构解码器加入到3D U-Net中,对共享编码器进行正则化,并在2018年BraTS中取得了第一名的分割性能。在BraTS 2019中,Jiang et al.提出了一种两级级联的U-Net模型,将脑肿瘤从粗到细划分子区域,其中第二级模型有更多的信道数,并使用两个解码器来提高性能。该方法在BraTS 2019分割任务中取得了最佳的分割效果。在作者之前的工作中,作者提出了一种高分辨率和非局部特征网络(HNF-Net),用于在多模态MR图像中分割脑肿瘤。该HNF-Net主要基于并行多尺度融合(PMF)模块构建,能够保持高分辨率的特征表示,聚合多尺度上下文信息。该模型还引入了期望最大化注意(EMA)模块,以可接受的计算复杂度为代价,增强了长期依赖的空间上下文信息。在BraTS 2020挑战赛中,作者设计了一个两阶段级联的HNFNet,从而构建了一个混合高分辨率和非局部特征网络(H2NF-Net),该网络使用单个和级联模型来分割不同的脑肿瘤子区域。提议的H2NF-Net在78名参与者中赢得了BraTS 2020挑战分段任务的第二名。
??浅层阶段的特征具有更详细的空间信息,而深层阶段的特征具有更强的语义识别能力。由于作者的PMF模块,在原始的HNF-Net中,高分辨率特征得到了很好的维护,但由于当前网络的全局上下文学习能力有限,对所获得的特征进行语义识别可能不够充分。为了解决这些问题,在本文中,作者进一步提出了一种HNF-Netv2,以提高这一挑战性任务的分割性能。具体而言,作者通过添加尺度间和尺度内的语义识别增强块(尺度间SDE和尺度内SDE)来扩展原有的HNF-Net,从而利用全局上下文增强当前具有语义识别的高分辨率特征。作者在BraTS 2021挑战数据集上评估了所提出的HNF-Netv2,在验证集和测试集上的结果表明作者的方法具有卓越的分割性能,而在训练集上的消融研究表明了所提出的尺度内和尺度间SDE块的有效性。
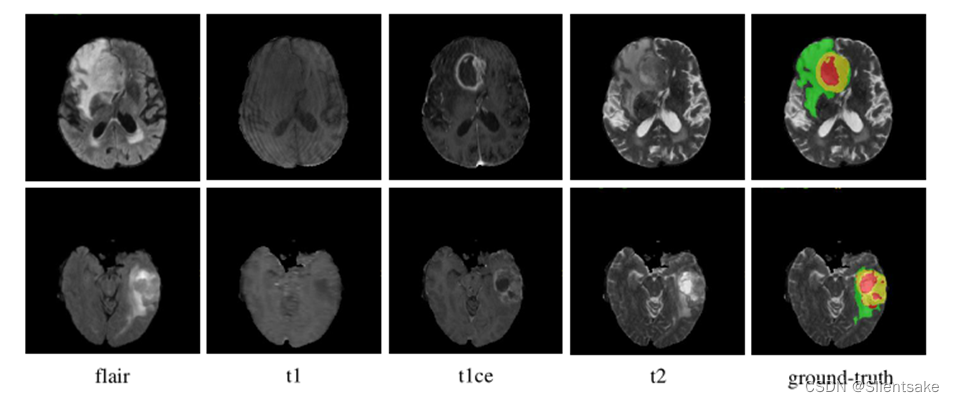
??图1所示。示例扫描所有的模式,并附上相应的基本事实。NCR/NET、ED和ET区域分别用红色、绿色和黄色表示。
模型方法:
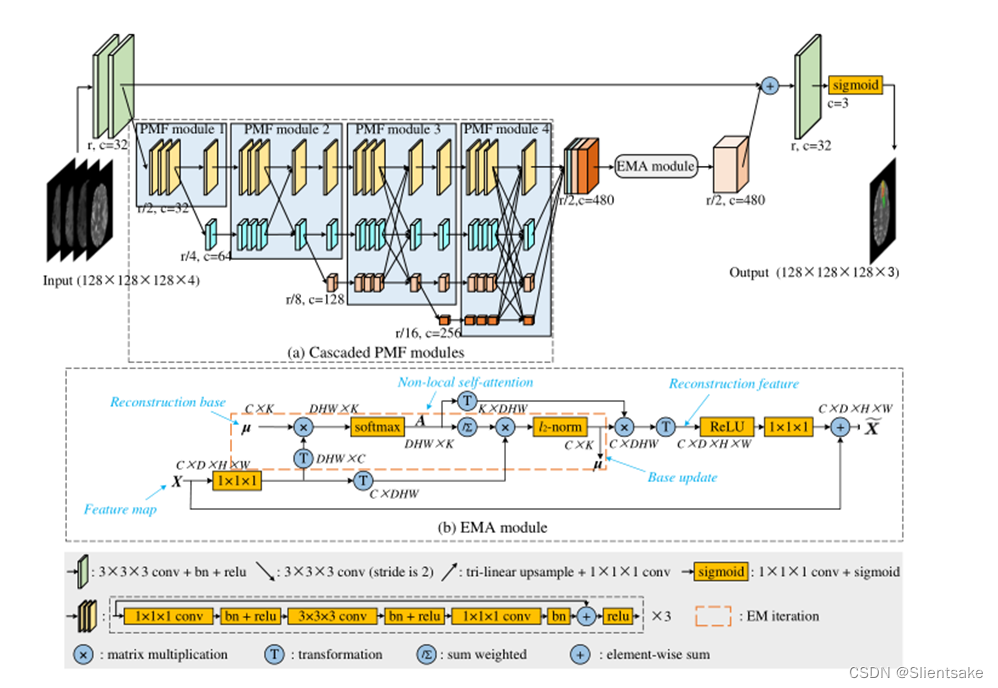
??图2所示。原始HNF-Net的体系结构。在每一项研究中,四个多模态脑磁共振序列首先被连接起来形成一个四通道输入,然后在五个尺度上进行处理。R为原始分辨率,c为feature map的通道数。所有下采样操作都是通过2步卷积实现的,所有上采样操作都是通过关节1 × 1 × 1卷积和三线性插值实现的。需要注意的是,由于图中不方便显示4D特征图(C × D × H × W),所以作者将所有的特征图都显示出来,没有深度信息,每个特征图的厚度可以显示其通道号。
??HNF-Net具有5个尺度的编解码器结构,如图2所示。在原始尺度r上,有四个卷积块,两个用于编码,另外两个用于解码。在其他四个尺度上,四个PMF模块被联合用作高分辨率和多尺度聚合特征提取器。在最后一个PMF模块的末尾,首先将四个尺度的输出特征映射恢复到1/2r尺度,然后将其拼接成混合特征。其次,利用EMA模块有效地捕获远程依赖上下文信息,并减少获得的混合特征的冗余。最后,EMA模块的输出通过1×1×1卷积和上采样恢复到原始比例r和32通道,然后添加到编码器生成的全分辨率特征图中,用于体素标签的密集预测。所有下样操作都通过2步卷积实现,所有上样操作都通过关节1 × 1 × 1卷积和三线性插值实现。
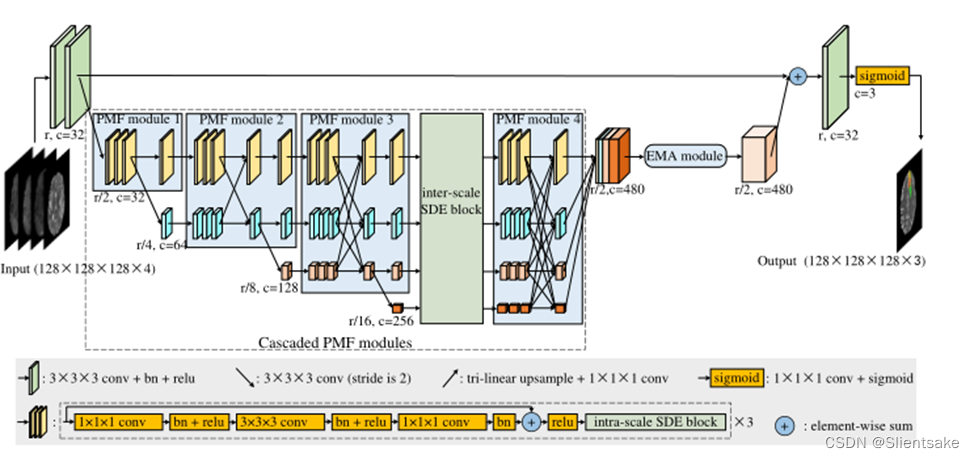
??图3所示。HNF-Netv2的架构。与图2相似,r为原始分辨率,c为feature map的通道数。同时,作者在没有深度信息的情况下显示所有地物图,每个地物图的厚度显示其通道号。与HNF-Net相比,作者在级联PMF模块中进一步增加了尺度间和尺度内的SDE块。
??已经证明,学习强分辨率表示对于小目标分割任务至关重要,例如医学图像中的肿瘤和病变分割。在此基础上,采用多尺度卷积分支和全连接融合设置构建PMF模块,前者可以充分利用多分辨率特征,但保持高分辨率特征表示,后者可以聚合丰富的多尺度上下文信息。此外,作者在作者的HNF-Net中串联了多个PMF模块,其中分支的数量随深度递增,如图2(a)所示。因此,从最高分辨率阶段来看,通过多尺度低分辨率表示的反复融合,提高了其高分辨率特征表示。
??非局部自注意机制虽然具有令人信服的从所有空间位置聚合上下文信息和捕获长期依赖关系的能力,但由于其潜在的高计算复杂度,难以应用于3D医学图像分割任务。为了解决这个问题,作者在作者的HNF-Net中引入了EMA模块,旨在将轻量级的非局部注意力机制纳入作者的模型中。EMA模块的主要概念(如图2 (b)所示)是在一组特征重建基础上操作非局部注意,而不是直接在高分辨率特征图上实现。由于重构基的元素比原始特征图少得多,可以显著降低非局部注意的计算成本。EMA模块的详细信息也可以在中找到。
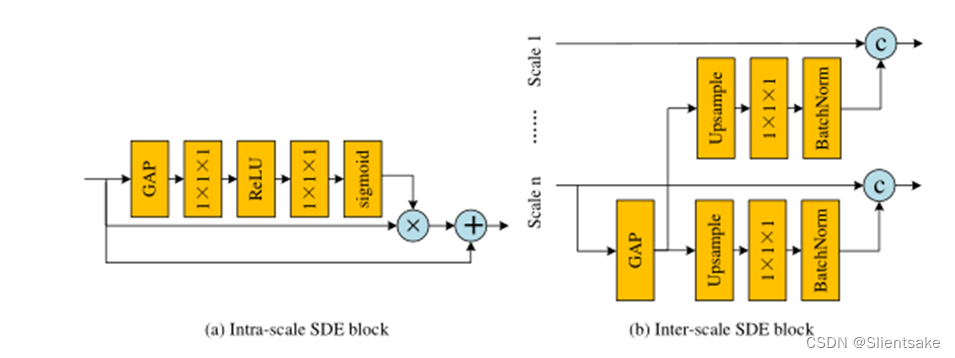
??图4所示。(a)尺度内语义辨别增强块,(b)尺度间语义辨别增强块,?多尺度注意模块的结构。GAP, c〇,+〇,×〇分别表示全局平均池化,串联,元素式求和,矩阵点乘法。
??在提议的HNF-Netv2中,作者在级联的PMF模块中部署了尺度间和尺度内的SDE块。如图3所示,作者在PMF模块的每个卷积分支内部构造尺度内的SDE块。同时,为了控制计算复杂度,作者只在第3个PMF模块和第4个PMF模块之间插入一个尺度间的SDE块。现在作者将深入研究这些关键组件的细节。
??尺度间SDE模块采用PMF模块全连接融合块部署,结构如图4 (a)所示。作者首先对规模n(当前PMF模块中规模最小的分支)的特征应用一个全局平均池(GAP)层,获得具有高度语义信息的全局上下文。然后,作者将得到的特征分别上采样到PMF模块各分支的分辨率。考虑到上采样特征的空间信息较差,作者使用1×1×1卷积层将信道数减少到1,然后将其与高分辨率特征连接。在此设置下,作者可以在减少对原始空间信息破坏的同时,在高分辨率特征中增加全局语义识别。不同于inter-scale端模块,intra-scale端块每个回旋的分支及模块的内部构造与图4中所示的结构(b)。作者还利用一个缺口层生成全局上下文的特性和应用两个1×1×1卷积层调整频道号码。与基于cnn的分类网络的预测层相似,获得的全局特征从各个空间位置收集信息,具有较强的语义信息。因此,作者可以利用这些全局特征对输入的高分辨率特征进行重新加权,从而进一步增强全局语义识别能力。在之前的工作[10,9]之后,作者最终将多尺度增强特征串联起来作为EMA模块的输入。
实验与结果:
数据与评价指标:
??BraTS挑战赛庆祝了10周年,由北美放射学会(RSNA)、美国神经放射学会(ASNR)和医学图像计算和计算机辅助干预学会(MICCAI)共同组织。BraTS21数据集包含2000个多模态脑MR研究(8000个mpMRI扫描),包括1521个训练、219个验证和260个测试案例。与前几年挑战的数据设置相同,每项研究有4张MR图像,分别为T1加权(T1)、对比度后T1加权(T1ce)、T2加权(T2)和液体衰减反演恢复(Flair)序列,如图1所示。所有MR图像的尺寸均为240 × 240 × 155,体素间距均为1 × 1 × 1mm3。对于每一项研究,专家都将肿瘤增强(ET)、瘤周水肿(ED)、坏死和非增强肿瘤核心(NCR/NET)按体素逐个进行标注。培训研究的注释是公开的,而验证研究和测试研究的注释将被保留,分别用于在线评估和最终细分竞争。
??为了评估在HNF-Netv2中提出的SDE块的有效性,作者首先使用五倍交叉验证对训练集进行消融研究。作者选择3D U-Net作为基线模型。此外,作者先后测试了原始HNF-Net的性能,使用了尺度内的SDE块,并进一步测试了尺度内的SDE块(提议的HNF-Netv2)。如表1所示,通过Dice评分和对分割性能进行评价
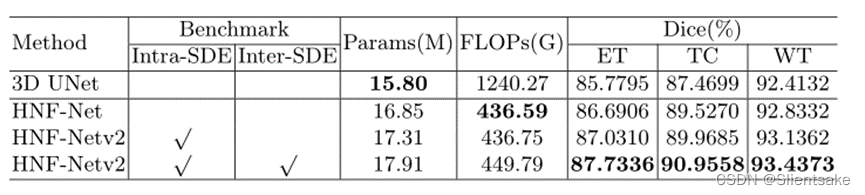
??表1:BraTS 2021训练装置的消融研究。Inter-SDE:尺度间SDE块,Intra-SDE:尺度内SDE块,DSC:骰子相似系数,HD95: Hausdorff距离(95%),WT:整个肿瘤,TC:肿瘤核心,ET:增强肿瘤核心。
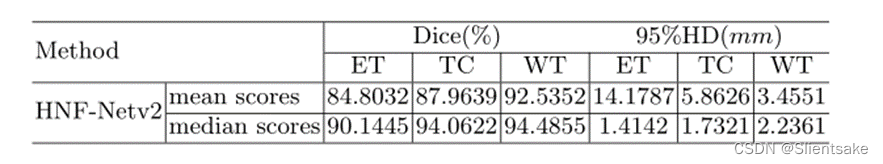
??表2:作者的方法在BraTS 2021验证集上的分割性能。DSC:骰子相似系数,HD95: Hausdorff距离(95%),WT:全瘤,TC:肿瘤核心,ET:增强肿瘤核心。提供了所有文件的分割结果的平均值和中值。
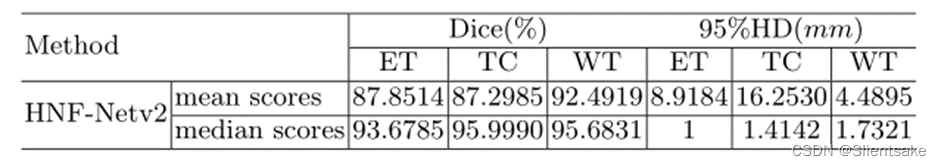
??表3:作者的方法在BraTS 2021测试集上的分割性能。DSC:骰子相似系数,HD95: Hausdorff距离(95%),WT:全瘤,TC:肿瘤核心,ET:增强肿瘤核心。数据由挑战组织者提供,列出了所有文件的分割结果的平均值和中值。
总结:
??在本文中,作者提出了一种基于多模态MR成像的脑肿瘤分割HNF-Netv2,它通过添加尺度间和尺度内的SDE块来扩展HNF-Net,增强特征的语义识别能力。作者在BraTS 2021挑战数据集上评估了作者的方法,令人信服的结果表明,新提出的关键块和HNF-Netv2的有效性。