生物医药领域当中,涉及到的细胞分割问题一直是视觉研究的重点和难点之一,虽然以深度学习为代表的分割模型取得了一定的效果,但分割网络会涉及到人工标注和GPU等高成本的资源占用,所以探索便捷、高效的分割算法进行一直是解决细胞分割的核心。今天我就基于自己的尝试,分享两种方法。
- 生物细胞图像
src = cv2.imread(img_Path)
src = cv2.resize(src, dsize=None, fy=0.5, fx=0.5)
cv2pil(src)
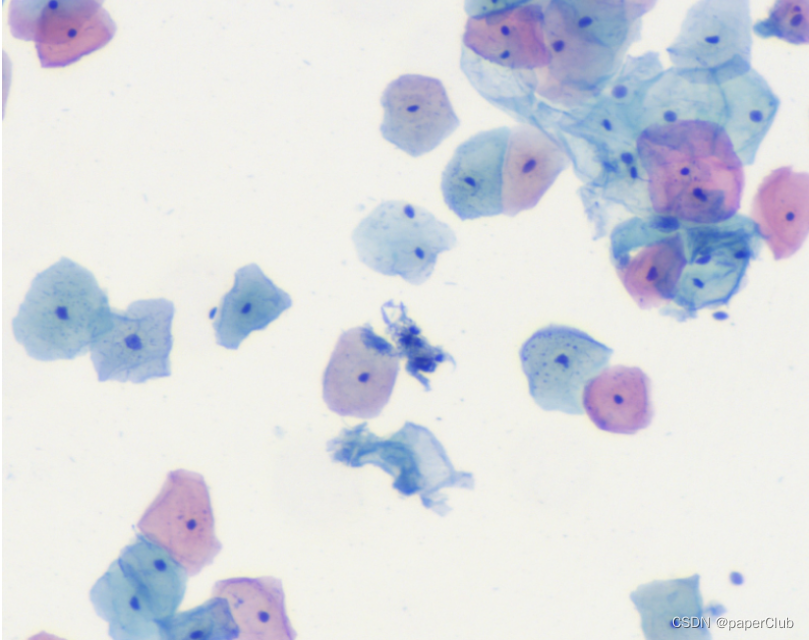
如图,动物细胞主要由细胞质(浅蓝色或浅红色)和细胞核(深蓝色)构成。我们看到的生物细胞颜色会有所不同,有浅蓝色的和浅红色的,颜色不同是由于细胞质和染色液结合后发生了不同的显色反应(主要是由于细胞内环境及PH差异引起),这也反应了细胞所处的阶段或生理转态不同,中间的深蓝色小点就是细胞核。
2.细胞核分割1:
针对这样的细胞图像,我们通过单一方法完成细胞分割,即便分割成功也不太可能有很强的的鲁棒性,由于时间关系,我本次简单分享2种细胞核分割方法。
细胞区域分割:
我们先拿到hsv图像, 完成细胞区域分离:
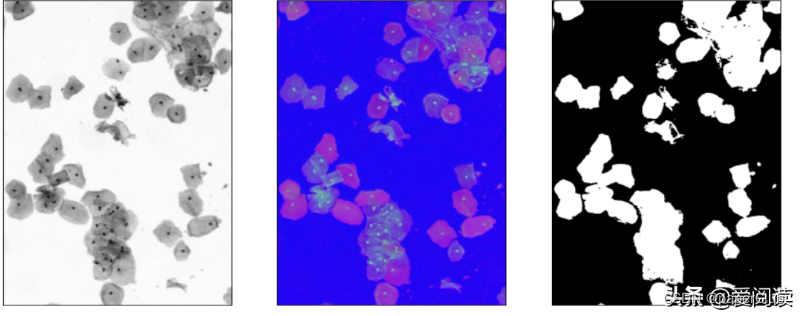
img_gray = cv2.cvtColor(src, cv2.COLOR_BGR2GRAY)
img_hsv = cv2.cvtColor(src, cv2.COLOR_BGR2HSV)
img_gray = cv2.cvtColor(src, cv2.COLOR_BGR2GRAY)
img_hsv = cv2.cvtColor(src, cv2.COLOR_BGR2HSV)
cell_threshold, bw_cell = cv2.threshold(img_gray, 0, 255, cv2.THRESH_OTSU | cv2.THRESH_BINARY_INV)
img_mask_mean = max(cv2.mean(img_gray, bw_cell)[0], 180.0)
fig = plt.figure(figsize=(15,8))
plt.subplot(131), plt.imshow(img_gray, 'gray'), plt.xticks([]), plt.yticks([])
plt.subplot(132), plt.imshow(img_hsv, 'gray'), plt.xticks([]), plt.yticks([])
plt.subplot(133), plt.imshow(bw_cell, 'gray'), plt.xticks([]), plt.yticks([])
plt.show()
?
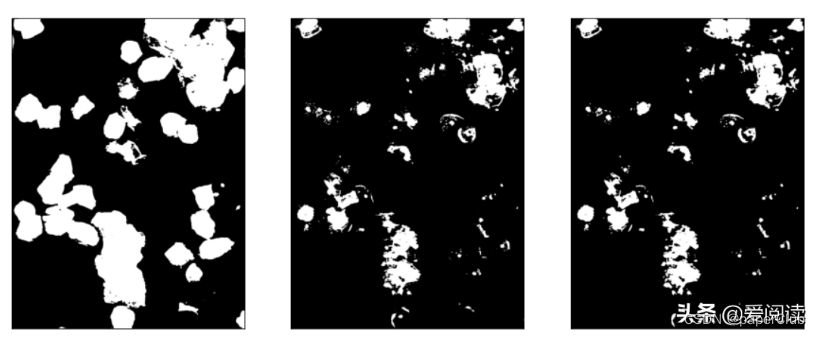
### 获取细胞区域
my_threshold = img_mask_mean - bg_gray_r
_, bw_cell1 = cv2.threshold(img_gray, my_threshold, 1, cv2.THRESH_BINARY_INV)
element = cv2.getStructuringElement(cv2.MORPH_ELLIPSE, (3, 3))
bw_cell2 = cv2.morphologyEx(bw_cell1, cv2.MORPH_OPEN, element)
fig = plt.figure(figsize=(15,8))
plt.subplot(131), plt.imshow(bw_cell, 'gray'), plt.xticks([]), plt.yticks([])
plt.subplot(132), plt.imshow(bw_cell1, 'gray'), plt.xticks([]), plt.yticks([])
plt.subplot(133), plt.imshow(bw_cell2, 'gray'), plt.xticks([]), plt.yticks([])
plt.show()
 ?
?
?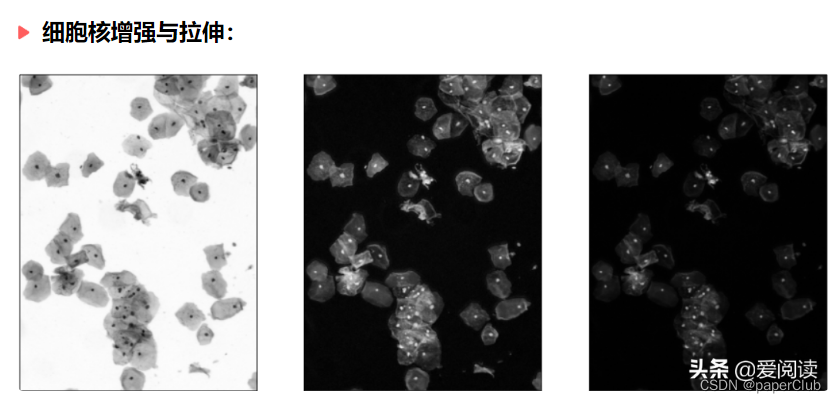

?
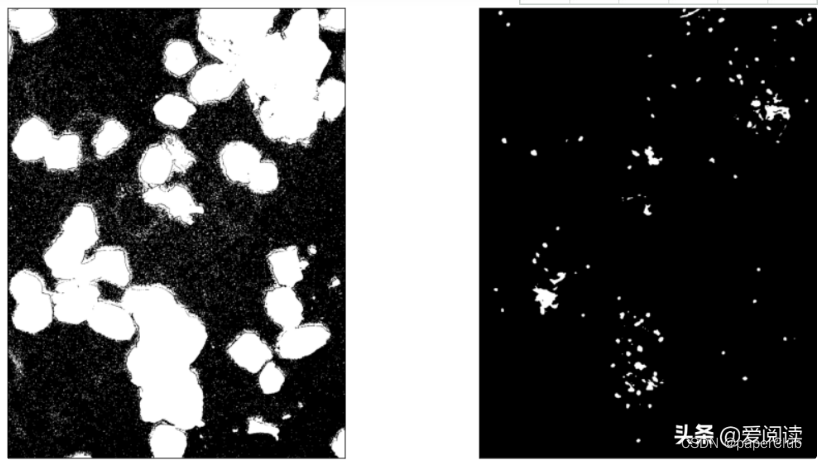
细胞区域初步分割结果:
_,bw_nucleus_s = cv2.threshold(img_s, 10, 255, cv2.THRESH_BINARY)
bw_nucleus = cv2.bitwise_and(bw_nucleus, bw_nucleus_s))
?
fig = plt.figure(figsize=(15,8))
plt.subplot(121),plt.imshow(bw_nucleus_s, 'gray') , plt.xticks([]),plt.yticks([])
plt.subplot(122),plt.imshow(bw_nucleus, 'gray'), plt.xticks([]),plt.yticks([])
plt.show()
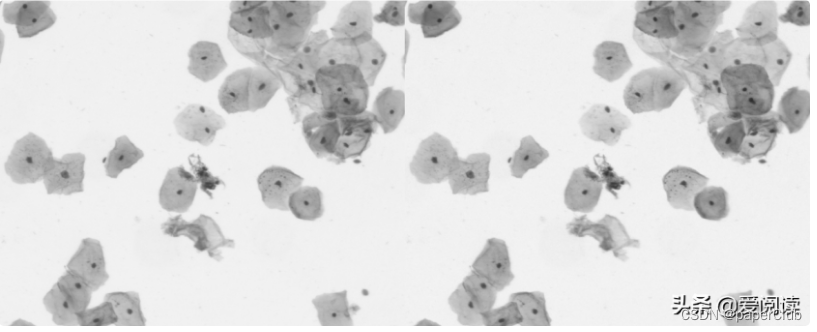
最终细胞核分割定位效果:左边是原图,右边是细胞核行为结果:
例子1:
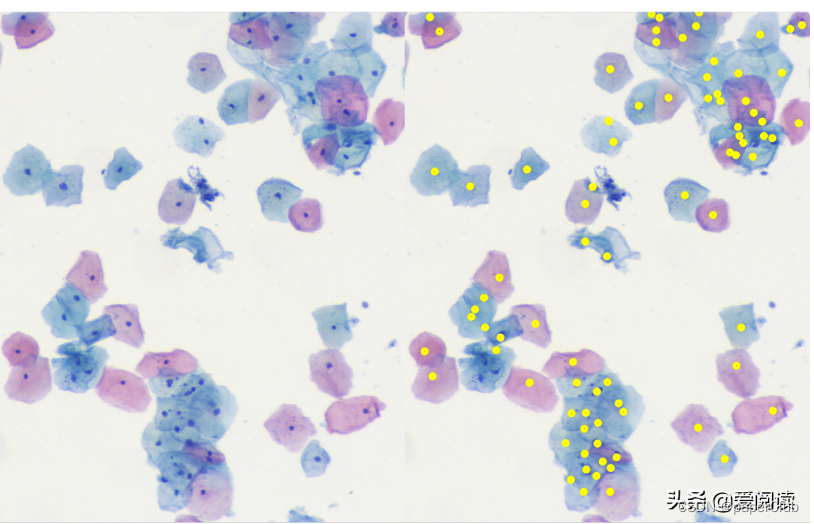
?
例子2:
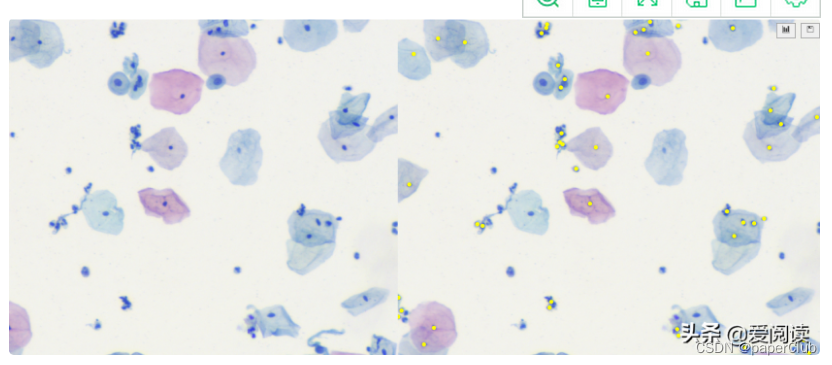
?
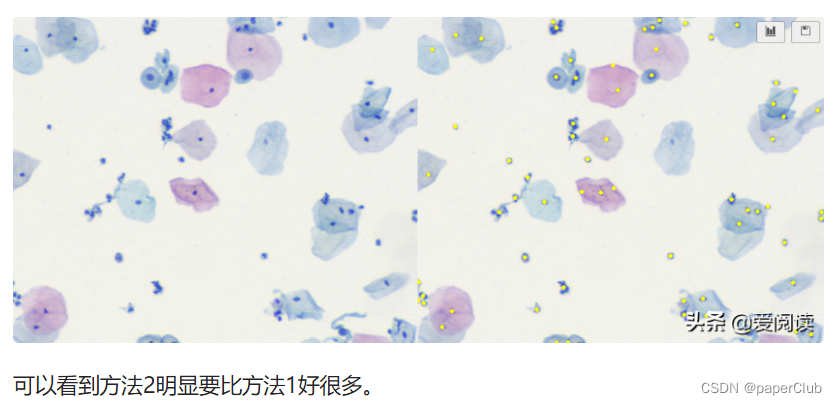
可以看到效果很得不错的。当然这里面还有很大的优化空间。
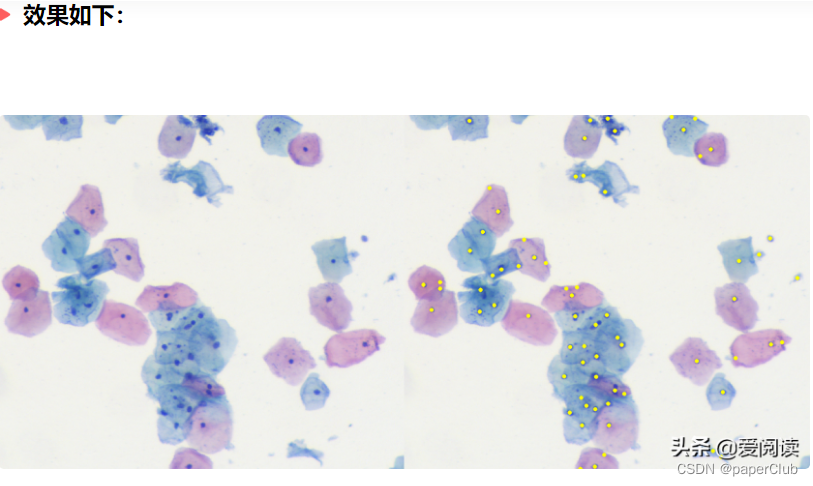
2.细胞核分割方法2:
img_gray = cv2.cvtColor(src, cv2.COLOR_BGR2GRAY)
b,g,r = cv2.split(src)
cv2pil(np.hstack((img_gray, g)))
左边为原始灰度图像, 右侧为g通道图像,由于图像本身的特征,我们选择g通道进行处理。
 ?
?
?
?
?
?
?
?