点击关注,桓峰基因
桓峰基因
生物信息分析,SCI文章撰写及生物信息基础知识学习:R语言学习,perl基础编程,linux系统命令,Python遇见更好的你
135篇原创内容
公众号
今天来介绍一个使用非常方便的在线分析工具——TIMER (Tumor Immune Estimation Resource),是由哈佛大学免疫信息学教授刘小乐领导建立的一个网站工具专门用于肿瘤浸润免疫细胞分析,网址:
https://cistrome.shinyapps.io/timer/
桓峰基因的教程不但教您怎么使用,还会定期分析一些相关的文章,学会教程只是基础,但是如果把分析结果整合到文章里面才是目的,觉得我们这些教程还不错,并且您按照我们的教程分析出来不错的结果发了文章记得告知我们,并在文章中感谢一下我们哦!
公司英文名称:Kyoho Gene Technology (Beijing) Co.,Ltd.
桓峰基因公众号推出转录组分析和临床预测模型教程,有需要生信的老师可以联系我们!首选看下转录分析教程整理如下:
RNA 1. 基因表达那些事–基于 GEO
RNA 2. SCI文章中基于GEO的差异表达基因之 limma
RNA 3. SCI 文章中基于T CGA 差异表达基因之 DESeq2
RNA 4. SCI 文章中基于TCGA 差异表达之 edgeR
RNA 5. SCI 文章中差异基因表达之 MA 图
RNA 6. 差异基因表达之-- 火山图 (volcano)
RNA 7. SCI 文章中的基因表达——主成分分析 (PCA)
RNA 8. SCI文章中差异基因表达–热图 (heatmap)
RNA 9. SCI 文章中基因表达之 GO 注释
RNA 10. SCI 文章中基因表达富集之–KEGG
RNA 11. SCI 文章中基因表达富集之 GSEA
RNA 12. SCI 文章中肿瘤免疫浸润计算方法之 CIBERSORT
RNA 13. SCI 文章中差异表达基因之 WGCNA
RNA 14. SCI 文章中差异表达基因之 蛋白互作网络 (PPI)
RNA 15. SCI 文章中的融合基因之 FusionGDB2
RNA 16. SCI 文章中的融合基因之可视化
RNA 17. SCI 文章中的筛选 Hub 基因 (Hub genes)
RNA 18. SCI 文章中基因集变异分析 GSVA
RNA 19. SCI 文章中无监督聚类法 (ConsensusClusterPlus)
RNA 20. SCI 文章中单样本免疫浸润分析 (ssGSEA)
RNA 21. SCI 文章中单基因富集分析
RNA 22. SCI 文章中基于表达估计恶性肿瘤组织的基质细胞和免疫细胞(ESTIMATE
RNA 23. SCI文章中表达基因模型的风险因子关联图(ggrisk)
RNA 24. SCI文章中基于TCGA的免疫浸润细胞分析 (TIMER)
临床预测模型整理如下:
Topic 1. _临床_标志物生信分析常规思路
Topic 2. 生存分析之 Kaplan-Meier
Topic 3. SCI文章第一张表格–基线表格
Topic 4. _临床_预测模型构建 Logistic 回归
Topic 5. 样本量确定及分割
Topic 6 计数变量泊松回归
Topic 7. _临床_预测模型–Cox回归
Topic 8. _临床_预测模型-Lasso回归
Topic 9. SCI 文章第二张表—单因素回归分析表
Topic 10. 单因素 Logistic 回归分析—单因素分析表格
Topic 11. SCI中多元变量筛选—单/多因素表 Topic 12 _临床_预测模型—列线表 (Nomogram)
Topic 13. _临床_预测模型—一致性指数 (C-index)
Topic 14. _临床_预测模型之校准曲线 (Calibration curve)
Topic 15. _临床_预测模型之决策曲线 (DCA)
Topic 16. _临床_预测模型之接收者操作特征曲线 (ROC)
Topic 17. 临床预测模型之缺失值识别及可视化
Topic 18. 临床预测模型之缺失值插补方法
TIMER同样也是利用RNA-Seq表达谱数据检测肿瘤组织中免疫细胞的浸润情况。TIMER则提供6种免疫细胞的浸润情况:
-
B cells
-
CD4+ T cells
-
CD8+ T cells
-
Neutrphils
-
Macrophages
-
Dendritic cells
TIMER网站分7个模块,其中前面6个模块为展示TCGA数据的展示和一些分析,最后一个模块则提供免疫细胞浸润比例的量化分析。网站首页有个视频,不过需要科学上网才能看,有兴趣的同学可以看看,不过关注桓峰基因视频号,B站,或者抖音都可以观看我自己录制的视频,下面我们逐个模块介绍。
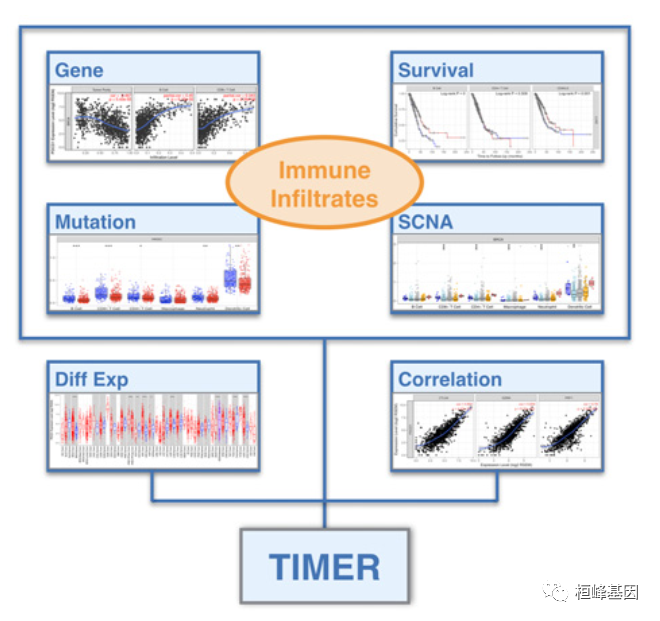
进入主页我们看到主要介绍可分析的内容:

1. 基因****模块
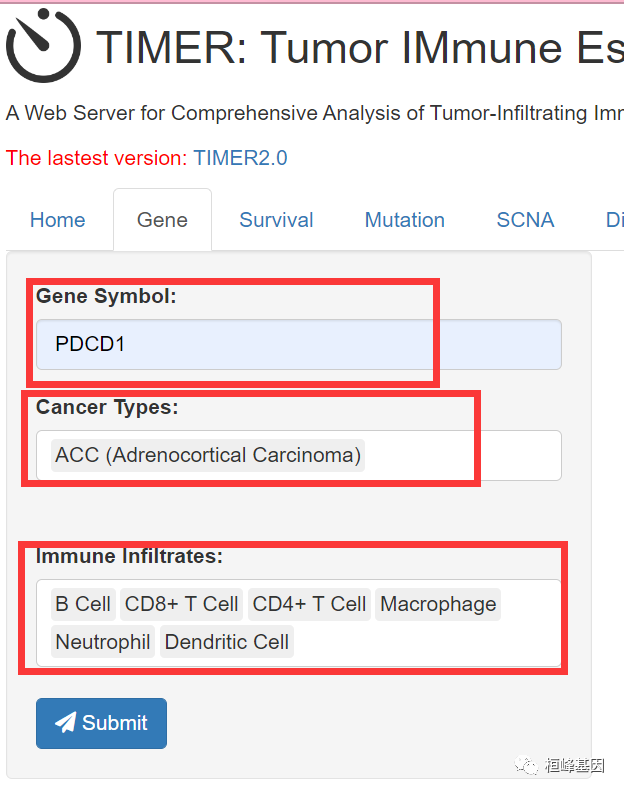
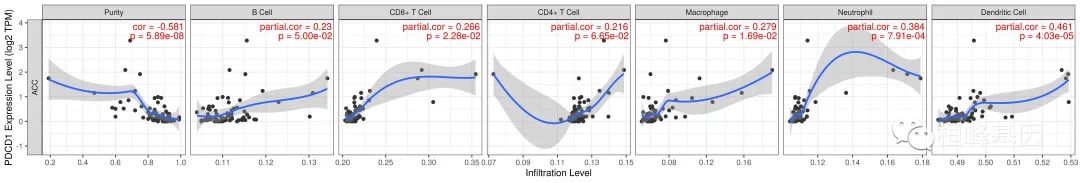
基因模块允许用户选择任何感兴趣的基因,并可视化其表达与不同癌症类型的免疫浸润水平的相关性。需要一个官方的基因符号和至少一种癌症类型作为输入。输入提交成功后,将生成散点图并显示,显示纯净校正后的偏Spearman s rho值和统计显著性。基因表达水平对肿瘤纯度的影响始终显示在最左边的面板上。因为微环境中高表达的基因与肿瘤纯度呈负相关,而肿瘤细胞中高表达的基因与肿瘤纯度呈负相关。由于GBM/OV的微阵列数据比RNA-seq数据包含更多的样本,我们使用GBM/OV的微阵列表达值来计算该基因是否可用。
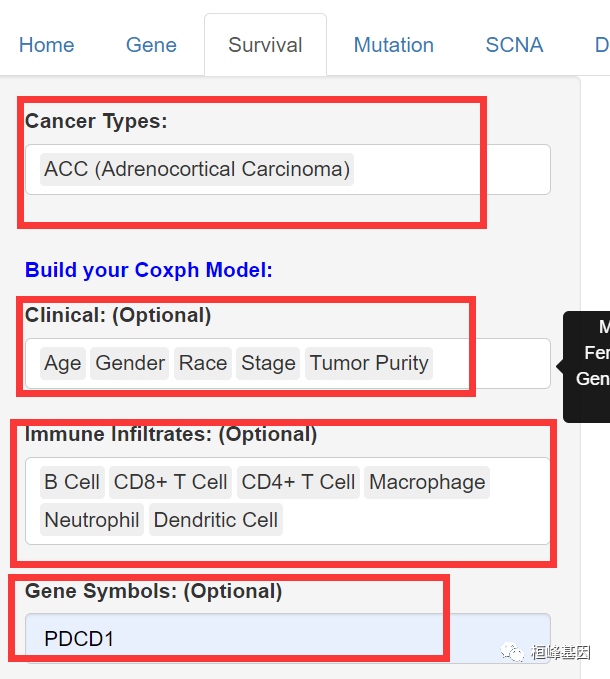
2. 生存模块
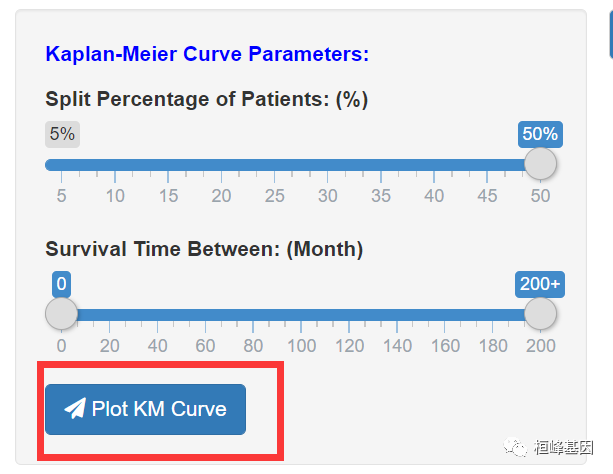
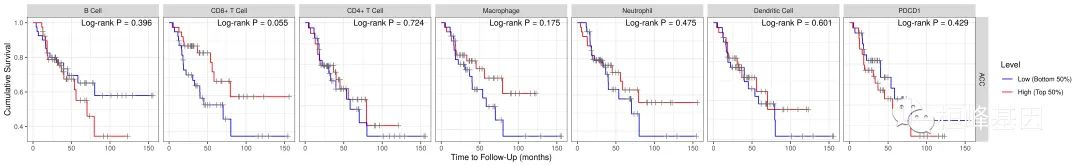
生存模块允许用户探索一个或多个肿瘤免疫亚群的临床相关性,并灵活地修正多变量Cox比例风险模型中的多协变量。协变量包括临床因素(年龄、性别、种族、肿瘤分期)和基因表达。一旦定义了所有输入,TIMER自动输出Cox回归结果,包括风险比和统计显著性。TIMER还绘制了免疫浸润和基因的Kaplan-Meier图,以可视化生存差异。级别由用户定义的滑块分为低级别和高级别。两组生存曲线比较log-rank检验的p值分别显示在每张图中。对于Cox模型的输出,Surv(CancerType)~变量是用户自定义Cox回归模型的公式。该模型由R包’survival’中的函数coxph()拟合;系数coef读作回归系数。HR给出了风险比,其上下95%的机密区间显示在95% ci_l &ci_u 95%。Cox比例风险模型
这里我们看到列出的R代码,也就是说其实我们自己做也非常方便,原理是一样的,做COX风险比率模型:
Model: Surv(ACC) ~ Age + Gender + Race + Stage + Purity + B_cell + CD8_Tcell + CD4_Tcell + Macrophage + Neutrophil + Dendritic + PDCD1
63 patients with 23 dying
coef HR 95%CI_l 95%CI_u p.value sig
Age -0.039 9.610000e-01 9.180000e-01 1.006000e+00 0.091 ·
gendermale 0.693 2.001000e+00 6.490000e-01 6.167000e+00 0.227
raceBlack 2.275 9.726000e+00 0.000000e+00 Inf 1.000 <NA>
raceWhite 18.569 1.159484e+08 0.000000e+00 Inf 1.000 <NA>
stage2 0.310 1.363000e+00 1.140000e-01 1.636100e+01 0.807
stage3 0.474 1.607000e+00 1.480000e-01 1.741600e+01 0.696
stage4 1.959 7.092000e+00 5.280000e-01 9.522800e+01 0.139
Purity 20.351 6.891871e+08 2.333229e+03 2.035714e+14 0.002 **
B_cell -197.721 0.000000e+00 0.000000e+00 1.248131e+06 0.067 ·
CD8_Tcell -220.300 0.000000e+00 0.000000e+00 0.000000e+00 0.001 **
CD4_Tcell 68.868 8.106935e+29 0.000000e+00 1.718436e+125 0.539
Macrophage -96.803 0.000000e+00 0.000000e+00 2.408022e+06 0.089 ·
Neutrophil 522.912 1.252504e+227 1.707646e+95 Inf 0.001 **
Dendritic 83.777 2.420769e+36 0.000000e+00 1.468696e+109 0.327
PDCD1 2.375 1.075100e+01 1.139000e+00 1.015110e+02 0.038 *
Rsquare= 0.528 (max possible= 9.34e-01 )
Likelihood ratio test p= 3.32e-05
Wald test p= 7.45e-02
Score (logrank) test p= 5.55e-04
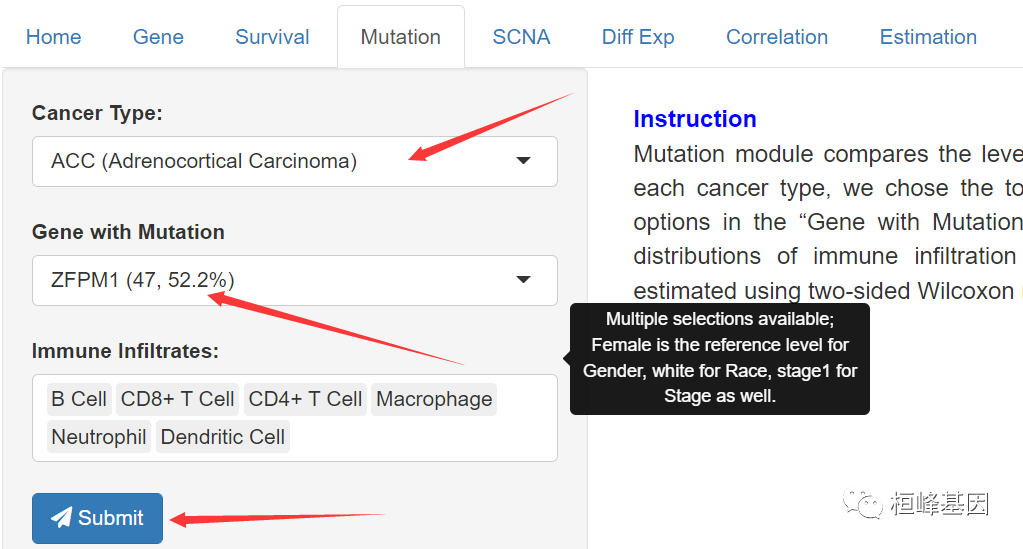
3. 突变模块
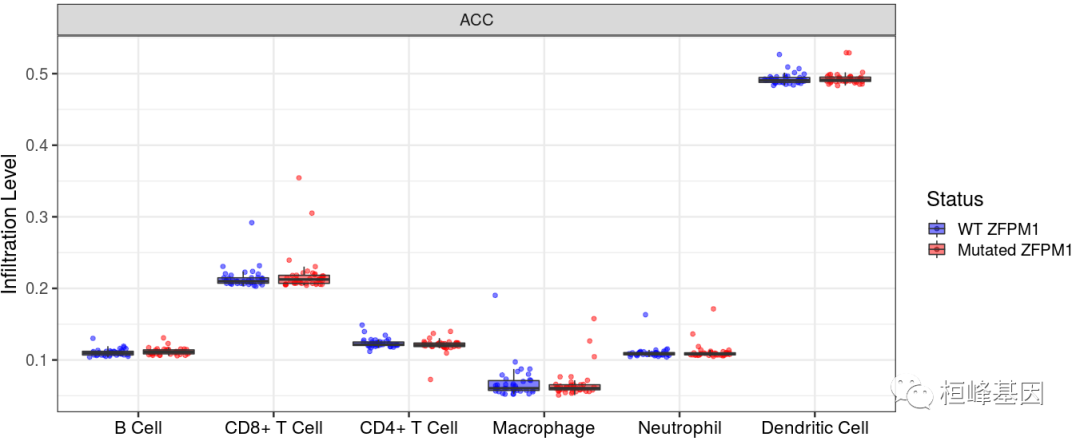
突变模块比较免疫渗透的水平与没有一个给定的突变的存在。在每一种癌症类型中,我们选择了前50个基因或10%最常见的非同义突变作为基因突变领域的选项。对每个免疫亚群生成箱形图,比较不同基因突变状态下的免疫浸润水平分布,使用双侧Wilcoxon秩和检验估计统计显著性。
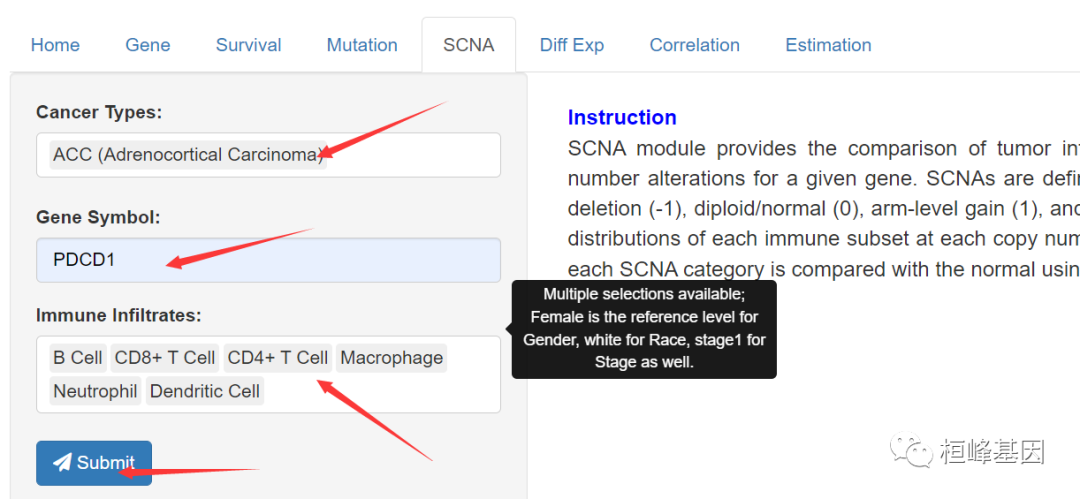
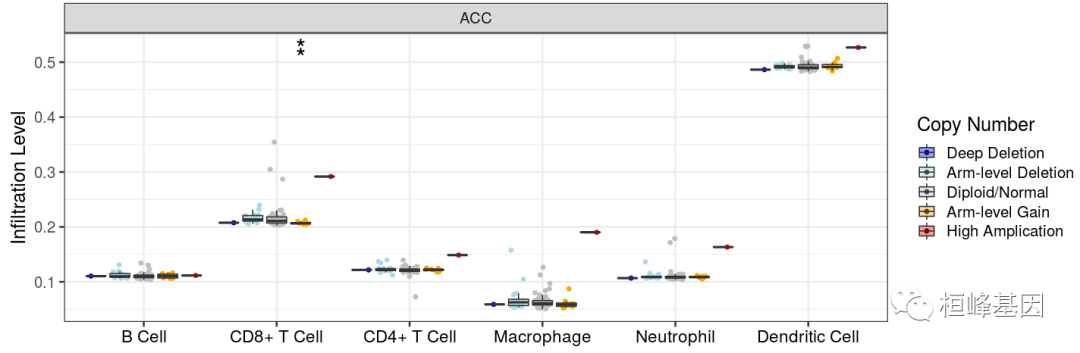
4. SCNA模块
SCNA模块提供了一个特定基因的不同体细胞拷贝数改变的肿瘤之间的肿瘤浸润水平的比较。SCNAs由GISTIC 2.0定义,包括深度删除(-2)、臂级删除(-1)、二倍体/正常(0)、臂级增益(1)和高扩增(2)。框图显示了选定癌症类型中每个拷贝数状态下每个免疫子集的分布。每个SCNA类别的渗透水平与正常比较使用双侧Wilcoxon秩和检验。
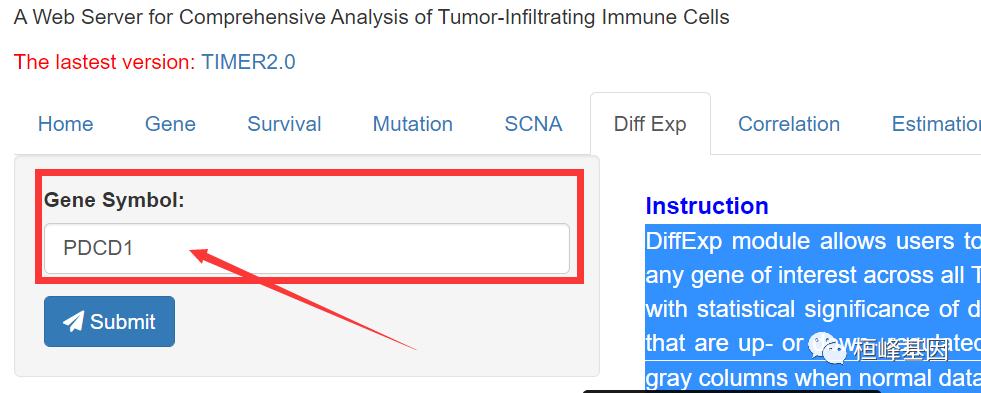
5. 差异表达模块
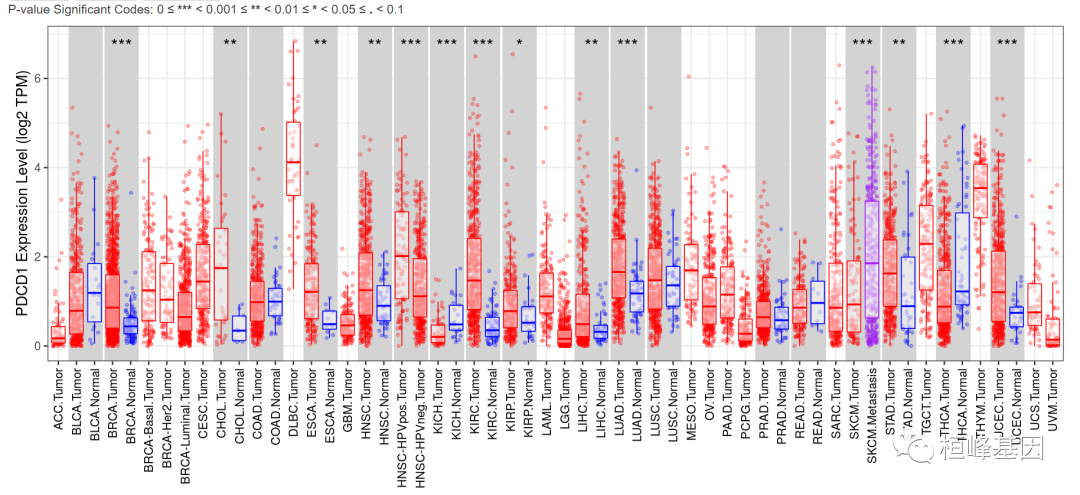
DiffExp模块允许用户研究所有TCGA肿瘤中任何感兴趣的基因在肿瘤和相邻正常组织之间的差异表达。基因表达水平分布采用箱线图显示,差异表达采用Wilcoxon检验评估差异有统计学意义。与正常组织相比,用户可以识别每种癌症类型的肿瘤中上调或下调的基因,当有正常数据时显示为灰色列。
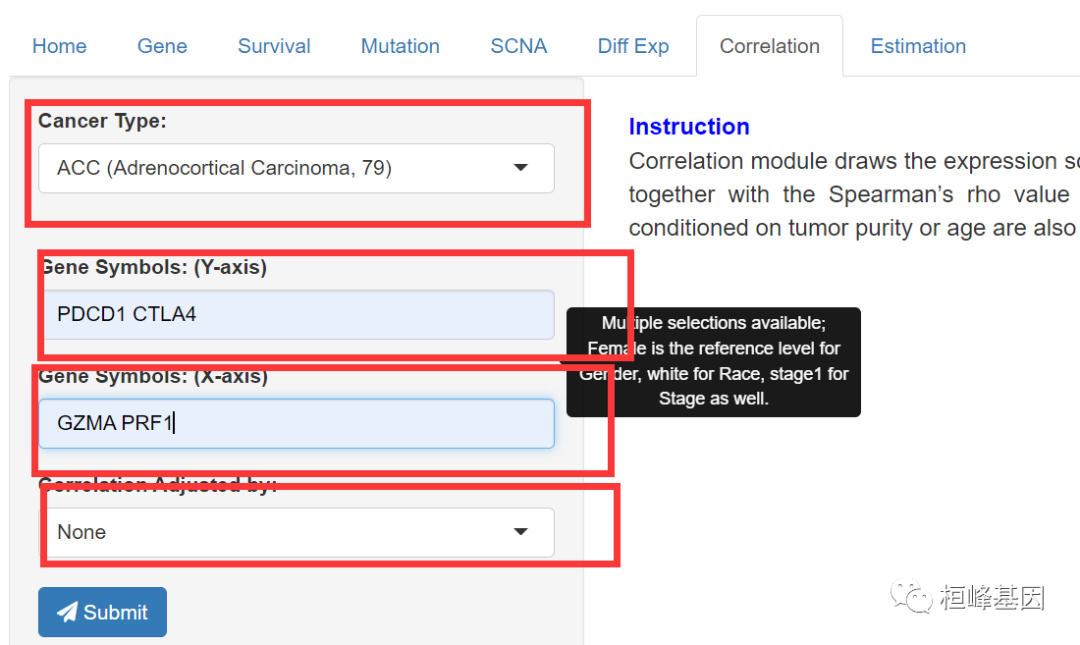
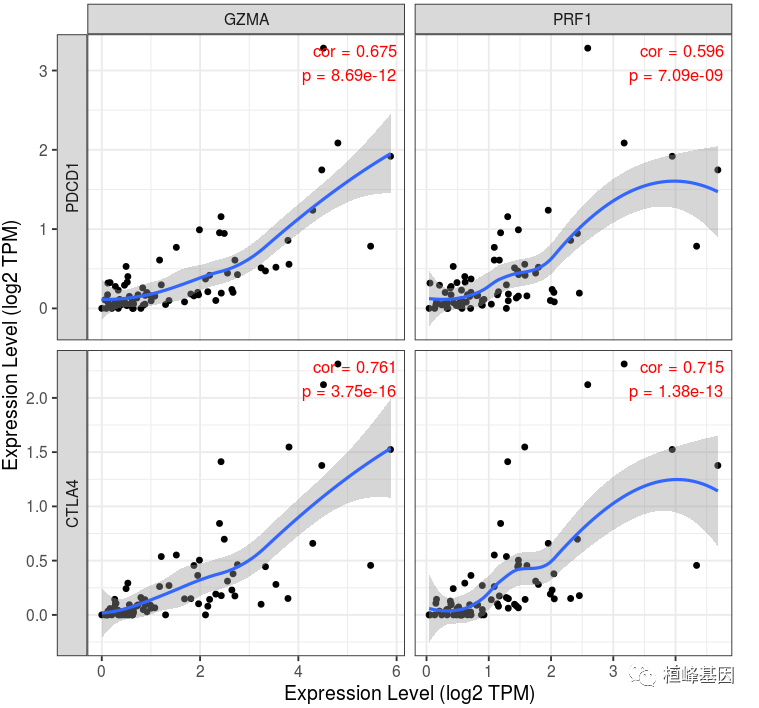
6. 基因相关模块
基因相关模块在给定的癌症类型中绘制一对用户定义的基因之间的表达散点图,以及斯皮尔曼的rho值和估计的统计显著性。还提供了基于肿瘤纯度或年龄的部分相关性选项。
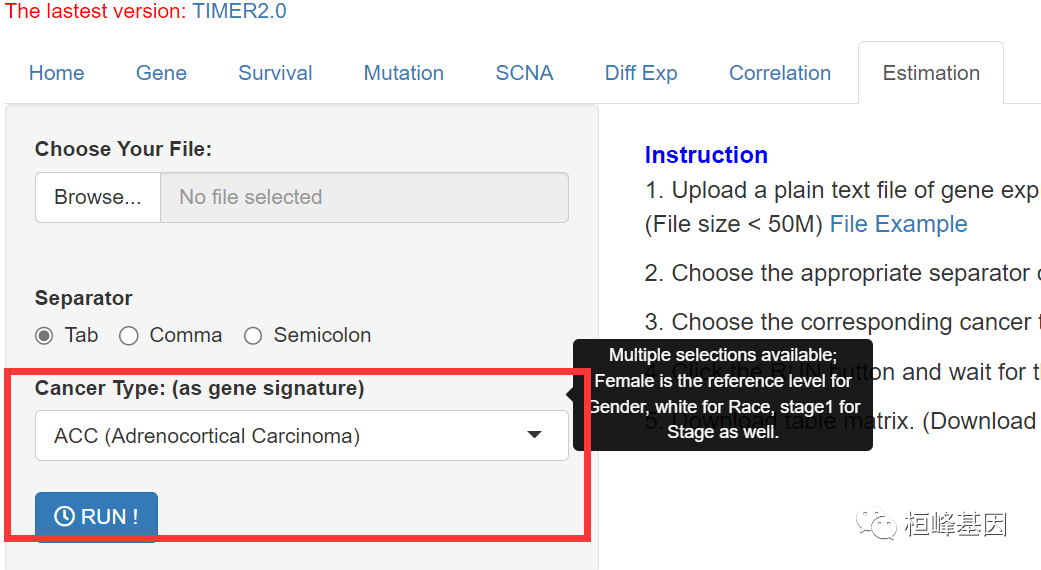
7. 免疫浸润估计
-
上传以HUGO基因符号为行名、样本id为姓氏的基因表达纯文本文件;(文件大小;
-
选择合适的文件分隔符(例如“Comma”文件);
-
选择相应的癌症类型(只有TCGA型癌症可用);
-
点击RUN按钮,等待下面的记分表出现;
-
下载表矩阵。(此处下载TCGA估计)
barcode B_cell CD4_Tcell CD8_Tcell Neutrophil Macrophage Dendritic
TCGA-KL-8323-01 0.0946283089154644 0.108337752166578 0.202262725529644 0.109738179426816 0.0369556203224999 0.449038365170569
TCGA-KL-8324-11 0.11994965968083 0.175790652792388 0.189856839177697 0.125519325190091 0.0490088691119843 0.563384175141639
TCGA-KL-8324-01 0.0761443359577819 0.112892939001227 0.19798990581833 0.107687535022284 0.0335292549991947 0.446329161702569
TCGA-KL-8325-01 0.102200865478893 0.082541054558658 0.238241965207092 0.102257676610089 0.0878383561787278 0.465910318209685
TCGA-KL-8326-11 0.199018372814469 0.185392956121084 0.184026681862867 0.124147715234841 0.0570364968493927 0.48253452966049
这么样是不是非常简单,这个在线工具比其他的绘图效果要好些,颜色搭配的也合理,提供PDF版本下载,可以整合到自己的文章当中,非常好用!
跟着桓峰基因教程学习,铸造成功的您,记得关注我们哦,如果觉得自己搞生信有难度,快来联系桓峰基因吧!!!
References:
-
Taiwen Li, Jingyu Fan, Binbin Wang, Nicole Traugh, Qianming Chen, Jun S. Liu, Bo Li, X. Shirley Liu. TIMER: A web server for comprehensive analysis of tumor-infiltrating immune cells. Cancer Research. 2017;77(21):e108-e110.
-
Bo Li, Eric Severson, Jean-Christophe Pignon, Haoquan Zhao, Taiwen Li, Jesse Novak, Peng Jiang, Hui Shen, Jon C. Aster, Scott Rodig, Sabina Signoretti, Jun S. Liu, X. Shirley Liu. Comprehensive analyses of tumor immunity: implications for cancer immunotherapy. Genome Biology. 2016;17(1):174.