#环境设置
import os
import re
import torch
import warnings
os.environ["KMP_DUPLICATE_LIB_OK"] = "True"
torch.backends.cudnn.benchmark=True
warnings.filterwarnings("ignore", category=DeprecationWarning)
warnings.filterwarnings("ignore", category=UserWarning)
warnings.filterwarnings("ignore", category=FutureWarning)
import cv2
import torchvision
from torch import nn
from torch import optim
from torchvision import transforms as T
from torchvision import models as M
from torch.utils.data import Dataset, DataLoader
import torch.nn.functional as F
import matplotlib as mlp
import matplotlib.pyplot as plt
import seaborn as sns
import random
import numpy as np
import pandas as pd
import datetime
from time import time
import gc
torch.manual_seed(1412)
random.seed(1412)
np.random.seed(1412)
torch.cuda.is_available()
#True
device = torch.device("cuda" if torch.cuda.is_available() else "cpu")
device
#device(type='cuda')
【案例】Kaggle医学影像识别
1 案例背景与benchmark建立
深度学习最为关键的应用领域之一是医疗医护及卫生领域。医疗是一个与人类生存紧密相关的行业,也是一个疑难问题浩如烟海、资源稀缺、供不应求的行业。在2020年,全球大约有3.8亿人缺乏最低程度的医疗保障,禽流感、新冠等全球传染病更是时刻冲击着全球医疗系统,数以亿计的患者与医护人员渴望更多的资源、更高的效率、更低的成本,深度学习正是一项提效减负的关键技术。今天,深度学习被广泛应用于医疗行业的各个领域,例如药物研究、蛋白质合成、基因组学分析、药方审计、基础诊断、心理健康等。其中,以计算机视觉技术为代表的医疗影像识别是最为成熟、需求也最为广泛的应用。
计算机视觉可以识别核磁共振图像、CT(断层扫描)图像、超声影像、病理学图像、放射学图像等常见的医疗成像数据,这些影像往往覆盖病变组织或健康组织,被广泛应用于临床诊断和手术辅助。深度学习模型可以通过结合这些成像数据的各个方面来做出有效的诊断和解释,例如组织大小、体积、形状、颜色、质感等。现在,深度学习在糖尿病视网膜病变检测、阿尔兹海默症的早期检测、乳腺结节等超声检测方面已经有较多研究成果。在2018年,卷积神经网络在皮肤科图像中识别出黑色素瘤疾病的准确率已经比专家高出10%以上。
在本次案例课程当中,我们要完成的是病理图像识别诊断。病理检查是当代最为可靠的肿瘤性质确诊方式,其结果就是临床的最后诊断,病理图像就是病变组织在光学显微镜下所呈现的形态图像,病理学家需要观察该图像判定肿瘤的性质(良性/恶性,如果恶性具体是什么病变亚型),同时也需要判定肿瘤的侵略性分级(早期/中期/晚期)。
然而,为执行病理检查,患者需要在疑似肿瘤的组织上提取部分细胞或器官。若肿瘤为恶性,此行为相当于在癌变组织上创造一个伤口,携带癌细胞的血液会外溢并扩散至全身,极大程度地提升癌症转移可能性、降低患者生存概率。因此,能够迅速、准确地做出病理诊断,对癌症患者具有极大的意义。
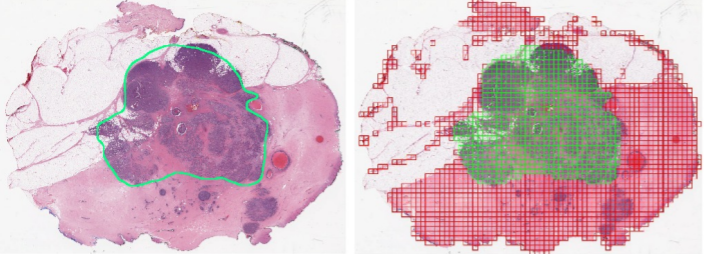
在我们的案例当中,我们将使用卷积神经网络代替病理专家和医护人员,识别出病理图像中所有带恶性病灶的区域。如图所示,左侧是带有恶性病灶的原始图像,右侧则是通过深度学习完成的像素级识别。在原始图像中,病理专家人为绘制了绿色线作为标签,绿色线内部的像素则被标记为恶性病灶(标记为1),绿色线外部的像素则被标记为非恶性病灶(标记为0)。而深度学习的工作,就是利用卷积神经网络精准判断出带有恶性病灶的像素块,以判断病理图像中是否含有恶性病灶,若存在恶性病灶,也可快速精准判断出病灶大小及扩散程度,帮助患者确定肿瘤的侵略性分级。
在本次案例中,我们所使用的数据是美国国家生物技术信息中心(NCBI)所提供真实的复现浸润性导管癌(IDC)病理检查图像,由搭载标本的整体载玻片按40倍放大扫描而来,图像的像素大约在200w~400w之间(图片大小为1200*1920以上)。该数据的完整版本首次出现于哥伦比亚大学的论文《使用卷积神经网络自动检测病理图像中的浸润性导管癌》(Automatic detection of invasive ductal carcinoma in whole slide images with Convolutional Neural Networks),后来Kaggle联合美国国家医学图书馆共同整理、披露了部分患者数据,专供深度学习研究者使用。
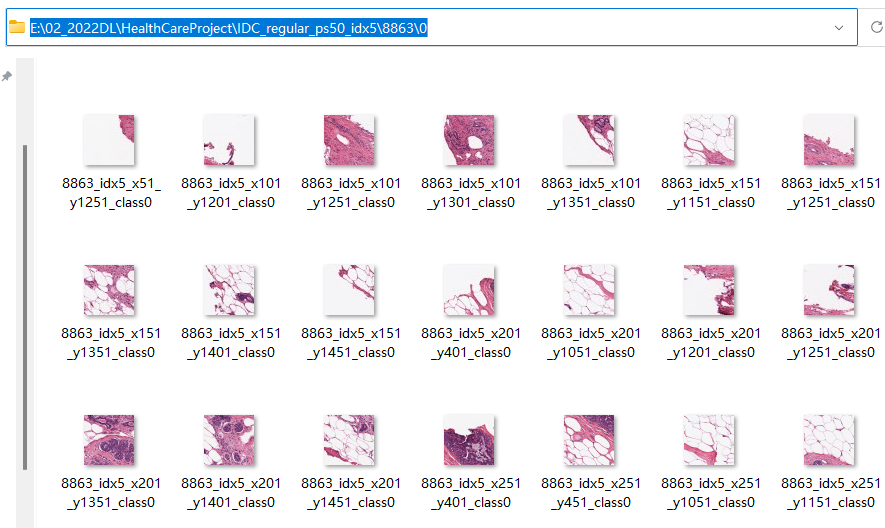
为实现像素级识别,研究团队在论文当中将所有图像切分成了50x50的方片数据(patch)。在课程当中,我们提供了分割后的数据压缩文件:IDC_regular_ps50_idx5.zip。解压之后,即可看到以患者ID为名的文件夹:
每个文件夹内部即是标签为0和为1的文件夹,其中标签0表示非恶性病灶,标签1表示恶性病灶:
标签文件夹下是被分割后的方片数据。分割后,每张图像文件名一般呈现为10253_idx5_x1351_y1101_class0.png的格式,其中idx5之前的数字为患者ID,最后的class为样本的性质,其中class0代表非恶性(也称阴性),class1代表恶性(阳性)。中间的x数字_y数字则表示该方片的左上角位于原始图像中的坐标。
在本次案例当中,我们的目标是超越论文《使用卷积神经网络自动检测病理图像中的浸润性导管癌》中呈现的结果。在论文当中,哥伦比亚大学使用了F1分数与平衡后的准确率(Balanced Accuracy,简写为BAC)。
- 将论文中提供的最好结果作为我们的benchmark
| 精确度 Pr | 召回率/敏感度 Rc/Sen | 特异度 Spc | F1分数 | BAC | |
|---|---|---|---|---|---|
| ColUni | 0.6540 | 0.7960 | 0.8886 | 0.7180 | 0.8423 |
其中,两个核心评估指标的计算公式为:
F 1 = 2 ? P r ? R c P r + R c F1 = \frac{2* Pr * Rc}{Pr + Rc} F1=Pr+Rc2?Pr?Rc?
B A C = S e n + S p c 2 BAC = \frac{Sen + Spc}{2} BAC=2Sen+Spc?
通常来说我们是向着某个损失最小化的方向训练卷积神经网络,因此PyTorch中默认提供的迭代目标基本都是损失函数。为保持与论文中评估指标一致,我们可以自定义损失及评估指标。接下来,就让我们以超越0.8423的BAC为目标开启我们的案例。
2 基于OpenCV批量分片高像素影像
为了更加精确的诊断和治疗,医疗影像往往是大像素(1920x1080)或超大像素图像(4k图像4096x2160)。这类图像的尺寸与深度学习实验数据常见尺寸(227x227,或32x32)有巨大的差别,不仅对网络的深度要求更高,所需的算力、内存、运算时间等成本也会更高。因此,在处理医疗图像或遥感图像这样的高像素图像时,往往都需要将其批量处理成小像素方片,并针对每一个方片进行识别或预测。
幸运的是,在本次案例当中所使用的IDC病理检查图像已经被哥伦比亚大学的论文团队分割成了小像素方片,因此无需我们再人工进行处理,然而掌握高像素图像的分片技巧十分必要,因此本节我们将以数张大像素的图像为例展示批量分片高像素图像的代码与相关知识。
注意,考虑到各类器官的高像素病灶影像可能引起不适,本节我们将不会使用IDC病理检查数据集的原始图像,而是使用与病理图像高度相似的星辰图来替代。星辰与病灶细胞一样,可能分布在图像的各个位置,也可能集中在图像上的某个区域,因此适合用来作为替代图。所有图像都位于文件夹Universe当中。我们先以一张图像为例进行分片:
#!pip install opencv-python
import cv2
import os
import matplotlib.pyplot as plt
cv2.__version__
#'4.5.5'
- 导入图像,进行查看
#设置图像所在的文件夹目录,你可以自由设置你自己的目录
PATH = "/Users/zhucan/Desktop/桌面内容/00 深度学习二期课程课件/Lesson 18/data/Universe"
#使用opencv读入图像
img = cv2.imread(os.path.join(PATH,"Universe01.jpg"))
#由于opencv在读取图像时,没有成功读取也不会报错
#因此在读取图像后需要检查图像是否为None
if img is None:
print("failed")
else:
print("succeed")
#succeed
img.shape #高,宽
#(1600, 2560, 3)
#将图像进行可视化,由于OpenCV的默认通道顺序是BGR,因此在可视化时需要转换为RGB
#对图像进行浅复制,避免在操作中误覆盖原图
imgcopy = cv2.cvtColor(img, cv2.COLOR_BGR2RGB).copy()
plt.figure(dpi=200)
plt.imshow(imgcopy)
plt.axis("off");

imgcopy.shape #注意判断导入后的高宽顺序。我们平时说1920x1080是宽x高,现在导入后明显是高x宽
#(1600, 2560, 3)
#像素值
imgcopy.shape[0] * imgcopy.shape[1]
#4096000
我们的最终目标:将图像分割为数千个小型方片:

- 将一张图像分割为两半
分割的本质就是先裁剪图像、再对裁剪后的图像进行保存。例如,假设我们现在要将以下图像按中线分割为两部分,最简单的手段就是先在原始图像上裁剪出左半边的图像,保存为一个单独的文件,再在原始图像上裁剪出右半边的图像,保存为一个单独的文件。在这个过程当中,我们没有对原始图像做任何的更改,却可以得到两张新的图像。
由于图像数据都是三维矩阵,因此只要通过索引就可以轻松将图像进行裁剪。对一张图像来说,索引的顺序是从上到下、从左到右,因此,如果要取出整个图像的左侧,则需要对图像进行如下索引:
imgcopy.shape #2560
#(1600, 2560, 3)
imgcopy[:,0:1280].shape #取出一半的宽,全部的高,不对通道做任何处理
#(1600, 1280, 3)
#尝试将索引出的图像可视化
plt.figure(dpi=140)
plt.imshow(imgcopy[:,0:1280])
plt.axis("off");

#也可以尝试索引出图像的右侧
plt.figure(dpi=140)
plt.imshow(imgcopy[:,1280:]) #依然是全部的高,不过宽是从1280中线开始向后取
plt.axis("off");

不难发现,对图像进行截取时,只需要按照图像[高的起点:高的终点,宽的起点:宽的终点]进行索引即可,只要我们知道图像当中的相对位置(四个坐标),就可以按照任意的起点和终点截取任何尺寸、任何形状的图片。接下来,当我们索引出想要的图像后,只需要将这张图像保存成新的图片文件即可。在这里我们要使用的是opencv中的经典方法imwrite:
cv2.imwrite(保存图像的目录+文件名,需要保存的图片对象)
例如,上述星辰图的左侧保存到之前设置好的PATH目录,则有:
PATH
#'/Users/zhucan/Desktop/桌面内容/00 深度学习二期课程课件/Lesson 18/data/Universe'
os.path.join(PATH,"star1.jpg") #确定文件名
#'/Users/zhucan/Desktop/桌面内容/00 深度学习二期课程课件/Lesson 18/data/Universe/star1.jpg'
cv2.imwrite(os.path.join(PATH,"star2.jpg"),imgcopy[:,0:1280])
#True
返回True,则说明保存成功,如果报错或返回False则保存失败。此时我们可以在PATH设定的目录下查看保存出的文件。
- 将一张图像分割为小尺寸方片
将图像分割为小尺寸方片的原理与将图像分割成两半的原理完全一致:首先,从图像中裁剪出我们需要的小尺寸方片,然后将该方片对象保存为一张新的图像。然而,一张大尺寸图像可以被分割为成千上万的小尺寸方片,因此我们需要将每个小方片一一索引出来,再一一保存:
我们必然不可能手动一一裁剪方片,因此我们需要一个循环来帮助我们进行分割。观察上图,每个方片的尺寸为50x50,左上角第一个被切分出的方片索引为图像[:50,:50],紧接着左数第二个方片的索引为图像[:50,50:100],第三个方片的索引为图像[:50,100:150]以此类推,第一行的所有方片都可以被表示为图像[:50,x:x+50]。可见,我们只需要在宽度上进行循环,每次让宽的起点增加50,宽的终点增加50,就可以实现第一行的截取和分割。同理,第一列的所有方片都可以被表示为图像[y:y+50,:50],所以只要在高度上进行循环,每次让高的起点增加50,高的终点增加50,就可以实现第一列的截取和分割。那只要同时在高和宽上进行循环,就可以实现对上图中所有方片的截取和分割。
以宽为例,我们可以有:
imgcopy.shape[1]
#2560
[*range(0,imgcopy.shape[1],50)] #从0开始,到宽的最大值结束,每50个数取出一个数
# [0,
# 50,
# 100,
# 150,
# 200,
# 250,
# 300,
# 350,
# 400,
# 450,
# 500,
# 550,
# 600,
# 650,
# 700,
# 750,
# 800,
# 850,
# 900,
# 950,
# 1000,
# 1050,
# 1100,
# 1150,
# 1200,
# 1250,
# 1300,
# 1350,
# 1400,
# 1450,
# 1500,
# 1550,
# 1600,
# 1650,
# 1700,
# 1750,
# 1800,
# 1850,
# 1900,
# 1950,
# 2000,
# 2050,
# 2100,
# 2150,
# 2200,
# 2250,
# 2300,
# 2350,
# 2400,
# 2450,
# 2500,
# 2550]
for x in range(0,imgcopy.shape[1],50): #把range中的值作为宽的起点,再给该起点加上50像素,作为终点
print("[{}:{}]".format(x,x+50))
[0:50]
# [50:100]
# [100:150]
# [150:200]
# [200:250]
# [250:300]
# [300:350]
# [350:400]
# [400:450]
# [450:500]
# [500:550]
# [550:600]
# [600:650]
# [650:700]
# [700:750]
# [750:800]
# [800:850]
# [850:900]
# [900:950]
# [950:1000]
# [1000:1050]
# [1050:1100]
# [1100:1150]
# [1150:1200]
# [1200:1250]
# [1250:1300]
# [1300:1350]
# [1350:1400]
# [1400:1450]
# [1450:1500]
# [1500:1550]
# [1550:1600]
# [1600:1650]
# [1650:1700]
# [1700:1750]
# [1750:1800]
# [1800:1850]
# [1850:1900]
# [1900:1950]
# [1950:2000]
# [2000:2050]
# [2050:2100]
# [2100:2150]
# [2150:2200]
# [2200:2250]
# [2250:2300]
# [2300:2350]
# [2350:2400]
# [2400:2450]
# [2450:2500]
# [2500:2550]
# [2550:2600]
imgcopy[:50,2550:2600].shape
#(50, 10, 3)
现在,同时对宽和高执行循环:
#每次分割前,获取完整的原始图像
imgcopy = cv2.cvtColor(img, cv2.COLOR_BGR2RGB).copy()
#获取图像的高度和宽度
imgheight = imgcopy.shape[0]
imgwidth = imgcopy.shape[1]
#确定每个方片的尺寸
patch_height = 50
patch_weight = 50
#令x为宽上的起点,y为高上的起点,开始循环
for y in range(0, imgheight, patch_height):
for x in range(0, imgwidth, patch_weight):
#首先检查:设置的方片尺寸是否大于原始图像尺寸?如果是,则直接打断循环,不进行分割
if patch_height > imgheight or patch_weight > imgwidth:
print("方片尺寸过大,超过原始尺寸")
break
#方片尺寸没有大于原始图像尺寸,则计算宽和高上的终点
y_ = y + patch_height
x_ = x + patch_weight
#接着检查,宽和高上索引的终点是否大于了原始图像尺寸?
#当起点位于图像上,但终点落在图像外的时候,我们可以规定几种选择
#舍弃所有不足规定方片尺寸的图像,或者保存这些残缺的图像
#对于病理识别来说,损失边缘的一点点信息并无大碍
#因此我们规定,只有当高和宽的终点都落在图像上时,才进行切片和保存
if imgheight >= y_ and imgwidth >= x_:
#索引出切片
patch = imgcopy[y:y_, x:x_]
#保存索引出的图像,如果想,可以在图像文件名中保存四角坐标,也可以选择只保存左上角坐标
cv2.imwrite(os.path.join(PATH,"patches"
,"universe"+"x"+str(x)+"_"+str(x_)+"y"+str(y)+"_"+ str(y_) +".jpg")
, patch)
#保存之后,在原始图像上对当前索引出的区域绘制白色边框
#注意这一操作将会在正在被切片的图像上进行
cv2.rectangle(imgcopy #要绘制长方体的对象
, (x, y), (x_, y_) #整个正方形的左上角和右下角的坐标
, (255, 255, 255) #使用的颜色
, 2 #线条的粗细,数字越大越粗
)
#循环完毕后,绘制被我们分割后的图像
plt.figure(dpi=300)
plt.imshow(imgcopy)
plt.axis("off");

现在可以检查目标文件夹,观察所有被分割的图像了。
- 批量处理大型图像
当我们有大量的大型图像需要处理时,我们首先要从文件夹中将所有的图像都读入。在这里,我们需要借助os库下著名函数listdir的力量:
os.listdir(path):读取目录中所有的文件名
只要有文件名,我们就可以在cv2.imread当中按照文件名一一读入相应的图像。同时,对于不是图片数据的文件名,cv.imread会读取失败,从而导致读入的对象为None。因此我们也需要对读入的对象进行检查,对于任何不是None的对象(也就是对于任意成功读取的图像),我们将其保存在列表当中:
def load_images_from_folder(folder):
"""批量读取文件夹中的图片"""
images = [] #将准备读取的图像保存在列表当中,因为arrays是不允许在循环中逐渐添加对象的
for filename in os.listdir(folder):
img = cv2.imread(os.path.join(folder,filename))
if img is not None:
images.append(img)
return images
images = load_images_from_folder(PATH)
PATH #现在该目录下总共有4张星辰图像,尺寸大小各不相同
#'/Users/zhucan/Desktop/桌面内容/00 深度学习二期课程课件/Lesson 18/data/Universe'
os.listdir(PATH) #其中patches是文件夹,因此被忽略了,没有被保存到列表images当中
#['patches',
# 'Universe01.jpg',
# 'Universe02.jpg',
# 'Universe03.webp',
# 'Universe04.jpg']
len(images) #列表,因此无法使用.shape功能
#4
#展示4张星辰图像
for i in images:
plt.figure(dpi=200)
plt.imshow(i)
plt.axis("off");
#现在的通道不是RGB而是GBR




接下来我们就可以批量分割现有图像了。每张图像会被分割成数千个切片,我们希望将来自一张图像的切片保存在同一个文件夹当中,并且该文件夹的名称就是该图片的名称。因此我们需要提取出每张图片的文件名(去掉拓展名),并且使用os中两个用于创建目录的函数:
os.mkdir(path):创建path所指定的目录。注意,只有当path所指定的目录还不存在时有效,如果该目录已经存在,则报错。
os.path.exists(path):检查path所制定的目录是否已经存在。
os.listdir(PATH)
#['patches',
# 'Universe01.jpg',
# 'Universe02.jpg',
# 'Universe03.webp',
# 'Universe04.jpg']
os.listdir(PATH)[1].split(".")[0] #取出文件名中不包括拓展名的部分
#'Universe01'
os.path.join(PATH,os.listdir(PATH)[1].split(".")[0])
#'/Users/zhucan/Desktop/桌面内容/00 深度学习二期课程课件/Lesson 18/data/Universe/patches'
os.path.exists(os.path.join(PATH,"Universe01"))#判断文件夹是否存在
#False
os.path.exists(os.path.join(PATH,"Universe01.jpg"))#判断文件是否存在
#True
def extract_images_from_folder(folder):
"""对图像进行批量分片(对一个文件夹中所有的图像进行分片)"""
#图像导入
for filename in os.listdir(folder):
img = cv2.imread(os.path.join(folder,filename))
if img is not None:
#如果导入成功,则创建该图片专属的文件夹
subfolder = os.path.join(PATH,filename.split(".")[0])
if os.path.exists(subfolder):
print("folder exists")
else:
os.mkdir(subfolder)
#开始分割,所有被分割出的切片都位于该图片的文件夹中
imgcopy = cv2.cvtColor(img, cv2.COLOR_BGR2RGB).copy()
imgheight = imgcopy.shape[0]
imgwidth = imgcopy.shape[1]
patch_height = 50
patch_weight = 50
for y in range(0, imgheight, patch_height):
for x in range(0, imgwidth, patch_weight):
if patch_height > imgheight or patch_weight > imgwidth:
break
y_ = y + patch_height
x_ = x + patch_weight
if imgheight >= y_ and imgwidth >= x_:
patch = imgcopy[y:y_, x:x_]
#将每一张图像保存到单独的文件夹
cv2.imwrite(os.path.join(subfolder,str(filename.split(".")[0])+"x"+str(x)+"_"+str(x_)+"y"+str(y)+"_"+ str(y_) +".jpg")
, patch)
#保存之后,在原始图像上对当前索引出的区域绘制白色边框
#注意这一操作将会在正在被切片的图像上进行
cv2.rectangle(imgcopy #要绘制长方体的对象
, (x, y), (x_, y_) #绘制长方体的4角的坐标
, (255, 255, 255) #使用的颜色
, 2 #线条的粗细,数字越大越粗
)
#循环完毕后,绘制被我们分割后的图像
plt.figure(dpi=300)
plt.imshow(imgcopy)
plt.axis("off");
extract_images_from_folder(PATH)




好了,现在你可以使用函数load_images_from_folder和extract_images_from_folder对任意图像进行批量分割处理了。你还可以改进这个函数,例如将每张图像需要分割的切片尺寸大小作为函数的参数,例如将是否在分割完毕后打印分割后图像作为函数的参数(毕竟如果需要分割的图像太多的话,也无需打印每一张的分割结果)。同时,你还可以加入更多功能,例如打印每一张待分割图像的尺寸,或者对每张图像分割后的切片数进行计数等等。你可以调用一切你需要的功能来完善这两个函数。
3 数据导入与数据探索
将数据分割为小方片后,我们可以开始进行数据探索了。对于一般图像数据,我们的探索一般包括以下几个步骤:
- 探索数据量/数据结构,包括图像的尺寸、数量、通道数,以确认所需的算力、架构深度以及验证策略
- 可视化图像,确认图像中的具体内容,以确定所需要使用的预处理方法
- 标签分布探索,探索类别数量、样本均衡情况,以确定迭代用的损失函数,以及是否进行均衡处理
对于被分片的病理图像,我们可能还需要:
- 查看完整数据的尺寸,以判断切除的病灶大小,切除病灶越小,说明患者的生存率越大
- 可视化完整数据,对病理数据来说,特别的,需要确定恶性病灶的占比
幸运的是,目前我们使用的数据集为普通zip压缩文件,因此只要解压缩之后就可以直接查看每张图片的大致情况,因此我们可以或许省略可视化图像这一步骤。但对于大部分深度学习所使用的图像数据集来说,还是需要读取数据后单独对图像进行查看。
- 探索数据量
#首先统计图像的总量,将包含所有患者ID文件夹的目录作为PATH,所有文件都在该PATH目录下
PATH = "/Users/zhucan/Desktop/桌面内容/00 深度学习二期课程课件/Lesson 18/data/IDC_regular_ps50_idx5"
#目录下一共有多少个患者(多少个文件夹)?
patients = os.listdir(PATH)
len(patients) #279个患者 - 279张原始病理图像
#279
patients[:10] #文件夹名称就是患者ID
#['9036',
# '10268',
# '10257',
# '8913',
# '13613',
# '8914',
# '15510',
# '10259',
# '16165',
# '10292']
#将PATH目录下所有的图像统计出来
positive_patches = 0
negative_patches = 0
#在患者ID中进行循环
for patient_id in patients:
#在每个患者ID文件夹下,还有标签文件夹,因此需要进入每个标签文件夹读取相应的图像
#阴性样本
#设置每个标签文件夹的目录:
class0_path = os.path.join(PATH,patient_id,str(0))
#读取当前标签文件夹下所有的文件(也就是所有的方片图像)
class0_patches = os.listdir(class0_path)
#计算所有方片图像的总数
negative_patches += len(class0_patches)
#阳性样本
class1_path = os.path.join(PATH,patient_id,str(1))
class1_patches = os.listdir(class1_path)
positive_patches += len(class1_patches)
positive_patches
#78786
negative_patches
#198738
total_patches = positive_patches + negative_patches
total_patches
#277524
现在我们可以获得的数据集中包含279个患者的病理图像,一共27.7w张方片图像,其中19.8w数据为阴性(良性病灶,标记为0),7.8w数据呈现为阳性(恶性病灶,标记为1)。图像的数据量超过10w级别,整体数据量较大,在运行时需要注意内存管理,如果可能尽量提供GPU作为运算资源。不过,这些数据来源于仅仅279位患者,患者整体数量较小,因此可能存在少数患者的病理信息对全局影响很大的情况。由于涉及到患者隐私及医疗伦理道德问题,医疗数据在现实当中是比较难以获取且公开的,因此基于医疗数据训练神经网络时必须时刻注意过拟合情况。
依据患者数量以及较高的过拟合风险,最佳方案是使用交叉验证,然而又考虑到较大的数据总量,在训练过程中多次执行交叉验证并不现实,因此综合来看我们需要将数据分割成训练、测试和验证集。我们首先在测试数据上寻找较好的分数,然后在验证集上进行验证,进一步验证模型的泛化能力。
- 探索数据结构
现在,我们将全部图像导入,进行可视化及结构查看。考虑到导入目录的复杂程度(先进入患者ID、再进入标签类别、在读取具体文件),我们最好将每张图片的具体目录和标签都统计成表,再做后续打算。先来建立DataFrame:
data = pd.DataFrame(index=np.arange(0, total_patches) #对应着每一张具体的图像
, columns=["patient_id", "path", "target"]
)
#批量读取目录、ID和标签
#由于需要对27w张图片的情况进行一一查看,因此本段循环运行较为缓慢
idx = 0
for patient_id in patients:
#对每个患者的文件夹,读取其中所有标签下的图像信息
for label in [0,1]:
class_path = os.path.join(PATH,patient_id,str(label))
class_patches = os.listdir(class_path)
#对每个标签下的每张图像,记录
for patch in class_patches:
data.loc[idx,"path"] = os.path.join(class_path,patch)
data.loc[idx,"target"] = label
data.loc[idx,"patient_id"] = patient_id
idx += 1
idx
#277524
data.shape #确认所有图像都被记录了
#(277524, 3)
data.head()

data.tail()

data["path"][0]
#'/Users/zhucan/Desktop/桌面内容/00 深度学习二期课程课件/Lesson 18/data/IDC_regular_ps50_idx5/9036/0/9036_idx5_x1051_y2401_class0.png'
(data.isnull()).sum() #没有任何缺失值
#patient_id 0
#path 0
#target 0
#dtype: int64
现在,我们可以随机选取图像进行可视化、并且检查图像的结构了。对于方片数据,我们可以直接实现5行10列的排列:
#从27w样本中抽样出需要被绘制的样本
selection = np.random.choice(data.index.values,size=50,replace=False)
selection
#array([237922, 188802, 23162, 277266, 101595, 149525, 55527, 174793,
# 250296, 175799, 258059, 176303, 117797, 161237, 159705, 124159,
# 222249, 205470, 205385, 79903, 34746, 143075, 75475, 110199,
# 11561, 84174, 16009, 10119, 17302, 243635, 196599, 110294,
# 119807, 122098, 150885, 162121, 247141, 34071, 253748, 253621,
# 95748, 109532, 208542, 239776, 215894, 229000, 107687, 9304,
# 101998, 24959], dtype=int64)
#尝试运行下面的代码,理解代码的优化
for n in range(5):
for m in range(10):
print(m + 10*n)
"""
for n in range(a):
for m in range(b):
m + b*n
range(0,a*b)
"""
#建立画布,5行10列,共50张图
fig, axs = plt.subplots(5,10,figsize=(20,10))
#需要为画布上的每张图填上内容,因此在行/列中循环
for n in range(5):
for m in range(10):
#按索引顺序,逐渐索引出selection中的图像
idx = selection[m+10*n]
#一个常见的替换代码是:随机抽样50张图像的索引
#idx = np.random.choice(data.index.values)
#但这一行代码的运行速度比我们使用的代码要慢得多
#导入图像,并检查图像是否存在
image = cv2.imread(data.loc[idx,"path"])
#图像存在,则转化为RGB,准备绘图。并且打印图像的结构。
if image is not None:
image = cv2.cvtColor(image, cv2.COLOR_BGR2RGB)
print(image.shape)
else:
print("No Image")
#导入成功后,在对应的行列位置显示图像
axs[n,m].imshow(image)
axs[n,m].grid(False)
axs[n,m].axis("off")
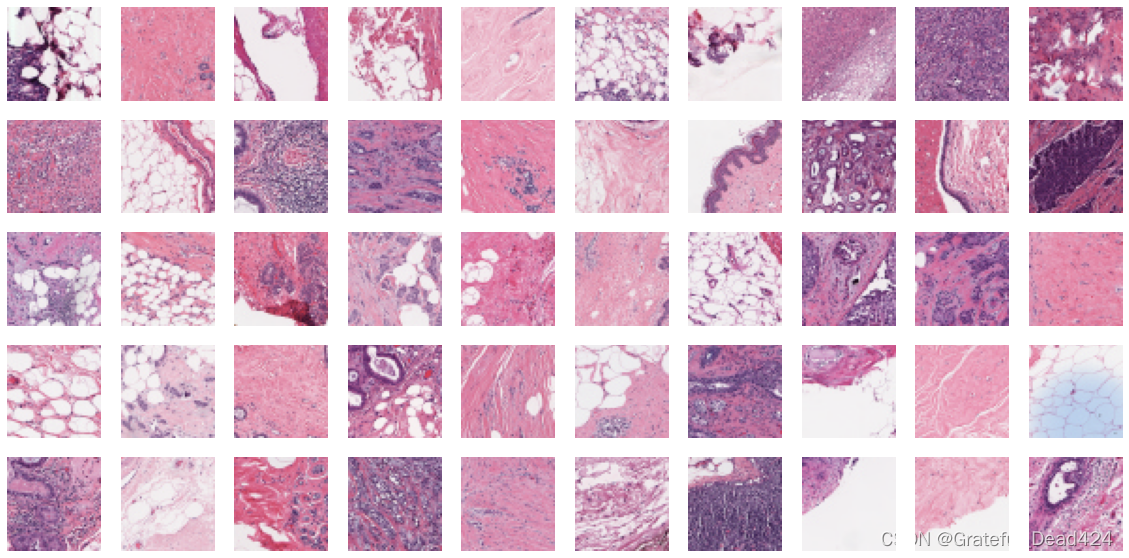
首先观察图像的结构,所有的图像都是50x50的3通道图像。一般切片图像的结构都较为规整,原始图像的结构会更加复杂。如果发现不同的图像呈现不同的尺寸,则需要进行规整尺寸的预处理,反之则可以选择不处理。然而,考虑到医学影像的处理难度,以及各类经典神经网络常见的图像输入尺寸,我们优先考虑将50x50的尺寸放大为227x227,并使用深度较深的网络进行训练。
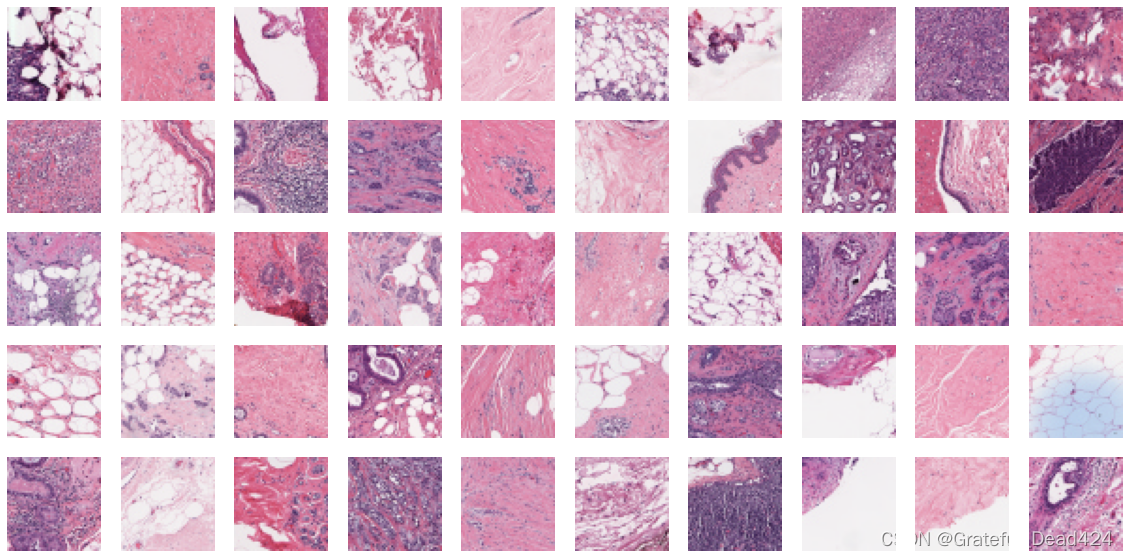
从可视化的结果来看,图像颜色深浅、线条粗细、轮廓等细节是千变万化的,在不具有专业知识的情况下,肉眼难以判断出具体病灶的情况。我们可以尝试单独可视化恶性区域和良性区域,看看能否发现一些区别:
pos_selection = np.random.choice(data[data.target==1].index.values, size=50, replace=False)
neg_selection = np.random.choice(data[data.target==0].index.values, size=50, replace=False)
#恶性样本可视化
fig, axs = plt.subplots(5,10,figsize=(20,10))
for n in range(5):
for m in range(10):
idx = pos_selection[m + 10*n]
image = cv2.imread(data.loc[idx, "path"])
if image is not None:
image = cv2.cvtColor(image, cv2.COLOR_BGR2RGB)
axs[n,m].imshow(image)
axs[n,m].grid(False)
axs[n,m].axis("off")
#良性样本可视化
fig, axs = plt.subplots(5,10,figsize=(20,10))
for n in range(5):
for m in range(10):
idx = neg_selection[m + 10*n]
image = cv2.imread(data.loc[idx, "path"])
if image is not None:
image = cv2.cvtColor(image, cv2.COLOR_BGR2RGB)
axs[n,m].imshow(image)
axs[n,m].grid(False)
axs[n,m].axis("off")
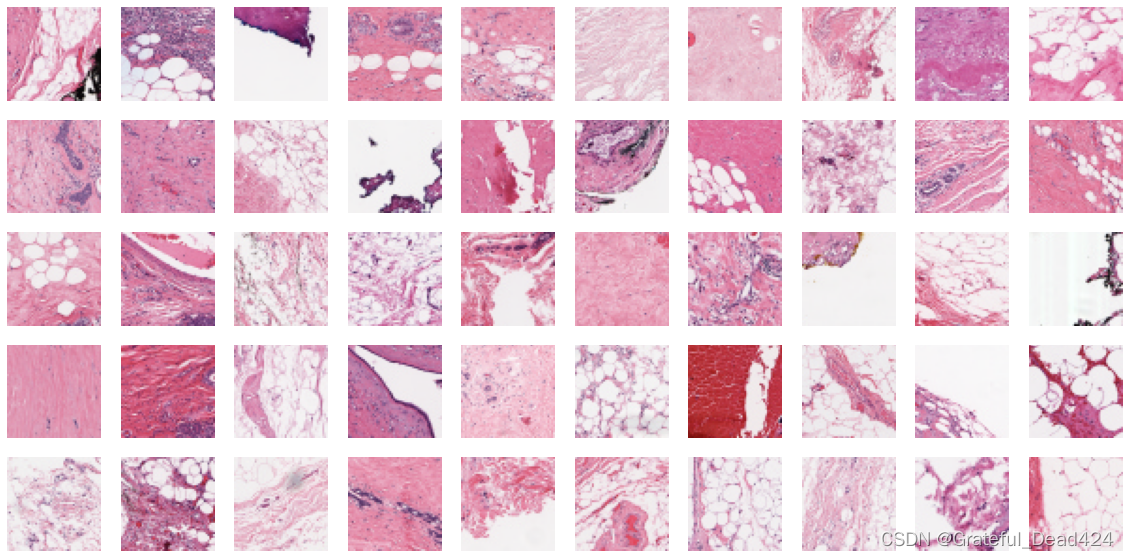
不难发现,良性样本整体呈现的颜色比恶性样本要浅,色彩饱和度更高、同时组织结构看起来也相对简单一些。恶性样本中会存在粗糙、密集且颜色较深的结构。当然,部分位于恶性病变边缘的方片可能混杂在两种样本当中,使得良性样本中也会出现一些深色的方片,而恶性样本中也会出现一切浅色的方片。
- 标签探索
data.head()

data["target"].value_counts() #样本不均衡,但情况不是非常严重
#0 198738
#1 78786
#Name: target, dtype: int64
data["target"].value_counts()[1]/data["target"].shape[0] #少数类占比为28.38%
#0.28388896095472826
在许多人看来,少数类比例较少(恶性比例较少)可能意味着患者的病情并不严重,但事实上并非如此。
首先,在肿瘤治疗过程中,如果能够只切除肿瘤、保留器官,则一定不会将器官整个切除。因此,如果整个病理图像上只有少许组织和器官(即一个患者文件夹当中的图像数量较少),那被切除的可能是肿瘤本身,恶性切片的覆盖率可能达到90%甚至100%。出现这种情况往往说明患者的病情较轻,可以通过清除肿瘤组织和肿瘤细胞来进行治疗。
相反,如果一个患者的病理图像尺寸越大(即一个患者文件夹当中的图像数量较多),则说明被切除的器官越大,通常意味着病情已经较为严重。例如,以肿瘤为核心,肿瘤细胞已经扩散到整个器官,必须切除整个器官,或者,病变的亚型恶性较高、发展较快,为降低转移风险而主动切除整个器官。这类患者恶性切片的比例可能较低,但是病变程度更加严重。因此我们的少数类比例反映出来的现实是:提供这批真实影像的患者中可能包含许多病情较为严重、需要切除整个器官的患者。
当然,在手术和病情的复杂具体情况下(如患者年龄较大、医生的流派较为保守、或患者本人要求切除整个器官、患者本人不再需要使用该器官),并不能排除有患者在病情较轻、且肿瘤较小的情况下依然切除了大部分组织甚至整个器官。同时,也不排除某些患者体型较大、身材魁梧,因此器官比一般器官更大的情况。这些患者一般属于少数人,因此我们可以不必考虑他们的情况。
#我们查看每个病人的病理图像尺寸,如果图像尺寸越大,则说明被切除的器官越大
#通常被切除的部分越大,意味着病情越严重
#当然,也有可能因为病变的亚型恶性较高、发展较快、为降低转移风险而主动切除整个器官
data.columns
#Index(['patient_id', 'path', 'target'], dtype='object')
data.head()

data.groupby("patient_id")["target"].count().describe()
#count 279.000000
#mean 994.709677
#std 550.603474
#min 63.000000
#25% 560.500000
#50% 967.000000
#75% 1362.000000
#max 2395.000000
#Name: target, dtype: float64
根据统计结果,这批患者的病理图像最小值接近0、最大值接近2400,均值为994,表现相对均匀。这说明大部分患者还是只切除了部分组织,但只切除少许肿瘤组织的患者很少。
#我们可以计算每个患者的图像中恶性方片所占的比例,整体图像较大且恶性方片比例较高的患者病情较为严重
#以患者ID为横轴,标签为纵轴,计数(count)为值整理表格
table = data.pivot_table(index="patient_id",columns="target"
,aggfunc="count")
table.head()

#整理索引,去掉索引的层次
table.columns = ["0","1"]
table.index = patients
table.head()

table["wholepicture"] = table["0"] + table["1"]
table["positive_ratio"] = table["1"]/table["wholepicture"]
pd.set_option('display.max_rows',None)
table.sort_values("positive_ratio",ascending=False).head(10)

不难发现,恶性切片占比较高的前10名患者中只有3人的病理图像包含超过1000个切片,说明大部分恶性切片占比较高的患者都是病症较轻、可以直接切除肿瘤组织的患者。
table.sort_values("positive_ratio",ascending=True).head(10)

相对的,恶性切片占比最低的前10名患者的病理图像尺寸都较大,只有1人的病理图像包含1000个以下切片,说明大部分恶性切片占比较低的患者反而是重症患者、需要切除整个器官。当然,如果患者的整体病理图像尺寸较大、且恶性切片占比也较大,则说明该患者的病情最为严重。
#我们还可以绘制单个患者完整的病理图像,并标注出其中恶性的部分,以观察病理图像尺寸是否与恶性程度相关
观察我们每一个切片的文件名,不难发现我们有该切片位于原始病理图像上的坐标点位置,只要我们找出每个切片所对应的坐标点位置,就可以复原完整的病理图像上、恶性组织所占的部分。
data.loc[0,"path"]
#'E:\\02_2022DL\\HealthCareProject\\IDC_regular_ps50_idx5\\10253\\0\\10253_idx5_x1001_y1001_class0.png'
data.head()

data.loc[10,"path"]
#'E:\\02_2022DL\\HealthCareProject\\IDC_regular_ps50_idx5\\10253\\0\\10253_idx5_x1001_y1701_class0.png'
data["x"] = data["path"].apply(lambda x: int(x.split("_")[-3][1:]))
data["y"] = data["path"].apply(lambda x: int(x.split("_")[-2][1:]))
data.head() #x和y分别为切片所在位置的横坐标与纵坐标
def visualize_single_patient(patient_id):
"""绘制单个患者的病理图像及恶性占比"""
#先从data中提取出该患者所对应的全部切片
singlepatient = data[data["patient_id"] == patient_id]
#以切片的横坐标与纵坐标绘制散点图,并使用标签标注颜色。恶性部分标注为红色,良性部分标注为紫色
sns.set()
plt.scatter(singlepatient["x"],singlepatient["y"],c=singlepatient["target"],cmap="coolwarm",s=10);
plt.grid(linewidth=1,color="white")
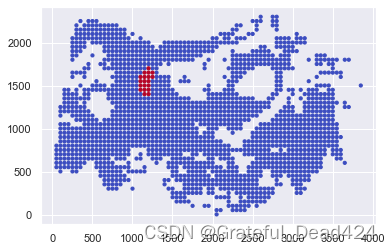
visualize_single_patient("10305") #恶性率最低的患者,这可能说明患者的肿瘤亚型较凶猛
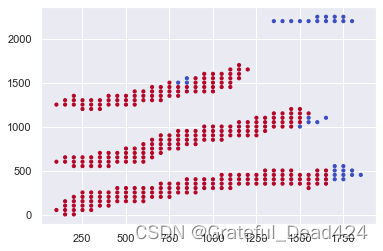
visualize_single_patient("14209") #恶性率最高的患者,病症较轻,只切除了部分组织
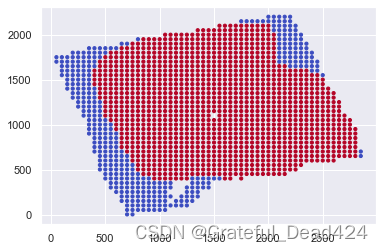
visualize_single_patient("9077") #恶性率高,且病理图像较大的患者,病症较重
for n in range(5):
for m in range(3):
print(m+3*n)
# 0
# 1
# 2
# 3
# 4
# 5
# 6
# 7
# 8
# 9
# 10
# 11
# 12
# 13
# 14
np.random.shuffle(data["patient_id"].unique())
#随机抽取15个患者进行绘图
fig, axs = plt.subplots(5,3,figsize=(20, 27))
patient_ids = data["patient_id"].unique()
np.random.shuffle(patient_ids)
for n in range(5):
for m in range(3):
patient_id = patient_ids[m+3*n] #与之前抽取切片进行绘制时相似的代码
singlepatient = data[data["patient_id"] == patient_id]
axs[n,m].scatter(singlepatient["x"],singlepatient["y"],c=singlepatient["target"],cmap="coolwarm",s=20)
axs[n,m].set_title("patient " + patient_id + " " + str(round(table.loc[patient_id,"positive_ratio"],3)))
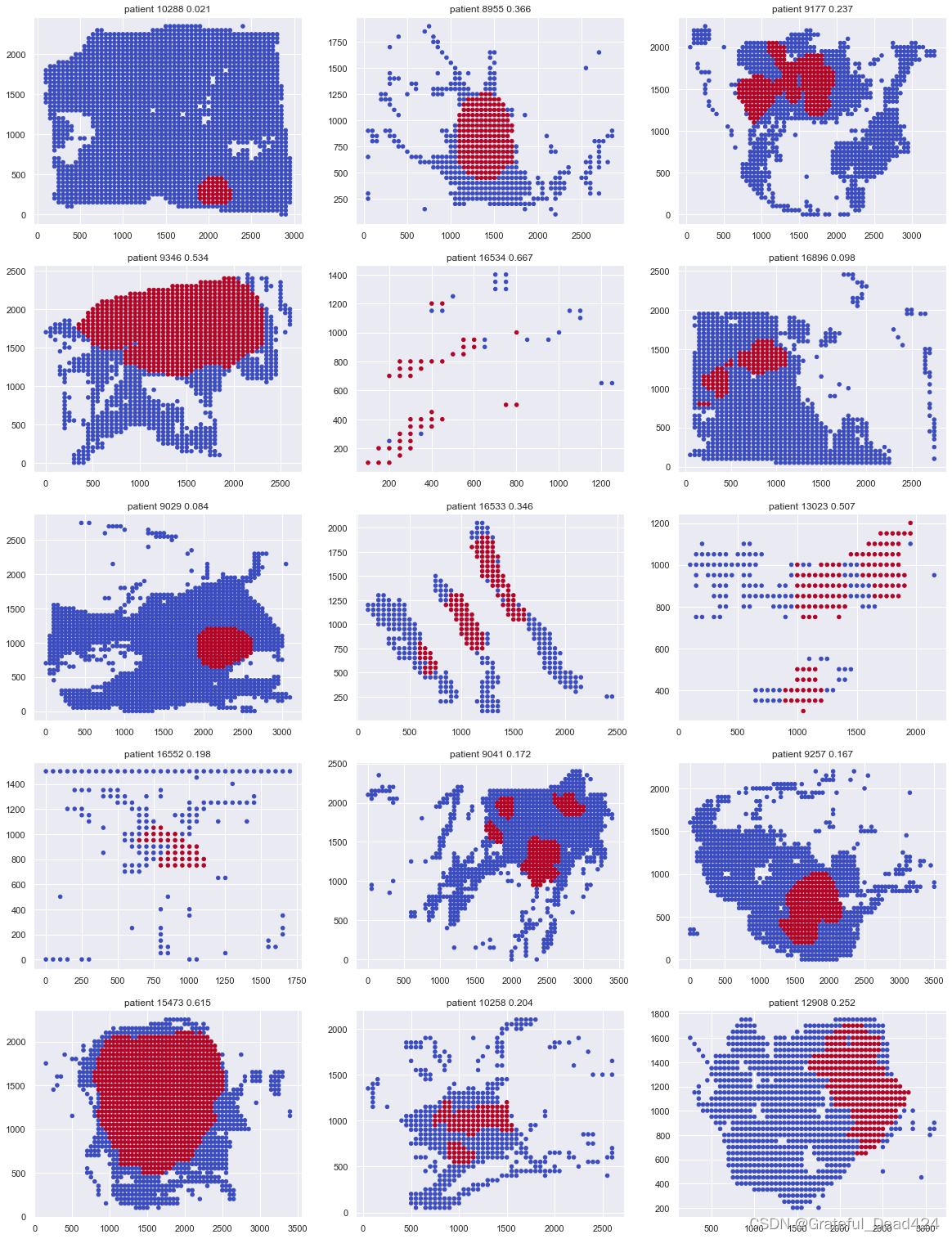
保留恶性组织为核心,非恶性组织选择性保留
到这里,我们已经理解了当前数据集的基本结构、图像形态以及相关的部分专业知识。接下来,我们就可以对数据集进行预处理和建模了。从下一节开始我们将讲解医疗图像的数据预处理。