Title:CLNN-loop: A deep learning model to predict CTCF-mediated chromatin loops in the different cell lines and CTCF-binding sites (CBS) pair types
期刊:Bioinformatics
中科院分区:2区
影响因子:6.931
服务器:CLNN
发表时间:2022年8月23日
目录
摘要
? ? ? ?动机:三维 (3D) 基因组组织在基因调控和疾病机制中至关重要。先前的研究表明,CTCF介导的染色质环对于研究细胞的3D结构至关重要。尽管已经开发了各种实验技术来检测染色质环,但发现它们既耗时又昂贵。如今,各种基于序列的计算方法可以捕获3D基因组组织的重要特征并帮助预测染色质环。然而,这些方法在预测染色质环方面性能低下,泛化能力差。
? ? ? ?结果:在这里,我们提出了一种新的深度学习模型,称为CLNN-loop,通过融合多个基于序列的特征来预测不同细胞系和CTCF结合位点 (CBS) 对类型中的染色质环。基于前人研究中的数据集进行的一系列检查分析表明,CLNN-loop 具有令人满意的性能,在预测染色质环方面优于现有方法。此外,我们应用 SHAP 框架来解释不同模型的预测,发现CTCF基序和序列保守性是不同细胞系和CBS对类型中染色质环的重要标志。
1 背景
? ? ?三维基因组组织在基因调控、DNA 复制中起着关键作用、进化和疾病机制。随着高通量染色体构象捕获技术(Hi-C)的发展,Rao et al. (2014) 发现结构和功能单元染色质环以 1 kb 的分辨率直接调节基因表达。先前的研究表明,染色质环由结构蛋白CCCTC结合因子 (CTCF) 和内聚素通过挤压介导,其中CTCF基序在两个位点以会聚方向与特定的非回文基序结合并充当环锚。
? ? ?在过去的几年里,各种实验技术被用于检测染色质环,如高通量染色体构型捕获(Hi-C) ,染色质与配对端标记的相互作用分析(ChIA-PET) 和捕获Hi-C 。然而,这些技术不仅在某些情况下不适用,而且耗时和昂贵。为了解决这些问题,已经提出了几种计算方法来预测染色质环。其中一些方法,如RIPPLE、TargetFinder、3DepiLoop、CTCF-MP、Lollipop和Peakachu依赖于机器学习模型,而其他方法则利用先进的深度学习方法构建有效的预测模型,如DeepTACT、SPEID和deepC。
? ? ??尽管这些计算方法促进了染色质环的预测,但它们需要大量的功能基因组信号,这在实践中难以实现。因此,吕等人。 (2021) 提出了一种名为 Deep-loop 的深度学习模型,仅使用序列来预测染色质环。然而,他们的方法中的几个缺点仍有待解决。首先,他们只使用了卷积神经网络(CNN),因此 Deep-loop 无法捕捉特征的顺序关系。第二个是它们的特定位置特征编码方案在不同 CBS 对的染色质环中显示出相反的分布。此外,他们只评估了四种特征编码方案,因此选择的特征可能不是最丰富的。总体而言,Deep-loop 的性能在某些情况下并不令人满意。特别是难以满足预测不同细胞系和CBS对类型中染色质环的要求。
? ??因此,我们提出了一种新的深度学习框架,称为 CLNN-loop(一种基于 CNN 和长短期记忆 (LSTM) 的神经网络),以准确预测CTCF介导的染色质环。具体来说,我们利用双向 LSTM 来捕获特征的顺序关系。此外,我们评估了 31 个基于序列的特征并确定了 5 个信息量最大的特征,并且我们根据染色质环的多方向特征改进了位置特异性特征。我们的主要贡献如下:(i)我们改进了位置评分函数(PSF)、基于单链的位置特异性三核苷酸倾向(PSTNPss)和基于双链的位置特异性三核苷酸倾向(PSTNPds)的性能:在预测不同细胞系和 CBS 对类型中的染色质环时染色质环的特征。 (ii) 我们确定了预测染色质环的五个最有用的特征。 (iii) 我们提出了一种新模型,称为 CLNN-loop,以准确预测相同细胞系/CBS 对类型中 CTCF 介导的染色质环和跨细胞系/CBS 对类型。 (iv) 我们解释了各种广泛使用的模型,并发现了不同 CBS 对类型和不同细胞系中染色质环的共同特征。本研究的框架如图 1 所示。
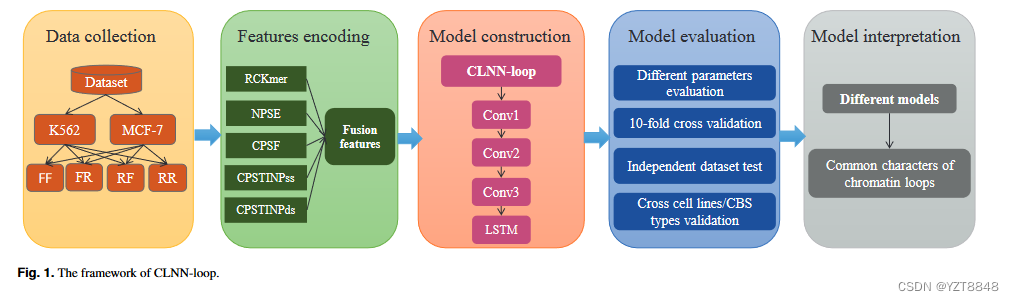
图1
2 数据集
? ? ?本研究中使用的数据集由 Lv 等人创建。 (2021),其中包含两个细胞系(K562和MCF-7),每个细胞系包含四种类型的CBS对(FF方向对,FR方向对,RF方向对和RR方向对)。具体而言,K562细胞系中四种CBS对类型的样本数分别为3184、23648、648和3012,MCF-7细胞系中四种CBS对类型的样本数分别为3182、18554、534和 3602 分别。四种类型的CBS对中正样本和负样本的比例为1:1。此外,为了客观评价模型的性能和鲁棒性,70%的数据作为训练集,30%的数据作为独立测试集。数据集的详细信息和使用情况见补充表 S1。
3 方法
3.1 特征编码
? ? ??在这项研究中,我们利用以下五种不同的序列编码方案来编码 DNA 序列,即反向互补 k-mer (RCKmer)、组合位置评分函数 (CPSF)、核苷酸对谱编码 (NPSE)、组合位置特异性三核苷酸倾向基于单链(CPSTNPss)和基于双链的组合位置特异性三核苷酸倾向(CPSTNPds)。所有的特征都是针对训练集和测试集分别计算的。这五种编码方案将在以下部分详细说明。
3.1.1 RCKmer
? ? ?RCKmer 是传统 k-mer 的一种变体,其中 k-mer 不是链特异性的,因此反向互补被折叠成一个单一的特征(Noble 等人,2005;Chen 等人,2020)。例如,“TT”是与“AA”的反向恭维,“AGT”是与“ACT”的反向恭维,“GTCAT”是与“ATGAC”的反向恭维。对于 DNA 序列,有 16 种类型的 2-mer(即 'AA'、'AC'、'AG'、'AT'、'CA'、'CC'、'CG'、'CT'、'GA' 、'GC'、'GG'、'GT'、'TA'、'TC'、'TG'、'TT')。去除反向补语k-mer后,只有10个不同的k-mer('AA','AC','AG','AT','CA','CC','CG','GA', 'GC', 'TA') 在反向补充 k-mer 方法中。在本研究中,我们设置 k = 5,因此基于 RCKmer 的特征的维度为 512。
3.1.2 CPSE
? ??位置评分函数(PSF)是一种用于挖掘序列位置信息的编码方案(Lv et al., 2021)。位置相关评分矩阵(PCSM)定义如下:
? ? ? ? ? ? ? ? ? ? ??
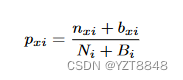
?
? ?其中 nxi 和 bxi 分别是 k-mer 片段 x 在第 i 个位置的真实计数和伪计数。 Ni和Bi分别是第i个位置的总实数和总伪数。 Bi 和 bxi 定义如下:
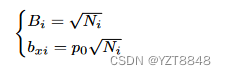
?其中 p0 是 k-mer 的背景频率,设置为 1/4k(这里,我们设置 k = 5)。基于 PSF 的特征定义如下:

?鉴于以往的研究表明数据集中序列的保守位点主要分布在9-31(Lv et al., 2021),我们只利用9-31(共42个)保守位点的PSF值序列中的位点)。另外,考虑到环序列在不同方向的保守性呈现相反的趋势,导致PSF值的分布相反,我们使用9-31位点的PSF值之和来代替传统的PSF特征。新功能被指定为组合位置评分功能(CPSF)。因此,基于 CPSF 的特征的维度为 1。
3.1.3 NPSE
? ? ? ? ?NPSE 策略用于计算被任何 k 个核苷酸分隔的所有可能核苷酸对的频率。例如,在序列“GXXXCXA”中,“GC”是一个三间隔的核苷酸对,“GA”是一个五间隔的核苷酸对。基于 NPSE 的特征定义如下:
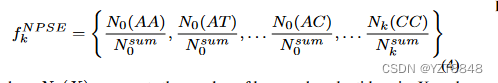
?其中 Nk(X) 表示 k 间隔核苷酸对 X 的数量,如果序列长度为 L,则 N sum k =L - k - 1。在本研究中,我们设置 k = 5。因此基于 NPSE 的特征是 4 × 4 × (5+1) = 96。
3.1.4 CPSTNPss
? ? ?基于单链的位置特异性三核苷酸倾向 (PSTNPss) 利用基于 DNA 序列单链特征的统计策略 (Chen et al., 2020; He et al., 2018, 2019)。使用 3-mer 核苷酸组成信息时,有 4的3次方 = 64 个三核苷酸(AAA、AAT、...、CCC)。假设序列长度为L,则三核苷酸位置特异性表示如下:

? ??其中 3meri 表示第 i 个三核苷酸,F +(3meri|j) 和 F -(3meri|j) 分别表示正数据集中和负数据集中第 j 个位置的第 i 个三核苷酸的出现频率。基于 PSTNPss 的特征定义如下:
![]()
?其中 φu 定义如下:
其中 Nu 是第 u 个位置的基数(‘A’、‘G’、‘C’或‘T’)。与特征 PSF 类似,我们使用 PSTNPss 值的总和来替代传统的 PSTNPss 特征,并将新特征命名为 CPSTNPss。因此,基于 CPSTNPss 的特征的维数为 1。

?其中 Nu 是第 u 个位置的基数(‘A’、‘G’、‘C’或‘T’)。与特征 PSF 类似,我们使用 PSTNPss 值的总和来替代传统的 PSTNPss 特征,并将新特征命名为 CPSTNPss。因此,基于 CPSTNPss 的特征的维数为 1。
3.1.5 CPSTNPds
? ? ??基于双链的位置特异性三核苷酸倾向 (PSTNPds) 利用基于互补碱基配对的 DNA 序列双链特征的统计策略 (Chen et al., 2020; He et al., 2018, 2019)。因此,我们认为“A”和“T”相同,“G”和“C”相同,因此有 2的3次方 = 8 个三核苷酸:AAA、AAC、...、CCC。假设序列长度为L,则三核苷酸位置特异性表示如下:

?其中 3meri 表示第 i 个三核苷酸,F +(3meri|j) 和 F -(3meri|j) 分别表示正数据集中和负数据集中第 j 个位置的第 i 个三核苷酸的出现频率。基于 PSTNPds 的功能定义如下:
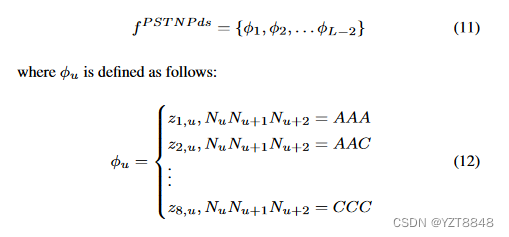
?其中 Nu 是第 u 个位置的基数(“A”或“C”)。与特征 PSF 类似,我们使用 PSTNPds 值的总和来替代传统的 PSTNPds 特征,并将新特征命名为 CPSTNPds。因此,基于 CPSTNPds 的特征的维度为 1。
4 深度学习框架
? ? ?
?图2
? ? ??图2所示。CLNN-loop的体系结构。CLNN包含三个卷积层和一个双向LSTM层。其中,每个卷积层的核个数和大小分别为32和5,LSTM单元数为32。
5. 结果
? ?补充文件
? ? ? 最近,一些研究表明,在基于序列的特征中,特征融合策略优于单个特征(Lv等人,2021;Hu等,2022;蔡等人,2021年;Wu等人,2022)。为了获得信息最丰富的特征进行特征融合,我们首先评估了不同基于序列的特征在相同CBS对类型中区分循环和非循环的能力。为了避免测试集的数据泄漏问题,我们从K562细胞系中四种类型CBS对的训练集中提取了31个特征,并使用LR分类器进行10倍交叉验证来评估这些特征的性能,包括Kmer(补充方法S3)、Mismatch k-tuple(补充方法S4)、Natural V ector(补充方法S5)、lsequence profile(补充方法S6)、CPSF、CPSTNPss、CPSTNPds、NPSE、Word2vec(补充方法S7)和来自iLearn的22个其他基于序列的特征(Chen等人,2020年)。从K562细胞系中四种CBS对类型的训练集中提取不同特征的详细结果见补充表S3;这些特征的平均结果见补充表S4,这些特征的平均AUC值见补充图S1。从补充图S1可以看出,在相同CBS对类型下,12个特征对环路的预测性能是令人满意的(这些特征的AUC值大于0.95),这说明这些特征能够准确预测相同CBS对类型下的环路。然而,该特性在区分同一CBS对中的循环和非循环方面的高性能在评估跨CBS对类型时不可转移。因此,我们进一步利用AUC值大于0.95的12个特征进行跨cbs对类型验证。考虑到FR方向对的数量最大,使训练模型更可靠,我们使用K562细胞系中FR方向对的样本作为训练集,其他三个方向对(FF, RF, RR)的样本作为独立测试集。交叉cbs类型验证的详细结果见补充表S5,平均结果见补充表S6。这些特征在预测相同和不同CBS对类型时的平均AUC值见补充图S2。从补充图S2可以看出,在评估交叉CBS对类型时,某些特征区分环路和非环路的能力大大降低。binary、EIIP、ENAC、NCP、Kmer、Mismatch和Word2vec的AUC值分别降低了30.98%、33.39%、38.58%、30.98%、16.78%、18.74%和20.24%。这些结果的主要原因可能是这些特征受序列顺序的影响较大,在不同方向上呈现相反的趋势,如前所述。因此,用这些特征训练模型可能会导致过拟合问题,从而降低模型识别不同CBS对类型的染色质环的能力。相反,在评估交叉CBS对类型时,其他五个特征在区分循环和非循环的能力上只略有下降。CPSTNPds、CPSTNPss、RCKmer、NPSE和CPSF的AUC值分别仅下降2.80%、0.37%、0.89%、8.85%和0.53%。这些结果表明,这5个特征对相同或不同CBS对类型的环路有较强的预测能力,用这些特征训练的模型可能具有较好的鲁棒性。因此,我们将这五个特征的融合作为我们模型的输入。
6 论文实验相关
6.1 模型优化
? ? ??为了得到性能最好的模型,我们用不同的参数对模型进行了评估。我们关注了对模型性能有很大影响的许多参数,包括学习率、核数、核的大小和LSTM单元的数量。我们使用网格搜索的方法来优化参数。同样,假定FR定向对的最大个数使训练的模型更加可靠,我们评估了K562细胞系中具有不同参数的模型在FR定向对上的性能。不同参数下模型的结果见补充表S7。可以看出,模型的性能取决于参数的设置,当学习率为0.001、内核数为32、内核大小为5、LSTM单位为32时,模型的性能最好。因此,我们利用这个参数组合来构建我们的模型。
6.2 十倍交叉验证
? ? ? 之前的研究表明,随机划分测试集可能会导致训练和测试集之间的性能有很大差异(Cai等人,2021;Lim等人,2021年;卡纳尔等人,2020年)。因此,我们对来自两个细胞系的四种CBS对的训练集进行10倍交叉验证,以全面评估CLNNloop的性能。结果见补充图S4和补充表S8。从两个细胞系可以看出,CLNN-loop在预测四种类型CBS对的环路时具有极高的性能。8个数据集的10倍交叉验证的AUC值均大于0.98,其他5个指标的值也非常理想。这些结果表明,CLNN-loop在融合多种特征预测染色质环方面具有具有竞争力的性能。
6.3 独立测试
? ? ?为了进一步验证CLNN-loop的性能,研究CLNN-loop是否优于其他分类算法,我们使用独立测试集来衡量CLNN-loop的稳定性,并将其与许多机器学习分类算法进行比较,包括KNearest Neighbor (KNN) (Altman, 2008)、决策树(DT)、随机梯度下降分类器(SGDC)、梯度提升(GB)和支持向量机(SVM) (Chang et al., 2011)。结果见补充图S5和补充表S9。从两个细胞系可以看出,在大多数类型的CBS对中,CLNN-loop的六个指标都高于其他算法。这些结果表明,CLNN-loop在所有评价指标上都优于其他预测器,这表明CLNN-loop是一种预测染色质循环的有效工具。
6.4 与前人做的结果比较
? ? ??为了更好地验证CLNN-loop模型在检测染色质环方面的性能,我们进一步将其应用于跨细胞系和CBS对类型,并与之前的研究进行比较。鉴于以往的研究大多使用非dna序列数据,我们只评估了CTCF-MP (kmer)、CTCF-MP (word2vec)、Deep-loop和CLNNloop的性能。为了保证这些方法的可比性,我们采用了与Deep-loop相同的评价方法。即在预测不同细胞系或CBS对类型的染色质环时,我们在一个细胞系的一个CBS对训练集上训练模型,并在其他7个细胞系和CBS对的独立测试集上分别计算AUC值;在预测同一细胞系和CBS对类型的染色质环时,我们执行10倍交叉验证来计算AUC值。四种型号的性能如图3和补充表S10-S13所示。从图3可以看出,CTCFMP (kmer)和CTCF-MP (word2vec)在预测同一细胞系和CBS对类型的染色质环方面表现较好,但在预测不同细胞系和CBS对类型的染色质环方面表现较差。尽管Deep-loop相对于CTCF-MP (kmer)和CTCF-MP (word2vec)提高了预测性能,但它在预测不同细胞系和CBS对类型的染色质环方面的性能并不理想。特别是在预测不同细胞系中完全相反的CBS对环路时,Deep-loop的性能大大降低。幸运的是,本研究提出的CLNN-loop在预测同一细胞系/CBS对类型和跨细胞系/CBS对类型的环路时,性能都非常高。在64个结果中,CLNN-loop对55个结果表现最好,64个结果的平均AUC值为0.9937,CTCF-MP的平均AUC值(kmer)为0.9937。CTCF-MP (word2vec)和Deep-loop分别为0.8470、0.8595和0.9523。这些结果表明,CLNN-loop在预测染色质环方面比目前最先进的方法更有效,并且具有更好的泛化和鲁棒性。
6.5 新细胞验证模型
? ??为了进一步验证我们提出的方法是否可以预测新的细胞系中的染色质环,我们预测了IMR90细胞系中的染色质环序列。Lv等人(2021)从IMR90细胞系中鉴定了四种类型的CBS对,数据可从https://github.com/linDing-group/Deep-loop下载。我们利用两个细胞系的四种类型的CBS对训练集构建了8个模型,预测了IMR90细胞系中四种类型CBS对的染色质环。结果载于补充表S14。可以看出,8个模型都能从IMR90细胞系中有效预测四种类型CBS对的染色质环,即使在预测跨细胞系和CBS对类型的环路时,也能很好地预测染色质环。此外,为了进一步证明CLNN-loop的泛化性,我们分别使用K562细胞系和MCF-7细胞系的训练集构建两个模型,从IMR90细胞系预测四种类型CBS对中的染色质环。结果表明,基于K562和MCF-7细胞系的模型对来自IMR90细胞系的4种CBS对的环的识别率分别为99.99%(90317/90320)和99.96%(90287/90320)。这些结果表明,我们提出的CLNN-loop模型可以用一个模型预测四种类型的CBS对中的环路,而不是针对不同类型的CBS对使用不同的模型,因此我们的模型是有效预测染色质环路的强大和鲁棒性。
6.6 不同算法的比较
? ? ?在预测新细胞系的循环时,通常不知道CBS对的类型。因此,之前的研究(Lv et al., 2021)利用位置权重矩阵(PWM)来识别CBS对的类型,然后构建相应的模型来预测染色质环。然而,这是非常耗时的。为了有效解决这一问题,我们利用SHapley Additive解释(SHAP) (Lundberg et al., 2017)框架来识别预测循环中最重要的特征,并进一步探索不同细胞系中不同类型CBS对的共同特征。考虑到不同分类算法识别的最重要特征可能不同,我们解释了多种分类算法的预测结果,包括广泛应用的机器学习算法KNN, LR, DT, SGDC, GB, SVM和广泛应用的深度学习算法CNN。对于每个分类算法,我们使用来自两个细胞系的四种类型CBS对的训练集构建多个模型,并使用来自两个细胞系的四种类型CBS对的独立测试集解释模型。我们为每个样本绘制特征的theSHAP值,它反映了特征对预测结果的影响。对不同模型预测影响最大的前10个特征的分布情况见补充图S6-S12。具有positiveSHAP值的高特征值意味着预测结果为正样本的概率随着特征值的增加而增加。相反,SHAP值为负的高特征值表示预测结果为正样本的概率随着特征值的增大而减小。采用补充图S6中的“特性1028”。以K562-FF为例,“特征1028”的特征值对于大多数阳性样本来说是较高的,而“特征1028”的特征值对于大多数阴性样本来说是较低的。因此,对于一个未知的新样本,如果样本中“feature 1028”的特征值较高,则预测结果有利于循环;否则,预测结果将倾向于非循环。
7 讨论
? ? ?在本研究中,我们解释了不同的模型,以确定来自不同细胞系的不同CBS对类型的染色质环的共同特征。然而,即使融合特征中包含了反映基序潜在关系的基于RCKmer的特征,也很难检测到在染色质环的形成中起重要作用的CTCF基序,这可能是由于以下两个原因:第一,RCKmer去除了反向补位k-mer,这可能削弱了k-mer片段的重要性。第二个原因是序列的长度只有42,所以RCKmer相对于其他特征来说是非常稀疏的,这可能会降低每个RCKmer片段的重要性。为了进一步探究motif对模型预测的影响,我们仅使用基于kmer (k=5)的特征来构建模型并解释模型的预测。我们将K562和MCF-7细胞系中最具影响力的前10个k-mer片段与JASPAR数据库(Khan et al., 2018)进行比较,ID为MA0139.1。结果见补充图S10。可以看出,许多特征对模型的预测有显著的影响(特征的值代表k-mer片段的频率)。巧合的是,大多数这些特征都与CTCF主题相匹配,它们是绑定因子,并与内聚体一起工作以挤出循环(Sanborn等人,2015年)。这些结果表明,CTCF基序和序列守恒是染色质环的重要标志。然而,也有一些k-mer片段与CTCF基序不匹配,但对模型预测有影响,这说明染色质环的形成非常复杂,与多种因素有关。这些结果表明,同时使用CTCF的功能基因组信号和序列守恒可以有效提高预测染色质环的准确性。希望我们利用CTCF的功能基因组信号和序列守恒预测染色质环的发现可以为三维基因组结构的研究提供帮助。??
? ? ? ?此外,我们还开发了一个用于预测染色质环的在线服务器。为了更好地提供服务和更好的性能,我们为不同的细胞系建立了不同的数据基准。由于我们已经收集了三个细胞系(K562, MCF-7, IMR90)的数据集,我们只建立了这三个细胞系的数据基准。在未来,我们将继续收集数据集,并将它们部署到web服务器上。当然,我们的模型在预测跨细胞系/CBS对类型的染色质环方面也有出色的表现,因此生物学家可以使用我们的web服务器通过提交任何细胞系的数据来预测染色质环。
8 结论
? ? ? ?在本研究中,我们提出了一个新的深度学习模型,称为CLNN-loop,用于预测不同细胞系和CBS对类型的染色质循环。我们首先根据顺序比较31个特征,以确定5个信息量最大的特征。基于10倍交叉验证、独立测试、跨细胞系/CBS对类型验证和新细胞系预测的实验证明,CLNN-loop是一种比现有方法更有效、更鲁棒的方法。此外,我们解释了不同的模型,以发现不同细胞系/CBS对类型的共同特征。最后,我们开发了一个在线网站服务器,以方便整个社区的学习。希望我们的方法能够帮助推进三维基因组结构的研究。