- 5.3 寻找远缘相关蛋白质:位置特异性迭代BLAST(PSI-BLAST)和DELTA-BLAST
- PAM250矩阵给探测远缘相关蛋白质提供了一个更好的打分系统,可以改变打分矩阵来检测远缘蛋白质,但仍然有局限性:
- BLASTP检测到匹配蛋白,但是否同源并不明确。
- PSI-BLAST
- 更深入地搜索数据库,以发现一个与你感兴趣地蛋白远缘相关的匹配蛋白。
- 比对的5个步骤:
- 使用某个打分矩阵对查询序列在一个目标数据库中进行搜索(同常规BLASTP)
- 基于成分的统计数据从起始搜索的结果中构建一个多序列比对,基于该多序列比对,构建专门的、个体化的搜索矩阵(或者叫谱profile)
- 得到的位置特异性打分矩阵(PSSM)会被用来进行再一次搜索
- PSI-BLAST对数据库匹配的统计显著性进行评估。
- 以上搜索被重复迭代,一般迭代五次。
- 图例:得到的位置特异性打分矩阵(PSSM)
-
- 图例:3次迭代后的比对结果
-
- PSI-BLAST的错误:污染问题
- 错误的主要来源于无关序列的虚假扩增,有三种措施阻止污染
- 1.使用过滤算法来去除由组成偏好性的氨基酸区域
- 2.将入选阈值从默认值调整为一个较低的值
- 3.肉眼检查每一次PSI-BLAST迭代的结果
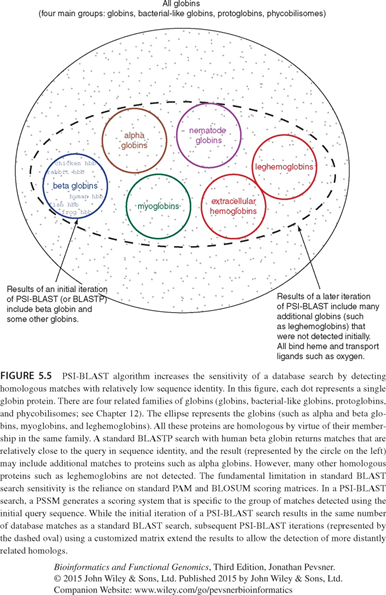
- 图示:所有的球蛋白及PSI-BLAST如何提升搜索灵敏度
-
- 错误的主要来源于无关序列的虚假扩增,有三种措施阻止污染
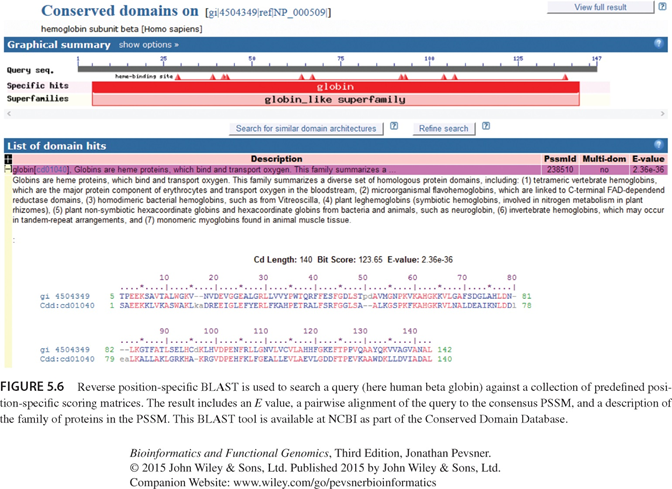
- 反向位置特异性BLAST(RPS-BLAST)
- RPS-BLAST可以把一个查询蛋白序列与一个预先定义好的PSSM数据库进行比对,可鉴别查询序列中保守的蛋白质结构域。
- 图示:RPS-BLAST查询
-
- 图示:RPS-BLAST查询
- RPS-BLAST可以把一个查询蛋白序列与一个预先定义好的PSSM数据库进行比对,可鉴别查询序列中保守的蛋白质结构域。
- DELTA-BLAST
- NCBI上最灵敏和准确的蛋白质搜索工具
- 优势
- 基于质量高的人工审核的CDD数据库,可以产生比PSI-BLAST更大、更完整的PSSM。
- 比BLASTP和PSI-BLAST更加灵敏,包括可更灵敏地搜索远缘相关蛋白
- 速度快
- 比BLASTP得到的序列比对质量更好。
- PSI-BLAST和DELTA-BLAST的性能评估
- DELTA-BLAST灵敏度由于PSI-BLAST、BLASTP和其他程序;
- 在给定数量的假阳性条件下,DELTA-BLAST可以发现三倍于BLASTP所发现的同源蛋白质
- 模式匹配发起的BLAST(PHI-BLAST,模式识别BLAST)
- 某个蛋白可能包含了某个氨基酸残基模式或者“特征信号”氨基酸残基,这可以帮助我们判断该蛋白质属于某个家族。PHI-BLAST可查找到既包含匹配模式又与查询序列相关的匹配结果;DELTA-BLAST灵敏性高,但它不会输出关于用户所选的模式的相关信息。
- 特征信号和模式的定义
- 自定义,并引入一定程度的模糊度,比如NDFX(5)GXW[YF]:
- X(5)代表这五个位置上可以是任意一种氨基酸残基;
- [YF]表明最后一个位置上的氨基酸残基必须是络氨酸或者苯丙氨酸中的一个;
- 所选择的模式不能出现地太频繁,算法只允许出现频率小于1/5000.
- 自定义,并引入一定程度的模糊度,比如NDFX(5)GXW[YF]:
- 图示:选择一个模式进行PHI-BLAST搜索
-
- PHI-BLAST算法根据跨越输入模式和其上下游的A1区域和A2区域得到的双序列比对A0进行分析,该序列比对结果利用有空位的延伸进行打分。
- PAM250矩阵给探测远缘相关蛋白质提供了一个更好的打分系统,可以改变打分矩阵来检测远缘蛋白质,但仍然有局限性:
- ?欢迎加群交流,或者加VX:bbplayer2021,邀请进群

?