本文是根据原版教程的介绍进行的总结。不得不说Relion是用过的cryoEM和cryoET里最好用、教程最详细最贴心的,respect。就是安装的时候有些许麻烦,使用体验还是不错的。这里就不点名拉踩其他的一些软件了,反正就是容易让人暴躁吧,难怪没什么人用……
📚教程指路:Relion4.0 Tutorial
安装注意事项
因为我之前安装过Relion3.1版本,所以为了使用最新版本,首先需要使用命令git checkout ver4.0更新到4.0版本的代码,然后重新按照教程cmake ..一遍再make,这里注意以前生成的命令所在的位置和现在的不要搞混了,我是把之前的删掉了又重新生成的。(之前保存到/usr/local/bin里面了,这次把相关的命令删了,直接将relion/build/bin所在的路径加到了环境变量PATH里,source一下bashrc,再打开relion的GUI,终于打开的是4.0 beta版本了!)
如图,长这样:(记得检查一下左上角版本)
准备实验数据
这里教程提供了一套数据,来自EMPIAR-10164。大小5个G多,第一次下载的时候中止了,结果解压出来发现少了好多东西,所以一定要注意一下是否下载成功了哈。
指令:
wget ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion40_sta_tutorial_data.tar
tar -xf relion40_sta_tutorial_data.tar
里面应该有三个子文件夹:tomograms,masks,input。
tomograms里面有5个子文件夹。
预处理
用户需要对层析图进行外部预处理。完成 tomogram 的 movies frames alignment, tilts series alignment, particle picking。
- Tilt series alignment: 使用IMOD完成。
- Initial CTF parameters: CTFFind / CtfPlotter。
- Order list: 倾斜序列获取的时间顺序。
- Fractional electron dose: 每个倾斜图像的电子剂量。
建议为每个项目创建一个目标,来针对每个你想确定的结构。然后在项目目录中启动RELION的GUI。在项目目录中,应该创建一个单独的目录来存储所有IMOD重构的tomograms文件夹,可以命名为tomograms/,在里面应该在TS_1等文件夹中找到不同的层析图。
relion使用倾斜周对齐钱的原始倾斜序列对阵,并从IMOD导入对齐数据。tomogram文件夹中应该包含一个堆栈文件(.st/ .mrc/ .mrcs),newst.com和tilt.com脚本文件,其中包含了转换和倾斜角度文件名和层析图尺寸、偏移量和排除视图。
粒子坐标应该参考使用该信息重建的层析图像。
使用命令来开始项目:
relion --tomo&
导入tomograms
导入的数据集中断层图已经使用IMOD配准过,用CTFFind或者CtfPlotter估计过CTF。
创建一个star文件来存储相关信息。每一行表示一个tomogram。基本字段有倾斜序列堆栈文件名、CTF估计数据文件名和IMOD项目文件夹。如果电子剂量不是恒定的,也要在文件中提供,而不是在导入时作为全局参数提供。
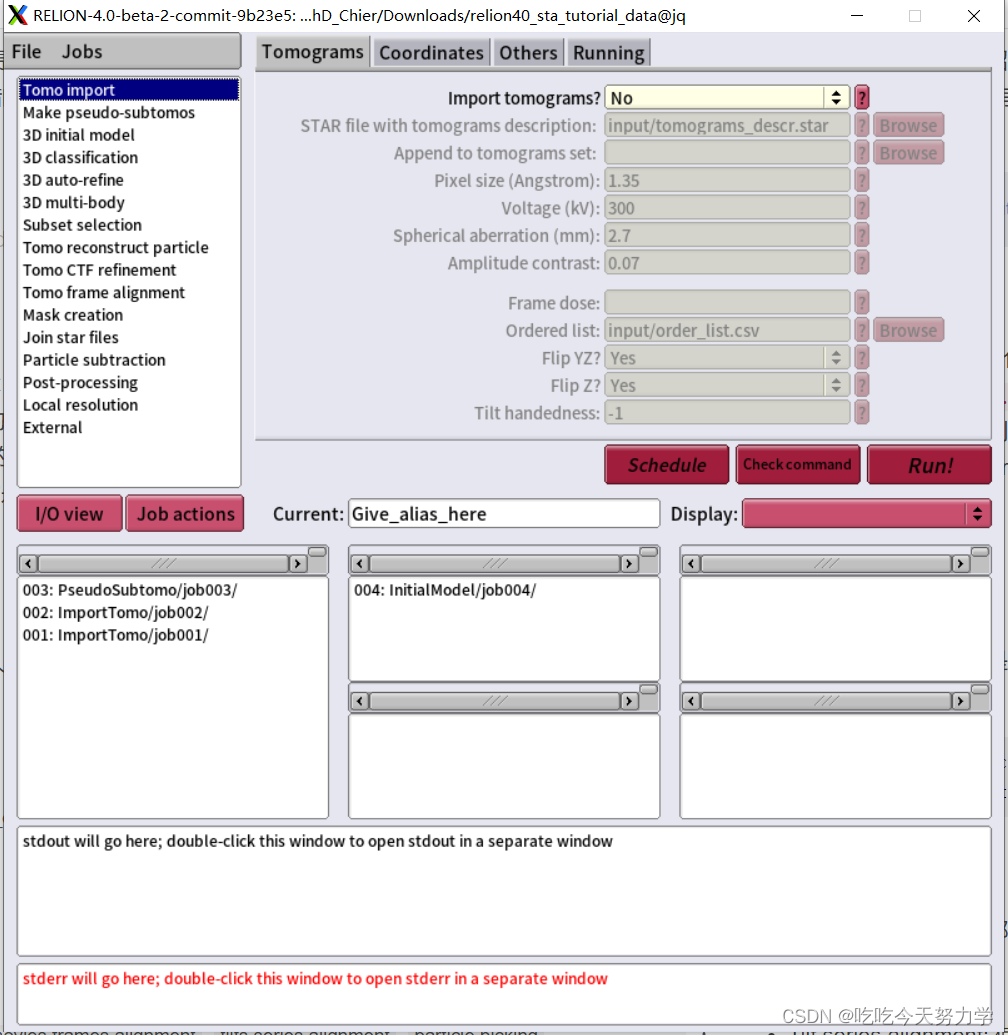
选择 Tomo import,填写 Tomograms 标签页。
运行 Run! 以后会生成文件夹ImportTomo/job001/,里面会生成 tomograms.star 文件。它包含一个具有一般层析图像属性的表,以及每个层析图的特定表,包括投影矩阵、散光散焦、累计辐射剂量和每个倾斜帧的变形等。
导入坐标
构造pseudo-subtomos必需的是每个颗粒的三维坐标集和对应的层析图。
还是选择 Tomo import ,这次填写的时 Coordinates 标签页,而且要在Tomograms 标签页把 Import tomograms 设为 No。
生成伪子断层
在使用任何常规relion程序(那些不是专门用于断层扫描的程序,如relion_refine)进行处理之前,我们首先需要构建单个伪亚层析图像粒子,这相当于SPA工作流中的粒子提取过程。
选择Make pseudo-subtomo,
从头生成3D模型
这一步需要的时间比较长。
