一般来说网络图都是R语言导出数据放入cytoscape里面绘制,今天想试试在R语言内部去画,确实有相应的R包(igraph)可以做,不是专门针对PPI网络来的,自己组织一下数据可以画。
尤其是基因数量比较少的情况下,还是很好用的,省掉手动点鼠标的重复操作。要是基因数量多了,可能会有基因重叠,得把图导出去修改,就不如cytoscape里直接拖动方便了。
1.输入数据
string_interactions.tsv是从string网页下载的PPI网络输出结果文件。图上基因的颜色是按照上下调来分配的,在这个例子里基因上下调信息是编的,实际应用时可以从差异分析结果中得到。
2.样图
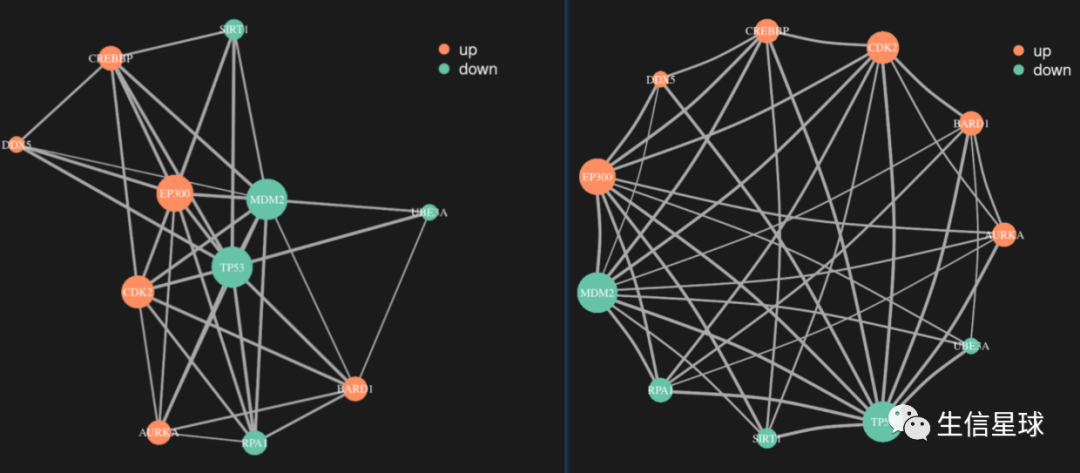
3.代码如下
library(igraph)
links= read.delim("string_interactions.tsv")
network <- graph_from_data_frame(d=links[,c(1:2,13)], directed=F)
deg <- degree(network, mode="all")
nodes <- data.frame(
name=unique(links$X.node1),
group=c( rep("up",6),rep("down",5)))
network <- graph_from_data_frame(d=links, vertices=nodes, directed=F)
my_color = c("#