MS在linux服务器上的并行运算脚本
MS使用超算的方式
windows端
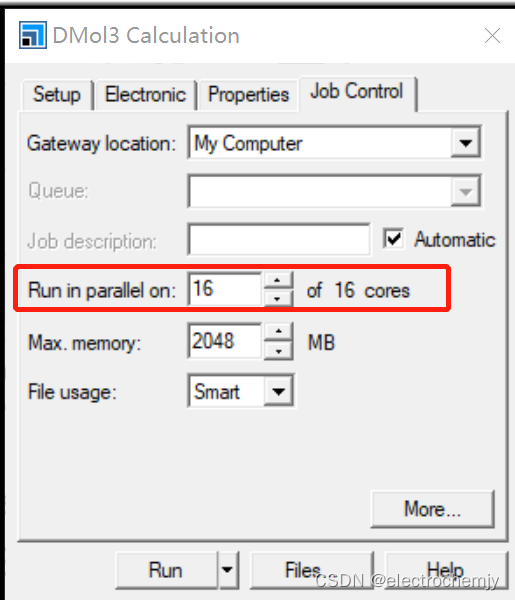
近期科研需要购买了超算资源,Materials Studio是生化环材天坑专业进行理论计算用到的软件。由于需要使用较为复杂的算法求解薛定谔方程,因此常常需要借助超算资源才能算得动。MS使用超算有两种方式,一种是通过windows版本的gateway将任务提交到超算平台,计算结束后会自动返回计算结果。这种方式可以直接在windows版本的job control设置任务并行计算时的核数。
linux命令端
另一种方式是通过脚本直接在服务器上提交运行,运行结果需要自行下载保存。当进行批量实验,且想要连夜跑实验时,灵活使用脚本运算将非常方便。
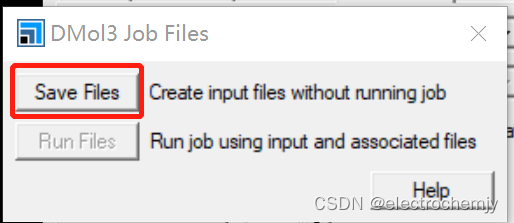
首先需要先在windows版本将设置好的条件输出为文件,将会在目录生成输入文件。

接着将整个文件夹复制到linux文件管理器(如Winscp),这个便是计算的输入文件,一个不能少,必须整个文件夹复制,不要一个个文件复制,因为文件夹中存在隐藏文件。
下面便需要一份脚本。
MS在linux上计算脚本-单份任务
既然要计算,便需要配置好计算环境。下面参考MS安装文件中的Dmol3使用脚本运行的命令,该命令在:/BIOVIA/MaterialsStudio19.1/etc/DMol3/bin中的RunDMol3.sh文件。在同文件夹的RunDMol3.Readme中有说明该脚本的使用方法:
RunDMol3.sh [-h] [ -q <queue name> ] [-nodelete] [-np <number of cores>] <basename> (Linux)
举个例子:
#!/bin/bash
#SBATCH -p v5_192
#SBATCH -N 1
#SBATCH -n 48
#上面三条命令是slurm作业系统的命令,如果没有作业系统,把这三条命令删除即可,注意这三条命令前面需要两个##才能达到忽略作用。
INPUTFILE=POSCAR20 #文件名,注意这是把脚本放进输入文件夹的情况
NUM_NODES=1 #计算节点数
PROCS_PER_NODE=48 #选择节点计算核数/处理器数目
MS_PATH=/public1/home/scfa0249/BIOVIA/MaterialsStudio19.1 #MS安装位置
NUM_PROCS=`expr $NUM_NODES \* $PROCS_PER_NODE`
srun -N $NUM_NODES -n $NUM_NODES hostname > .names.log
awk -v iPROCS_PER_NODE="$PROCS_PER_NODE" '{print $1":"iPROCS_PER_NODE}' .names.log > ./machines.LINUX
export DSD_MachineList="./machines.LINUX"
$MS_PATH/etc/DMol3/bin/RunDMol3.sh -np $NUM_PROCS $INPUTFILE #计算命令
该脚本的使用:将该脚本放进前面MS生成输入文件的文件夹中,接着将INPUTFILE=POSCAR20中的POSCAR20改为文件夹的名字,MS安装位置也要根据自己的情况进行修改。其他的基本可以不变,或者根据计算任务改一下节点数跟处理器数目就行。
然后在linux命令行窗口进入该文件夹,使用:
bash 脚本名字.sh
即可进行运算。
如何查看是否进行并行计算
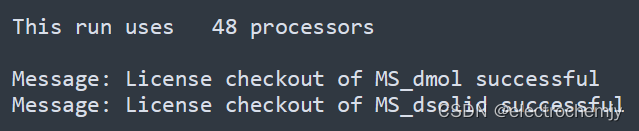
打开计算结果文件outmol,可以查看该份计算使用了多少个处理器。
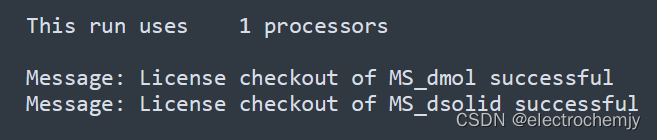
如果脚本指定了运行核数,而实际运行仍是使用一个处理器。此时需要对MS中的几份安装文件进行环境配置。
详情可见: [CASTEP/Dmol3/MS] MS2018不能够并行运算,该怎么去编写脚本呢?
概要的说,需要使用cd命令进入以下路径(根据自己的安装位置有所变化):
/BIOVIA/MaterialsStudio19.1/etc/Gateway/root_default/dsd/conf
使用命令:ls
可以看到以下文件:

需要分别进入文件(命令:vim 文件名+文件后缀):gw-info.sbd,gwparams.cfg
修改以下项目(进入文件后,输入i,即可进行编辑):
- 对于gw-info.sbd文件,做以下修改:
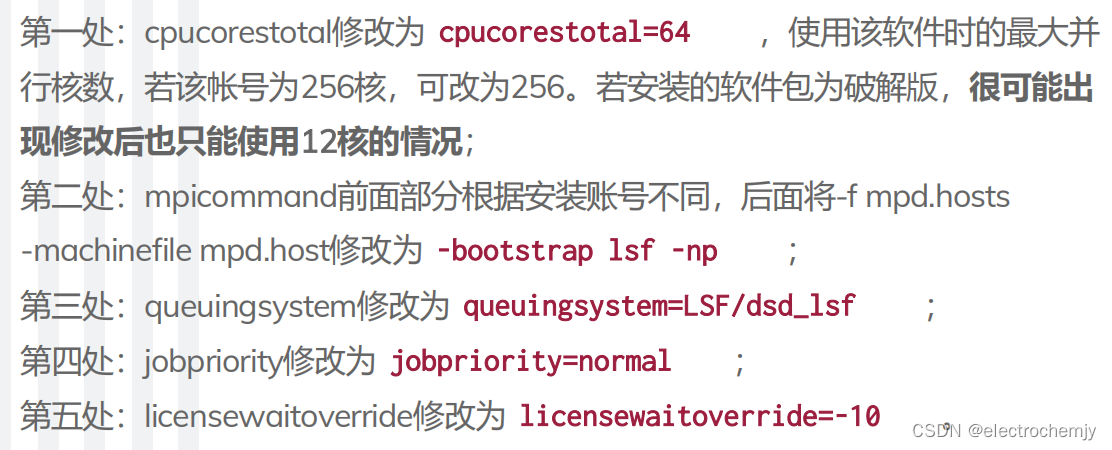
其中本人实践发现,最重要的是第一条,如果发现五条都改了以后,程序不能正常运行(比如输出文件不正确等问题),建议只修改第一条。此外,代码中LSF/dsd_lsf是因为使用的是LSF作业管理系统,如果是其他作业系统,比如slurm,则需要改为SLURM/dsd_slurm。
修改完成后,按下键盘Esc键,接着输入:wq即可完成文件修改并退出。 - 接下来修改gwparams.cfg文件,
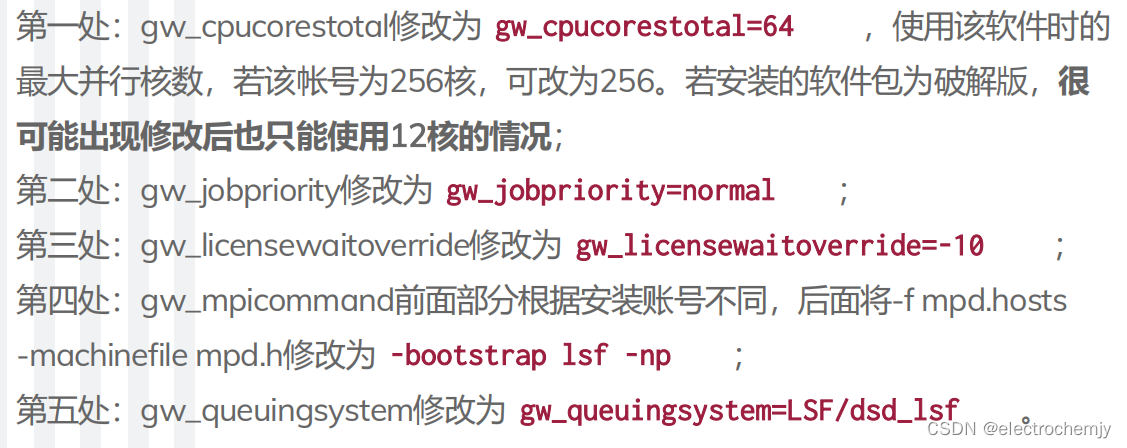
与修改gw-info.sbd文件的注意事项相同,第一条最重要,如果出现异常,后四条不要进行修改。
完成以上两份文件的修改后,需要更新作业管理系统的配置,使用cd命令进入以下路径:
/BIOVIA/MaterialsStudio19.1/etc/Gateway/config
进入该文件夹后,在linux命令行窗口输入以下命令即可完成作业系统的配置:
./configure queue -queuepath SLURM/dsd_slurm -activate
将在命令行窗口出现以下提示,便完成了作业系统配置的更新。
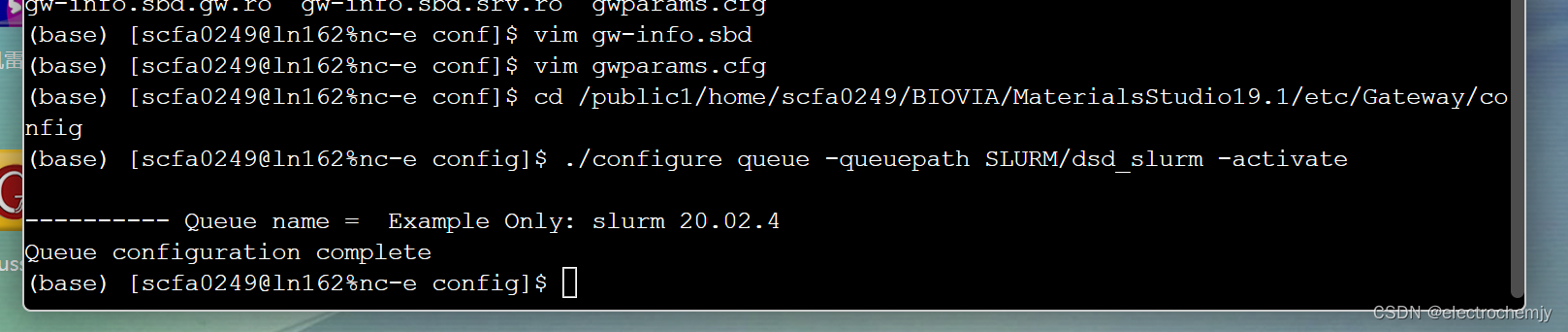
再次运行前面的脚本,检查是否使用了多个处理器机型运算,如果没有,则需要考虑是否在其他方面出现了问题,在此不再赘述。
MS在linux上计算脚本-批量任务
此份代码适用于以下情况:
一个文件夹中放有多份待计算的文件夹,每一个文件夹内是一个任务的所有输入文件。

批量计算的思想设计:
- 进入大文件夹,使用for循环遍历该文件夹下含有的小文件夹
- 接着根据文件后缀搜索每个小文件夹中的input文件,得到input文件的文件名,用于计算
- 接着使用计算模块的计算模板命令,即可完成运算。
例如:
#!/bin/bash
##SBATCH -p v5_192
##SBATCH -N 1
##SBATCH -n 48
#不使用作业系统,所以上面三条命令使用两个##进行忽略,不执行
NUM_NODES=1 #节点数
PROCS_PER_NODE=48 #核数
MS_PATH=/public1/home/scfa0249/BIOVIA/MaterialsStudio19.1 #MS安装路径
NUM_PROCS=`expr $NUM_NODES \* $PROCS_PER_NODE`
srun -N $NUM_NODES -n $NUM_NODES hostname > .names.log
awk -v iPROCS_PER_NODE="$PROCS_PER_NODE" '{print $1":"iPROCS_PER_NODE}' .names.log > ./machines.LINUX
export DSD_MachineList="./machines.LINUX"
#$MS_PATH/etc/DMol3/bin/RunDMol3.sh -np $NUM_PROCS $INPUTFILE
export poscar_dir=./check_for_1N1Vac-H/ #大文件夹名字
#下面利用循环实现批量运行
for file in `ls -l ${poscar_dir} |awk '/^d/ {print $NF}'`;do
# 打印实验文件夹名
echo "对文件夹${file}执行实验..."
# 切换到需要进行实验的目录
cd "${poscar_dir}${file}"
# 查找.input后缀文件
for expname in `ls . | grep .input`;
do
echo "实验文件名:${expname%%.input*}"
#运行实验
$MS_PATH/etc/DMol3/bin/RunDMol3.sh -np $NUM_PROCS "${expname%%.input*}"
done
echo "${file}计算完成"
cd ..
cd ..
done
echo "所有POSCAR计算已经完成"
以上便是本人这几天的记录,欢迎来到这里的各位批评指正。
